Diluizioni seriali e piastratura: la conta microbica
English
소셜에 공유하기
개요
Fonte: Jonathan F. Blaize1, Elizabeth Suter1e Christopher P. Corbo1
1 Dipartimento di Scienze Biologiche, Wagner College, 1 Campus Road, Staten Island NY, 10301
La valutazione quantitativa dei procarioti può essere onerosa data la loro abbondanza, la propensione alla proliferazione esponenziale, la diversità delle specie all’interno di una popolazione e le specifiche esigenze fisiologiche. Ad aggravare questa sfida, è la natura a quattro fasi in cui i batteri si replicano (ritardo, log, stazionario e morte). La capacità di stimare con precisione la concentrazione di microrganismi è necessaria per il successo dell’identificazione, dell’isolamento, della coltivazione e della caratterizzazione (6). Pertanto, i microbiologi hanno impiegato la diluizione seriale e varie tecniche di placcatura per oltre un secolo per quantificare in modo affidabile la carica batterica e virale in ambienti di laboratorio clinici, industriali, farmaceutici e accademici (2,4,6). Le descrizioni di questa metodologia apparvero per la prima volta nel 1883 quando lo scienziato e medico tedesco Robert Koch pubblicò il suo lavoro sugli agenti infettivi che causano malattie (2). Spesso indicato come il padre della batteriologia moderna, le tecniche prenominate di Koch sono diventate il gold standard per l’enumerazione di microrganismi, coltivabili o meno, in tutto il mondo.
La diluizione seriale è una riduzione sistematica di un’entità nota o sconosciuta (un soluto, un organismo, ecc.) attraverso la successiva risos sospensione di una soluzione iniziale(soluzione 0) in volumi fissi di un diluente liquido (spazi vuoti). Questi spazi vuoti di solito consistono di 0,45% di soluzione salina, anche se la composizione può essere variata (7). Mentre uno sperimentatore può scegliere qualsiasi volume per ogni diluente, è più spesso un multiplo di 10, facilitando la riduzione logaritmica del campione. Ad esempio, lasoluzione 0 contiene un totale di 100 cellule di E. coli sospese in 10 ml di brodo nutritivo. Se 1 mL disoluzione 0 viene rimosso e aggiunto a 9 mL di soluzione salina (diluente1), la nuovasoluzione (soluzione 1) conterrebbe 1/10 della concentrazione iniziale di E. coli. In questo esempio, la nuova soluzione(soluzione 1) conterrebbe 10 cellule di E. coli. Ripetendo questo processo rimuovendo 1 mL disoluzione 1 e aggiungendolo ad altri 9 mL di soluzione salina (diluente2)si produrrebbela soluzione 2, contenente solo una singola cellula di E. coli. Poiché ogni nuova soluzione (9 mL di diluente + 1 mL di soluzione) contiene un totale di 10mL, possiamo concludere che il fattore di diluizione per questa riduzione è 10 o che si è tratta di una diluizione seriale 10 volte (Figura 1). Poiché abbiamo iniziato solo con 100 cellule in questo esempio e stiamo diluendo di un fattore 10, sono necessari solo due passaggi per raggiungere la concentrazione minima assoluta di 1 cellula.
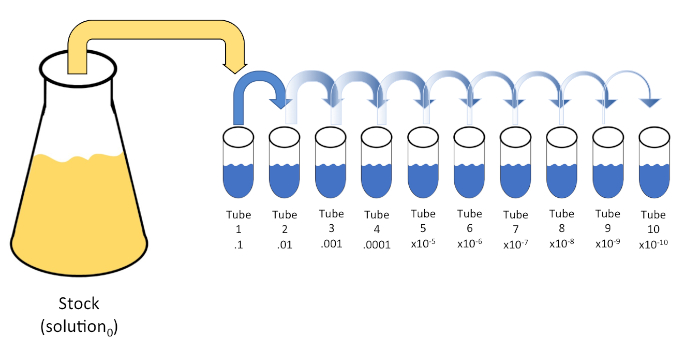
Figura 1: Diluizione seriale di una soluzione stock. Un’aliquota di 1 mL della soluzione stock(soluzione 0) viene aggiunta al tubo 1 che contiene 9 mL di soluzione salina allo 0,45% (dilent1); il prodotto di questa miscela è lasoluzione 1. Ripetere aliquotando 1 mL dellasoluzione appena creata 1 e aggiungendola al tubo 2. L’aliquotazione e la ricaspensione continuano in questo modo fino al raggiungimento del tubo finale, diluendo la concentrazione di stock di un fattore di 10 ciascuno ad ogni passo. Fare clic qui per visualizzare una versione più grande di questa figura.
La diluizione seriale è la tecnica più semplice per ottenere concentrazioni gestibili di un organismo desiderato ed è completata da striature e spalmature di Petri, solo due delle molte tecniche di placcatura utilizzate dai microbiologi. Questo vantaggio di questo approccio è che lo sperimentatore può raccogliere ceppi puri di una singola specie o ceppi separati da una popolazione mista (7). La striatura si ottiene introducendo un organismo in un mezzo solido (generalmente costituito da agarose) su cui crescerà se sono disponibili i nutrienti appropriati. Spazzare delicatamente un ciclo inoculante sterile attraverso il mezzo (in modo che rimanga una striscia sottile) in un modello sinusoidale rigido distribuirà l’organismo proporzionalmente alla frequenza della forma d’onda dello sperimentatore. Dividere la capsula di Petri in terzi o quarti (striscia di quadrante) e diminuire la frequenza di ogni striscia quando viene inserita una nuova regione del piatto ridurrà gradualmente il numero di microrganismi che possono occupare quella regione, producendo singole colonie invece di un prato batterico non quantificabile. La placcatura diffusa non diluisce ulteriormente i campioni; uno spandi vetro sterile viene utilizzato per distribuire un’aliquota di mezzi di sospensione su un’intera capsula di Petri (Figura 2). Le colonie che crescono sulla piastra diffusa derivano da una singola cellula e ogni colonia sul piatto può essere contata per stimare il numero di unità formanti colonie per millilitro (CFU) in una data sospensione, rappresentata come CFU/mL (6) (Figura 3) L’agar morbido e la replica placcatura sono variazioni delle tecniche di cui sopra e consentono l’isolamento del batteriofago e lo screening mutante, rispettivamente (1,7).
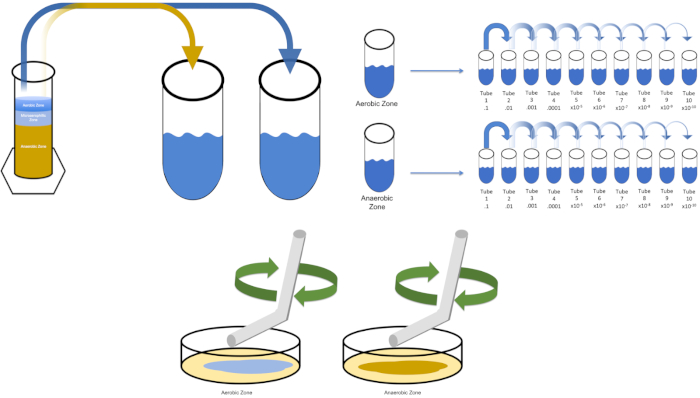
Figura 2: Striature di piastre per l’enumerazione batterica e l’isolamento del ceppo. Etichettare il fondo di una capsula di Petri con informazioni di identificazione (nome dei batteri, data, supporto) e dividere in terzi. Dopo aver selezionato una diluizione appropriata del campione di riserva, prendere un anello di inoculazione sterile (monouso o fiammato) e immergerlo nella provetta (qui, T3). Sollevare leggermente il coperchio della capsula di Petri su un lato in modo che solo l’anello inoculante possa accedere all’agar. Fai scivolare il loop inoculante attraverso la parte superiore del supporto a zig-zag facendo attenzione a non compromettere l’agar. Ruotare la piastra di circa 1/3 (~118°) e ridurre la frequenza del movimento a zig-zag. Ruota un ultimo tempo e riduci ancora una volta la frequenza a zig-zag. Fare clic qui per visualizzare una versione più grande di questa figura.
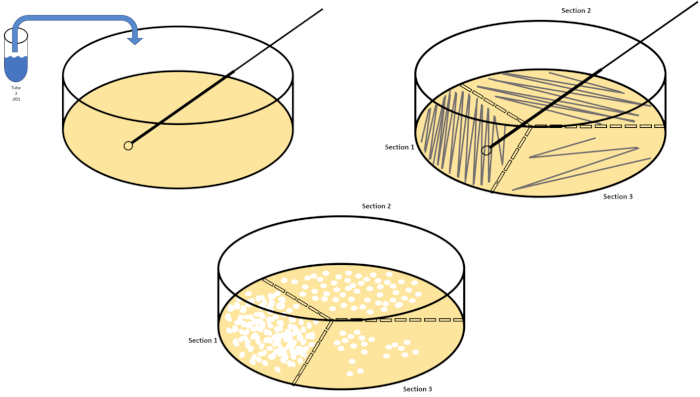
Figura 3: Placcatura diffusa. 1 g della zona aerobica è stato sospenso in T1 e poi diluito in serie. Un’asta sterile di spargimento monouso in vetro o plastica viene utilizzata per distribuire l’inoculo in ogni piatto. Questo è stato ripetuto con 1 g della zona anaerobica. Fare clic qui per visualizzare una versione più grande di questa figura.
Come per le diluizioni seriali, viene utilizzata una scala logaritmica per esprimere la concentrazione dell’organismo. Il numero di colonie coltivate in piastre di Petri standard di 100 mm x15 mm può essere enumerato manualmente (o automatizzato con l’aiuto dell’elaborazione computazionale) identificando cluster isolati di crescita. I conteggi che ammontano a meno di 30 o superiore a 300 devono essere definiti rispettivamente come troppo pochi da contare (TFTC) o troppo numerosi da contare (TNTC). Nel caso di quest’ultimo, deve essere eseguita una diluizione seriale per ridurre la concentrazione prima di riaffermire una nuova capsula di Petri. Facendo la media del numero di colonie autonome identificate da tre piastre di Petri separate e moltiplicando la media per il fattore di diluizione si otterrà CFU/mL; tracciare il log10 di CFU/mL contro il tempo rivelerà il tempo medio di generazione dell’organismo (7).
Procedure
Applications and Summary
Bacterial enumeration and strain isolation by plating requires manageable concentrations of target organisms. Successful plating is therefore contingent upon serial dilution. As such, the aforementioned techniques remain the cornerstone of microbiological examination and experimentation. Though simple by design, dilution factors and plating technique can be modified to by the experimenter to bolster outcomes without compromising the integrity of each method. Plotting the four phases of bacterial growth can be helpful when characterizing desired microbes. These phases, lag, log, stationary, and death, are marked by changes in bacterial replication. The lag phase features slow growth due to physiological adaptation, the log phase is the period of maximum proliferation featuring an exponential rise in viable cells, stationary phase is then reached due to environmental limitations and accumulations of toxins, before the death phase where cell counts begin to fall. This can be accomplished by serially diluting (or 1-step diluting to avoid confusion) Solution0 every hour for a total of 8 hours, beginning at Time0 (Solution0 should be returned to a shaking incubator after each dilution). Calculate the log10 of CFU/ml for a single diluent of Time0 and plot on the Y-axis. Repeat this calculation for the sample Time1 (make sure calculate CFU/mL using the same dilution factor as Time0). Repeat until each time (Time1-Time8) are plotted on the X-axis.
References
- Allen, M.E., Gyure, R.A. (2013) An Undergraduate Laboratory Activity Demonstrating Bacteriophage Specificity. Journal of Microbial Biological Education 14: 84-92.
- Ben-David, A., Davidson, C.E. (2014) Estimation Method for Serial Dilution Experiments. Journal of Microbiological Methods 107:214-221.
- Goldman, E., Green, L.H. (2008) Practical Handbook of Microbiology.
- Koch, R. (1883) New Research Methods for Detection of Microcosms in Soil, Air and Water.
- Lederberg, J., Lederberg, E.M. (1952) Replica Plating and Indirect Selection of Bacterial Mutants. Journal of Bacteriology 63:399-406
- Pepper, I., Gerba, C., Ikner, L. (2019) Bacterial Growth Curve Analysis and its Environmental Changes. JoVE Science Education Database. Environmental Microbiology.
- Sanders., E.R. (2012) Aseptic Laboratory Technique: Plating Methods. JoVE 63:e3063.
내레이션 대본
Sometimes, in order to identify and study bacteria we first need to isolate and enrich them from a sample. For example, samples obtained from a Winogradsky Column are mixed, meaning they contain multiple species or strains of bacteria, so studying an individual bacterium or enumerating the different kinds present can be challenging. To this end, serial dilution and plating techniques are typically employed to reliably quantify bacterial load and isolate individual colonies.
Serial dilution is a process through which the concentration of an organism, bacteria in this example, is systematically reduced through successive resuspension in fixed volumes of liquid diluent. Usually the volume of the diluent is a multiple of 10 to facilitate logarithmic reduction of the sample organism. For example, one gram of sediment is first removed from the Winogradsky zone of interest and added to 10 milliliters of an appropriate liquid medium. Then, one milliliter of this first dilution is added to another tube containing nine milliliters of medium. The process can be repeated until several different concentrations of bacteria have been prepared. Serial dilution is the key to enumeration of bacteria in this example, since mixed samples from a Winogradsky Column contain an unknown, often large, number of bacteria.
Next, streak plating and spread plating enable the isolation and enumeration of bacteria within a sample, respectively. Streaking is accomplished by introducing a diluted sample to one section of the solid medium supplemented with nutrient, which is divided into thirds. This inoculum is then spread over each third of the plate in a zig-zag pattern. As different sections of the plate are streaked, crossing from the previous sample only once, the sample is spread more thinly. This means that you may only need to streak from one dilution to achieve individual colonies in the later sections. After incubation, the streaked plates allow for observations of colony morphology, information that can help differentiate between different bacterial species.
Alternatively, if the main goal is the enumeration of the bacteria in the sample spread plating may be used. In spread plating, an aliquot of a single sample is spread evenly over the entire surface of solid medium. Typically, because we don’t know the bacterial numbers in the mixed sample, a spread plate is made for each of the dilutions or a representative sample of them. After incubation, enumeration can be performed using these spread plates. Any plates with colony counts fewer than 30 should be discarded, since small counts are subject to greater error. Similarly, any counts over 300 should be discarded because colony crowding and overlapping can lead to underestimation of colony count. If the colony counts of each of these remaining dishes is recorded and multiplied by the dilution factor, and then divided by the volume plated, this yields the colony forming units, or CFUs, per milliliter of suspension. In this video, you will learn how to qualitatively and quantitatively evaluate a sample containing a known bacterium, and the microbial communities contained in various regions of a Winogradsky Column via serial dilution, spread plating, and streak plating.
First, put on any appropriate personal protective equipment including a lab coat, gloves, and goggles. Next, sterilize the workspace with 70% ethanol and wipe down the surface. Next, gather two 500 milliliter Erlenmeyer flasks and label one broth and the other agar. To prepare LB agar solution, mix approximately 6.25 grams of LB agar, three grams of technical agar, and 250 milliliters of distilled water in the flask labeled agar.
Then, prepare LB broth by combining 2. 5 grams of LB media and 100 milliliters of distilled water in the flask labeled broth. After autoclaving the flasks, use a heat resistant glove to remove the flasks from the autoclave and place them in a 40 to 50 degree Celsius water bath. Once the flasks are 50 degrees Celsius, carefully prepare three 100 milliliter aliquots of the broth solution and label each aliquot solution zero. Next, gather 10 sterile petri dishes and label them with the date, name, type of media used, and the Winogradsky Column zone that the organisms will be harvested from. Pipette 15 milliliters of agar from the agar flask into each petri dish. Then, use the pipette tip to remove any bubbles, replace the plate lids, and allow them to solidify on the bench top overnight.
The next day, wipe down the bench top with 70% ethanol. Next, label 10 20 milliliter test tubes T1 through T10 and place them in a rack. Pipette nine milliliters of .45% saline into each tube. Now, cover each of the 10 test tubes loosely with their caps and transfer them to an autoclave-compatible test tube rack. After the cycle is complete, remove the saline blanks using heat resistant gloves and allow them to cool. Store the tubes at room temperature until they have reached approximately 22 degrees Celsius.
To cultivate a known target organism, E. coli in this example, inoculate 100 milliliters of solution zero with a single colony from a previously streaked plate. Then, cover the tube and incubate it over night at 37 degrees Celsius. To evaluate the regions of a Winogradsky Column, add approximately one gram of material from the aerobic zone to T1 and resuspend by vortexing. Then, repeat this process with one gram of material from the anaerobic zone.
Remove the tube containing solution zero inoculated with E. coli from the incubator and shake it. Then, pipette one milliliter of the solution into a T1 test tube and vortex to mix. Remove one milliliter of solution from T1 and transfer it to T2, vortexing to mix. Repeat this process through tube T10. To evaluate the aerobic and anaerobic zones of the Winogradsky Column, remove one milliliter of solution from each of the previously prepared T1 tubes and transfer it to the appropriate T2 tubes. Then, continue the serial dilutions through the T10 tubes as previously demonstrated.
To spread plate, pipette 100 microliters of the diluted sample from each T3 tube on to the corresponding petri dish. Then, use a sterile spreading rod to gently distribute the sample on to the petri dish and replace the plate lid. Repeat this process for the T6 and T9 dilutions, as previously demonstrated. Incubate the plates containing aerobic organisms in a 37 degree Celsius incubator for 24 hours. Incubate the plates containing anaerobic organisms in an anaerobic chamber set to 37 degrees Celsius for 24 hours. The next day remove the T3, T6, and T9 dilution plates from the incubator and the anaerobic chamber and transfer them to the bench top. Working with one plate at a time, glide a sterile inoculating loop across the top of the media in a zig-zag pattern. Then, replace the petri dish lid. Next, rotate the plate by 1/3 and sterilize the loop to reduce the frequency of the previously made zig-zag pattern. Again, after sterilizing the loop, rotate the plate by 1/3, reduce the frequency of the zig-zag pattern one last time, and replace the lid. Repeat this streaking method for the remaining plates, as previously shown. Then, place the streaked plates containing aerobic organisms in a 37 degree Celsius incubator overnight and the streaked plates containing anaerobic organisms in an anaerobic chamber set to 37 degrees Celsius overnight.
Cultures were harvested from the aerobic and anaerobic zones of a seven day Winogradsky Column. Then, the cultures were serially diluted prior to streaking and spreading on LB agar plates. Streaking revealed a mixed population from each of the evaluated Winogradsky zones, and the spread plates produced similar results. A plate streaked from a mixed population will result in bacterial colonies of different shapes, sizes, textures, and colors. In contrast, the streaked and spread plates containing the known organism, E. coli, demonstrated a homologous population. Generally, it is best to calculate CFUs per milliliter using the average colony count of three plates spread with the same sample and dilution factor. Multiply the average number of colonies by the dilution factor and divide by the amount aliquoted. Finally, isolated colonies chosen from each plate can be used in further enrichment assays to determine species identity.
