Transformation von E. coli-Zellen mit einem adaptierten Calciumchlorid-Verfahren
English
소셜에 공유하기
개요
Quelle: Natalia Martin1, Andrew J. Van Alst1, Rhiannon M. LeVeque1, und Victor J. DiRita1
1 Department of Microbiology and Molecular Genetics, Michigan State University
Bakterien haben die Fähigkeit, genetisches Material (DeoxyriboNucleic Acid, DNA) in einem Prozess auszutauschen, der als horizontaler Gentransfer bekannt ist. Die Einbeziehung exogener DNA bietet einen Mechanismus, mit dem Bakterien neue genetische Merkmale erwerben können, die es ihnen ermöglichen, sich an sich verändernde Umweltbedingungen anzupassen, wie das Vorhandensein von Antibiotika oder Antikörpern (1) oder Molekülen, die in natürlichen Lebensräumen gefunden werden (2). Es gibt drei Mechanismen des horizontalen Gentransfers: Transformation, Transduktion und Konjugation (3). Hier konzentrieren wir uns auf transformation, die Fähigkeit von Bakterien, freie DNA aus der Umwelt zu nehmen. Im Labor hat der Transformationsprozess vier allgemeine Schritte: 1) Vorbereitung kompetenter Zellen, 2) Inkubation kompetenter Zellen mit DNA, 3) Wiederherstellung der Zellen und 4) Beschichtung der Zellen für das Wachstum der Transformanten (Abbildung 1).
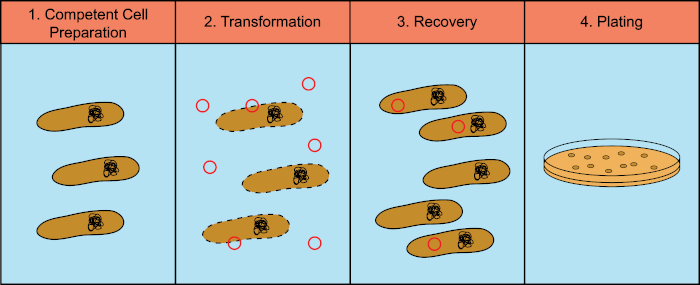
Abbildung 1: Allgemeine Schritte des Transformationsprozesses. Der Transformationsprozess besteht aus vier allgemeinen Schritten: 1) Vorbereitung kompetenter Zellen, 2) Inkubation mit DNA, 3) Wiederherstellung der Zellen und 4) Beschichtungszellen für das Wachstum der Transformanten.
Damit die Transformation stattfindet, müssen sich die Empfängerbakterien in einem Zustand befinden, der als Kompetenz bekannt ist. Einige Bakterien haben die Fähigkeit, natürlich kompetent als Reaktion auf bestimmte Umweltbedingungen zu werden. Jedoch, viele andere Bakterien werden nicht kompetent natürlich, oder die Bedingungen für diesen Prozess sind noch unbekannt. Die Fähigkeit, DNA in Bakterien einzuführen, hat eine Reihe von Forschungsanwendungen: mehrere Kopien eines DNA-Moleküls von Interesse zu erzeugen, eine große Menge an Proteinen auszudrücken, als Bestandteil in Klonverfahren, und andere. Aufgrund des Wertes der Transformation zur Molekularbiologie gibt es mehrere Protokolle, die darauf abzielen, Zellen künstlich kompetent zu machen, wenn Bedingungen für natürliche Kompetenz unbekannt sind. Zwei Hauptmethoden werden verwendet, um künstlich kompetente Zellen vorzubereiten: 1) durch chemische Behandlung von Zellen und 2) Die Zellen elektrischen Impulsen aussetzen (Elektroporation). Erstere verwendet je nach Verfahren unterschiedliche Chemikalien, um Anziehungskraft zwischen der DNA und der Zelloberfläche zu erzeugen, während letztere elektrische Felder verwendet, um Poren in der bakteriellen Zellmembran zu erzeugen, durch die DNA-Moleküle eindringen können. Der effizienteste Ansatz für die chemische Kompetenz ist die Inkubation mit divalenten Kationen, insbesondere Kalzium (Ca2+)(4,5) Calcium-induzierte Kompetenz ist das Verfahren, das hier beschrieben wird (6). Diese Methode wird hauptsächlich für die Transformation von Gram-negativen Bakterien verwendet, und das wird der Schwerpunkt dieses Protokolls sein.
Das Verfahren der chemischen Umwandlung umfasst eine Reihe von Schritten, in denen Zellen Kationen ausgesetzt sind, um chemische Kompetenz zu induzieren. Auf diese Schritte folgt anschließend eine Temperaturänderung – Hitzeschock -, die die Aufnahme fremder DNA durch die zuständige Zelle begünstigt (7). Die bakteriellen Zellhüllen sind negativ geladen. Bei gramnegativen Bakterien wie Escherichia coliist die äußere Membran aufgrund des Vorhandenseins von Lipopolysaccharid (LPS) negativ geladen (8). Dies führt zur Abstoßung der ähnlich negativ geladenen DNA-Moleküle. In der chemischen Kompetenzinduktion neutralisieren positiv geladene Calciumionen diese Ladungsabstoßung und ermöglichen die DNA-Absorption auf die Zelloberfläche (9). Die Kalziumbehandlung und Inkubation mit DNA erfolgt auf Eis. Anschließend wird eine Inkubation bei höheren Temperaturen (42°C), dem Hitzeschock, durchgeführt. Dieses Temperaturungleichgewicht begünstigt die DNA-Aufnahme weiter. Bakterielle Zellen müssen sich in der mittleren exponentiellen Wachstumsphase befinden, um der Hitzeschockbehandlung standzuhalten; in anderen Wachstumsstadien sind die Bakterienzellen zu empfindlich gegenüber der Hitze, was zu einem Verlust der Lebensfähigkeit führt, was die Transformationseffizienz deutlich verringert.
Für die Transformation können verschiedene DNA-Quellen verwendet werden. Typischerweise werden Plasmide, kleine kreisförmige, doppelsträngige DNA-Moleküle, für die Transformation in den meisten Laborverfahren in E. coli verwendet. Damit Plasmide nach der Transformation in der Bakterienzelle erhalten bleiben können, müssen sie einen Ursprung der Replikation enthalten. Dadurch können sie unabhängig vom bakteriellen Chromosom in der Bakterienzelle repliziert werden. Nicht alle Bakterienzellen werden während des Transformationsvorgangs transformiert. So ergibt die Transformation eine Mischung aus transformierten Zellen und nicht transformierten Zellen. Um zwischen diesen beiden Populationen zu unterscheiden, wird eine Selektionsmethode verwendet, um die Zellen zu identifizieren, die das Plasmid erworben haben. Plasmide enthalten in der Regel wählbare Marker, d. h. Gene, die eine Eigenschaft kodieren, die einen Vorteil für das Wachstum verleiht (d. h. Resistenz gegen ein Antibiotikum oder eine Chemikalie oder Rettung aus einer Wachstumsauxotrophie). Nach der Transformation werden Bakterienzellen auf selektiven Medien plattiert, was nur das Wachstum der transformierten Zellen ermöglicht. Im Falle von Zellen, die mit einem Plasmid transformiert werden, das eine Resistenz gegen ein bestimmtes Antibiotikum verleiht, werden die selektiven Medien Wachstumsmedien sein, die dieses Antibiotikum enthalten. Verschiedene Methoden können verwendet werden, um zu bestätigen, dass die in den selektiven Medien angebauten Kolonien Transformanten sind (d.h. das Plasmid eingebaut haben). Beispielsweise können Plasmide aus diesen Zellen mit Plasmidpräparationsmethoden (10) zurückgewonnen und verdaut werden, um die Plasmidgröße zu bestätigen. Alternativ kann Kolonie-PCR verwendet werden, um das Vorhandensein des Plasmids von Interesse zu bestätigen (11).
Ziel dieses Experiments ist es, chemisch kompetente E. coli DH5-Zellen unter Verwendung einer Anpassung des Calciumchloridverfahrens (12) vorzubereiten und mit dem Plasmid pUC19 zu transformieren, um die Transformationseffizienz zu bestimmen. Der E. coli-Stamm DH5 ist ein häufig verwendeter Stamm in molekularbiologischen Anwendungen. Aufgrund seines Genotyps, insbesondere des recA1 und des endA1,ermöglicht dieser Stamm eine erhöhte Insert-Stabilität und verbessert die Qualität der Plasmid-DNA in nachfolgenden Präparaten. Da die Transformationseffizienz mit zunehmender Größe der DNA abnimmt, wurde das Plasmid pUC19 in diesem Protokoll wegen seiner geringen Größe (2686 bp) verwendet (siehe https://www.mobitec.com/cms/products/bio/04_vector_sys/standard_cloning_vectors.html für eine Vektorkarte). pUC19 verleiht Resistenz gegen Ampicillin und somit war dies das Antibiotikum, das zur Selektion verwendet wurde.
Procedure
Results
Although TE is dependent on many factors, non-commercial competent cell preparations, like this one, normally yield 106 to 107 transformants per microgram of plasmid. Therefore, this preparation, with a TE = 2.46 x 108 cfu/µg, yielded a TE well beyond the expected range. Additional protocols are available for making supercompetent cells when higher transformation efficiencies are required for a given application (13).
Analysis of the digestion of the plasmid DNA recovered from the transformed cells indicated that this plasmid has the expected size of pUC19 DNA (2686 bp).
Applications and Summary
Transformation is a powerful method for introducing exogenous DNA into bacterial cells that is key to many molecular biology applications in the laboratory. Additionally, it plays a major role in nature by allowing bacterial cells to exchange genetic material that could result in increased genetic variation and allow for acquisition of different beneficial traits for survival under a wide range of conditions. Many bacterial strains encode the genes required for natural competence. However, the conditions in which these genes are induced are still unknown. Further research is required to determine these conditions.
References
- Croucher, N. J. et al. Rapid pneumococcal evolution in response to clinical interventions. Science. 331 (6016):430-434. (2011)
- Borgeaud, S. et al. The type VI secretion system of Vibrio cholerae fosters horizontal gene transfer. Science. 347(6217):63-67. (2015)
- Burmeister, A. R. Horizontal Gene Transfer. Evol Med Public Health. 2015 (1):193-194. (2015)
- Weston A, Brown MG, Perkins HR, Saunders JR, Humphreys GO. Transformation of Escherichia coli with plasmid deoxyribonucleic acid: calcium-induced binding of deoxyribonucleic acid to whole cells and to isolated membrane fractions. J Bacteriol. 145 (2):780-7. (1981)
- Dagert M, Ehrlich SD. Prolonged incubation in calcium chloride improves the competence of Escherichia coli cells. Gene. 6 (1):23-8. (1979)
- Asif A, Mohsin H, Tanvir R, and Rehman Y. Revisiting the Mechanisms Involved in Calcium Chloride Induced Bacterial Transformation. Front Microbiol. 8:2169. (2017)
- Panja S, Aich P, Jana B, Basu T. How does plasmid DNA penetrate cell membranes in artificial transformation process of Escherichia coli? Mol Membr Biol. 25 (5):411-22. (2008)
- Silhavy, TJ, Kahne D, Walker S. The Bacterial Cell Envelope. Cold Spring Harb Perspect Biol. 2 (5): a000414. (2010)
- Panja S, Aich P, Jana B, Basu T. (2008) Plasmid DNA binds to the core oligosaccharide domain of LPS molecules of E. coli cell surface in the CaCl2-mediated transformation process. Biomacromolecules. 9 (9):2501-9.
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. Plasmid Purification. JoVE, Cambridge, MA. (2018)
- Bergkessel M and Guthrie C. Colony PCR. Methods in Enzymology. 529: 299-309. (2013)
- Sambrook J and Russell DW. Molecular Cloning A Laboratory Manual. Cold Spring Harbor Laboratory Press, Cold Spring Harbor, NY.Protocol 25 (1.116-118). (2001)
- Wirth R, Friesenegger A, Fiedler S. Transformation of various species of gram-negative bacteria belonging to 11 different genera by electroporation. Molecular & General Genetics. 216 (1): 175-7. (1989)
내레이션 대본
Bacteria are remarkably adaptable and one mechanism which facilitates this adaptation is their ability to take in external DNA molecules. One type of DNA that bacteria can uptake is called a plasmid, a circular piece of DNA that frequently contains useful information, such as antibiotic resistance genes. The process of bacteria being modified by new genetic information incorporated from an external source is referred to as transformation. Transformation can easily be performed in the laboratory using Escherichia coli, or E. coli.
In order to be transformed, E. coli cells must first be made competent, which means capable of taking in DNA molecules from their environment. The protocol for accomplishing this is surprisingly simple, a short incubation of the cells in a calcium chloride solution. This incubation causes the cells to become permeable to DNA molecules. After the cells are pelleted by centrifugation, the supernatant is removed. The plasmid DNA is now added to the competent cells. After incubating the cells with DNA, the mix is briefly heated to 42 degrees Celsius, followed by rapid cooling on ice. This heat shock causes the DNA to be transferred across the cell’s wall and membranes. The cells are then incubated in fresh media. Then, the bacteria are placed at 37 degrees to allow them to reseal their membranes and express resistant proteins.
Those cells which have taken in the plasmids will faithfully copy the DNA and pass it to their progeny and express any proteins that might be encoded by it, including antibiotic resistance mediators. Those resistance genes can be used as selectable markers to identify bacteria which have been successfully transformed because cells that have not taken up the plasmid will not express the resistance gene product. This means that when the cells are plated on a solid medium which contains the appropriate antibiotic, only cells that have taken up the plasmid will grow. Transformation of the cells in a growing colony can be further confirmed by culturing those cells in liquid media overnight to increase the yield before extracting the DNA from the sample. Once the DNA is isolated, a diagnostic restriction enzyme digest can be carried out. Because restriction enzymes cut DNA in predictable locations, running these digests on a gel should show a predictable pattern if the desired plasmid was successfully transformed. For example, if pUC19 is prepared and cut with the restriction enzyme HindIII, a single band of 2686 nucleotides should be seen on the gel.
In this lab, you will transform E. coli strain DH-5 Alpha with pUC19, and then confirm the successful transformation by DNA gel electrophoresis.
Before starting the procedure, put on the appropriate personal protective equipment, including a lab coat and gloves. Next, sterilize the workspace with 70% ethanol.
Now, prepare chemically competent cells by depositing a loopfull of bacteria onto a sterile LB agar plate and streaking the bacteria with a new loop. Then, incubate the plate at 37 degrees Celsius overnight. The next day, sterilize the bench top with 70% ethanol again, and remove the plate from the incubator.
Inoculate a single, well-isolated colony into 3 milliliters of LB broth in a tube with a sterile loop. Then, grow the culture at 37 degrees Celsius overnight, with shaking at 210 RPM. The next day, measure the optical density of the overnight culture with a spectrophotometer. Then, add 100 milliliters of LB broth to a one-liter flask, and inoculate it with the overnight culture at an optical density of 0. 01. Now, incubate the culture at 37 degrees Celsius with shaking, and check the OD600 every 15 to 20 minutes until the culture reaches mid-exponential growth phase.
After approximately three hours, transfer 50 milliliters of the culture to two ice-cold polypropylene bottles. Then, place the bottles back on ice for 20 minutes to cool. Next, recover the cells via centrifugation. Discard the supernatants and place the bottles upside down on a paper towel. Next, resuspend the bacterial pellet in five milliliters of ice-cold calcium chloride magnesium chloride solution and swirl carefully until the pellet has dissolved completely. Then, add another 25 milliliters of the solution to the dissolved bacterial pellet. Resuspend the other bacterial pellet as previously demonstrated. After this, repeat the centrifugation, and remove the supernatants.
If the competent cells are going to be directly transformed, resuspend each bacterial pellet in two milliliters of an ice-cold 0.1 molar calcium chloride solution by swirling the tubes carefully. To begin the transformation procedure, transfer 50 microliters of competent cells to two labeled 1.5 milliliter polypropylene tubes. Then, add one microliter of pUC19 plasmid DNA to one of the tubes. Mix gently, avoiding bubble formation, and incubate both tubes for 30 minutes on ice. After incubation, transfer the tubes to a heat block and incubate at 42 degrees Celsius for 45 seconds. Immediately transfer the tubes to ice, and incubate for two minutes. Now, add 950 microliters of SOC media to each tube and incubate them for one hour at 37 degrees Celsius to allow the bacteria to recover, and express the antibiotic resistant marker encoded in the plasmid.
To make a 1 to 100 dilution, add 990 microliters of SOC media and 10 microliters of cell suspension to a 1.5 milliliter tube. Then, make a 1 to 10 dilution by adding 900 microliters of SOC media and 100 microliters of cell suspension to a 1.5 milliliter tube. Next, plate 100 microliters of the diluted cell suspensions and 100 microliters of the negative control, onto separate selective plates containing ampicillin using a spreader and incubate the plates at 37 degrees Celsius for 12 to 16 hours. After incubation, count the colony-forming units, or CFUs, per plate, obtained through transformation, and record these data. To verify that the transformants have the pUC19 plasmid, pick a single, well-isolated colony from a plate with a sterile loop, and introduce it to a tube containing 3 milliliters of LB broth. Then, incubate the culture at 37 degrees Celsius with shaking, overnight. The next day, use a DNA mini prep kit to isolate DNA from 3 milliliters of the culture, according to the manufacturer’s instructions. After completing the DNA mini prep, digest the 1 microgram of purified pUC19 with a restriction enzyme at 37 degrees Celsius for 1 hour. Now, load 20 microliters of a molecular weight ladder, 1 microgram of digested plasmid DNA, and 1 microgram of undigested plasmid DNA into consecutive wells of a 1% agarose gel containing 1 microgram per milliliter ethidium bromide. Then, run the gel for 1 hour at 95 volts. Finally, visualize the gel with a UV illuminator.
In this experiment, E. coli DH5 Alpha chemically competent cells were prepared using an adaptation of the calcium chloride procedure, and then transformed with the plasmid pUC19 to determine transformation efficiency. To calculate the transformation efficiency, use the recorded CFU counts for the 1 in 100 and 1 in 10 dilutions, and any other dilutions with CFU counts between 30 and 300. First, the recorded CFU count, 246 in this example, is divided by the amount of DNA, .0001 micrograms here, that was plated. Then, this number is divided by the dilution factor used to give the transformation efficiency in CFUs per microgram. In this example, a 1 to 10 dilution was used and 100 microliters of a 1 milliliter solution was plated, giving a final dilution factor of 0.01. In the undigested plasmid lane, the circular DNA may appear as two or three different bands of varying brightness. This is because the circular, uncut DNA may exist in several different conformation states, such as supercoiled, open circle, or more linear, and each of these move through the gel at different rates. Analysis of the recovered plasmid DNA digestion indicated that the plasmid used has an expected size of pUC19 DNA, 2,686 base pairs.
