Transformación de células E. coli mediante un procedimiento con cloruro de calcio
English
소셜에 공유하기
개요
Fuente: Natalia Martin1, Andrew J. Van Alst1, Rhiannon M. LeVeque1, y Victor J. DiRita1
1 Departamento de Microbiología y Genética Molecular de la Universidad Estatal de Michigan
Las bacterias tienen la capacidad de intercambiar material genético (ácido desoxirribonucleico, ADN) en un proceso conocido como transferencia de genes horizontal. La incorporación de ADN exógeno proporciona un mecanismo mediante el cual las bacterias pueden adquirir nuevos rasgos genéticos que les permiten adaptarse a las condiciones ambientales cambiantes, como la presencia de antibióticos o anticuerpos (1) o moléculas que se encuentran en hábitats naturales (2). Existen tres mecanismos de transferencia génica horizontal: transformación, transducción y conjugación (3). Aquí nos centraremos en la transformación, la capacidad de las bacterias para tomar ADN libre del medio ambiente. En el laboratorio, el proceso de transformación consta de cuatro pasos generales: 1) Preparación de células competentes, 2) Incubación de células competentes con ADN, 3) Recuperación de células y 4) Placado de las células para el crecimiento de los transformadores (Figura 1).
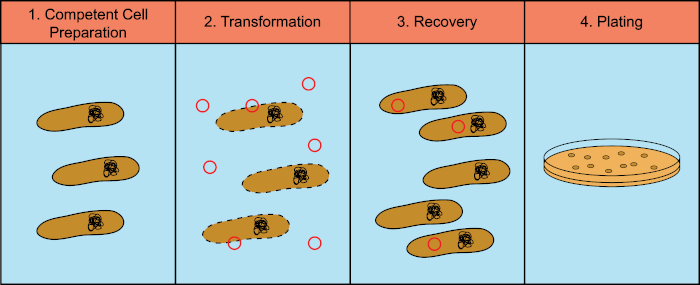
Figura 1: Pasos generales del proceso de transformación. El proceso de transformación tiene cuatro pasos generales: 1) Preparación de células competentes, 2) Incubación con ADN, 3) Recuperación de las células y 4) Células de chapado para el crecimiento de los transformadores.
Para que se produzca la transformación, las bacterias receptoras deben estar en un estado conocido como competencia. Algunas bacterias tienen la capacidad de llegar a ser naturalmente competentes en respuesta a ciertas condiciones ambientales. Sin embargo, muchas otras bacterias no se vuelven competentes naturalmente, o las condiciones para este proceso son aún desconocidas. La capacidad de introducir ADN en bacterias tiene una gama de aplicaciones de investigación: generar múltiples copias de una molécula de ADN de interés, para expresar una gran cantidad de proteínas, como un componente en los procedimientos de clonación, y otros. Debido al valor de la transformación a la biología molecular, existen varios protocolos destinados a hacer que las células sean artificialmente competentes cuando se desconocen las condiciones de competencia natural. Se utilizan dos métodos principales para preparar células artificialmente competentes: 1) a través del tratamiento químico de las células y 2) la exposición de las células a pulsos eléctricos (electroporación). El primero utiliza diferentes productos químicos dependiendo del procedimiento para crear atracción entre el ADN y la superficie celular, mientras que el segundo utiliza campos eléctricos para generar poros en la membrana celular bacteriana a través de la cual pueden entrar las moléculas de ADN. El enfoque más eficiente para la competencia química es la incubación con cationes divalentes, sobre todo calcio (Ca2+) (4,5) Competencia inducida por calcio es el procedimiento que se describirá aquí (6). Este método se utiliza principalmente para la transformación de bacterias Gram-negativas, y ese será el foco de este protocolo.
El procedimiento de transformación química implica una serie de pasos en los que las células están expuestas a cationes para inducir la competencia química. Estos pasos son seguidos posteriormente por un cambio de temperatura – choque de calor – que favorece la absorción de ADN extraño por la célula competente (7). Los sobres de las células bacterianas se cargan negativamente. En bacterias Gram-negativas como Escherichia coli, la membrana externa se carga negativamente debido a la presencia de lipopolisacárido (LPS) (8). Esto resulta en la repulsión de las moléculas de ADN cargadas negativamente de manera similar. En la inducción de competencia química, los iones de calcio cargados positivamente neutralizan esta repulsión de carga permitiendo la absorción del ADN en la superficie celular (9). El tratamiento del calcio y la incubación con ADN se realizan sobre hielo. Posteriormente, se realiza una incubación a temperaturas más altas (42oC), el choque de calor. Este desequilibrio de temperatura favorece aún más la aceptación del ADN. Las células bacterianas deben estar en la fase de crecimiento exponencial medio para soportar el tratamiento de choque térmico; en otras etapas de crecimiento las células bacterianas son demasiado sensibles al calor, lo que resulta en una pérdida de viabilidad que disminuye significativamente la eficiencia de transformación.
Diferentes fuentes de ADN se pueden utilizar para la transformación. Típicamente, plásmidos, pequeñas moléculas circulares de ADN de doble cadena, se utilizan para la transformación en la mayoría de los procedimientos de laboratorio en E. coli. Para que los plásmidos se mantengan en la célula bacteriana después de la transformación, deben contener un origen de replicación. Esto permite que se repliquen en la célula bacteriana independientemente del cromosoma bacteriano. No todas las células bacterianas se transforman durante el procedimiento de transformación. Por lo tanto, la transformación produce una mezcla de células transformadas y células no transformadas. Para distinguir entre estas dos poblaciones, se utiliza un método de selección para identificar las celdas que han adquirido el plásmido. Los plásmidos suelen contener marcadores seleccionables, que son genes que codifican un rasgo que confiere una ventaja para el crecimiento (es decir, resistencia a un antibiótico o químico o rescate de una auxotrofia de crecimiento). Después de la transformación, las células bacterianas se chapan en medios selectivos, lo que sólo permite el crecimiento de las células transformadas. En el caso de las células transformadas con un plásmido que confiere resistencia a un antibiótico dado, los medios selectivos serán medios de crecimiento que contengan ese antibiótico. Se pueden utilizar varios métodos diferentes para confirmar que las colonias cultivadas en los medios selectivos son transformadores (es decir, han incorporado el plásmido). Por ejemplo, los plásmidos pueden recuperarse de estas células utilizando métodos de preparación de plásmidos (10) y digeridos para confirmar el tamaño del plásmido. Alternativamente, la PCR de colonia se puede utilizar para confirmar la presencia del plásmido de interés (11).
El objetivo de este experimento es preparar las células químicamente competentes de E. coli DH5, utilizando una adaptación del procedimiento de cloruro de calcio (12), y transformarlas con el plásmido pUC19 para determinar la eficiencia de transformación. La cepa de E. coli DH5 es una cepa comúnmente utilizada en aplicaciones de biología molecular. Debido a su genotipo, específicamente el recA1 y endA1, esta cepa permite una mayor estabilidad de la plaquita y mejorar la calidad del ADN plásmido en preparaciones posteriores. Dado que la eficiencia de transformación disminuye con el aumento del tamaño del ADN, el plásmido pUC19 se utilizó en este protocolo debido a su pequeño tamaño (2686 bp) (ver https://www.mobitec.com/cms/products/bio/04_vector_sys/standard_cloning_vectors.html para un vectores). pUC19 confiere resistencia a la ampicilina y, por lo tanto, este fue el antibiótico utilizado para la selección.
Procedure
Results
Although TE is dependent on many factors, non-commercial competent cell preparations, like this one, normally yield 106 to 107 transformants per microgram of plasmid. Therefore, this preparation, with a TE = 2.46 x 108 cfu/µg, yielded a TE well beyond the expected range. Additional protocols are available for making supercompetent cells when higher transformation efficiencies are required for a given application (13).
Analysis of the digestion of the plasmid DNA recovered from the transformed cells indicated that this plasmid has the expected size of pUC19 DNA (2686 bp).
Applications and Summary
Transformation is a powerful method for introducing exogenous DNA into bacterial cells that is key to many molecular biology applications in the laboratory. Additionally, it plays a major role in nature by allowing bacterial cells to exchange genetic material that could result in increased genetic variation and allow for acquisition of different beneficial traits for survival under a wide range of conditions. Many bacterial strains encode the genes required for natural competence. However, the conditions in which these genes are induced are still unknown. Further research is required to determine these conditions.
References
- Croucher, N. J. et al. Rapid pneumococcal evolution in response to clinical interventions. Science. 331 (6016):430-434. (2011)
- Borgeaud, S. et al. The type VI secretion system of Vibrio cholerae fosters horizontal gene transfer. Science. 347(6217):63-67. (2015)
- Burmeister, A. R. Horizontal Gene Transfer. Evol Med Public Health. 2015 (1):193-194. (2015)
- Weston A, Brown MG, Perkins HR, Saunders JR, Humphreys GO. Transformation of Escherichia coli with plasmid deoxyribonucleic acid: calcium-induced binding of deoxyribonucleic acid to whole cells and to isolated membrane fractions. J Bacteriol. 145 (2):780-7. (1981)
- Dagert M, Ehrlich SD. Prolonged incubation in calcium chloride improves the competence of Escherichia coli cells. Gene. 6 (1):23-8. (1979)
- Asif A, Mohsin H, Tanvir R, and Rehman Y. Revisiting the Mechanisms Involved in Calcium Chloride Induced Bacterial Transformation. Front Microbiol. 8:2169. (2017)
- Panja S, Aich P, Jana B, Basu T. How does plasmid DNA penetrate cell membranes in artificial transformation process of Escherichia coli? Mol Membr Biol. 25 (5):411-22. (2008)
- Silhavy, TJ, Kahne D, Walker S. The Bacterial Cell Envelope. Cold Spring Harb Perspect Biol. 2 (5): a000414. (2010)
- Panja S, Aich P, Jana B, Basu T. (2008) Plasmid DNA binds to the core oligosaccharide domain of LPS molecules of E. coli cell surface in the CaCl2-mediated transformation process. Biomacromolecules. 9 (9):2501-9.
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. Plasmid Purification. JoVE, Cambridge, MA. (2018)
- Bergkessel M and Guthrie C. Colony PCR. Methods in Enzymology. 529: 299-309. (2013)
- Sambrook J and Russell DW. Molecular Cloning A Laboratory Manual. Cold Spring Harbor Laboratory Press, Cold Spring Harbor, NY.Protocol 25 (1.116-118). (2001)
- Wirth R, Friesenegger A, Fiedler S. Transformation of various species of gram-negative bacteria belonging to 11 different genera by electroporation. Molecular & General Genetics. 216 (1): 175-7. (1989)
내레이션 대본
Bacteria are remarkably adaptable and one mechanism which facilitates this adaptation is their ability to take in external DNA molecules. One type of DNA that bacteria can uptake is called a plasmid, a circular piece of DNA that frequently contains useful information, such as antibiotic resistance genes. The process of bacteria being modified by new genetic information incorporated from an external source is referred to as transformation. Transformation can easily be performed in the laboratory using Escherichia coli, or E. coli.
In order to be transformed, E. coli cells must first be made competent, which means capable of taking in DNA molecules from their environment. The protocol for accomplishing this is surprisingly simple, a short incubation of the cells in a calcium chloride solution. This incubation causes the cells to become permeable to DNA molecules. After the cells are pelleted by centrifugation, the supernatant is removed. The plasmid DNA is now added to the competent cells. After incubating the cells with DNA, the mix is briefly heated to 42 degrees Celsius, followed by rapid cooling on ice. This heat shock causes the DNA to be transferred across the cell’s wall and membranes. The cells are then incubated in fresh media. Then, the bacteria are placed at 37 degrees to allow them to reseal their membranes and express resistant proteins.
Those cells which have taken in the plasmids will faithfully copy the DNA and pass it to their progeny and express any proteins that might be encoded by it, including antibiotic resistance mediators. Those resistance genes can be used as selectable markers to identify bacteria which have been successfully transformed because cells that have not taken up the plasmid will not express the resistance gene product. This means that when the cells are plated on a solid medium which contains the appropriate antibiotic, only cells that have taken up the plasmid will grow. Transformation of the cells in a growing colony can be further confirmed by culturing those cells in liquid media overnight to increase the yield before extracting the DNA from the sample. Once the DNA is isolated, a diagnostic restriction enzyme digest can be carried out. Because restriction enzymes cut DNA in predictable locations, running these digests on a gel should show a predictable pattern if the desired plasmid was successfully transformed. For example, if pUC19 is prepared and cut with the restriction enzyme HindIII, a single band of 2686 nucleotides should be seen on the gel.
In this lab, you will transform E. coli strain DH-5 Alpha with pUC19, and then confirm the successful transformation by DNA gel electrophoresis.
Before starting the procedure, put on the appropriate personal protective equipment, including a lab coat and gloves. Next, sterilize the workspace with 70% ethanol.
Now, prepare chemically competent cells by depositing a loopfull of bacteria onto a sterile LB agar plate and streaking the bacteria with a new loop. Then, incubate the plate at 37 degrees Celsius overnight. The next day, sterilize the bench top with 70% ethanol again, and remove the plate from the incubator.
Inoculate a single, well-isolated colony into 3 milliliters of LB broth in a tube with a sterile loop. Then, grow the culture at 37 degrees Celsius overnight, with shaking at 210 RPM. The next day, measure the optical density of the overnight culture with a spectrophotometer. Then, add 100 milliliters of LB broth to a one-liter flask, and inoculate it with the overnight culture at an optical density of 0. 01. Now, incubate the culture at 37 degrees Celsius with shaking, and check the OD600 every 15 to 20 minutes until the culture reaches mid-exponential growth phase.
After approximately three hours, transfer 50 milliliters of the culture to two ice-cold polypropylene bottles. Then, place the bottles back on ice for 20 minutes to cool. Next, recover the cells via centrifugation. Discard the supernatants and place the bottles upside down on a paper towel. Next, resuspend the bacterial pellet in five milliliters of ice-cold calcium chloride magnesium chloride solution and swirl carefully until the pellet has dissolved completely. Then, add another 25 milliliters of the solution to the dissolved bacterial pellet. Resuspend the other bacterial pellet as previously demonstrated. After this, repeat the centrifugation, and remove the supernatants.
If the competent cells are going to be directly transformed, resuspend each bacterial pellet in two milliliters of an ice-cold 0.1 molar calcium chloride solution by swirling the tubes carefully. To begin the transformation procedure, transfer 50 microliters of competent cells to two labeled 1.5 milliliter polypropylene tubes. Then, add one microliter of pUC19 plasmid DNA to one of the tubes. Mix gently, avoiding bubble formation, and incubate both tubes for 30 minutes on ice. After incubation, transfer the tubes to a heat block and incubate at 42 degrees Celsius for 45 seconds. Immediately transfer the tubes to ice, and incubate for two minutes. Now, add 950 microliters of SOC media to each tube and incubate them for one hour at 37 degrees Celsius to allow the bacteria to recover, and express the antibiotic resistant marker encoded in the plasmid.
To make a 1 to 100 dilution, add 990 microliters of SOC media and 10 microliters of cell suspension to a 1.5 milliliter tube. Then, make a 1 to 10 dilution by adding 900 microliters of SOC media and 100 microliters of cell suspension to a 1.5 milliliter tube. Next, plate 100 microliters of the diluted cell suspensions and 100 microliters of the negative control, onto separate selective plates containing ampicillin using a spreader and incubate the plates at 37 degrees Celsius for 12 to 16 hours. After incubation, count the colony-forming units, or CFUs, per plate, obtained through transformation, and record these data. To verify that the transformants have the pUC19 plasmid, pick a single, well-isolated colony from a plate with a sterile loop, and introduce it to a tube containing 3 milliliters of LB broth. Then, incubate the culture at 37 degrees Celsius with shaking, overnight. The next day, use a DNA mini prep kit to isolate DNA from 3 milliliters of the culture, according to the manufacturer’s instructions. After completing the DNA mini prep, digest the 1 microgram of purified pUC19 with a restriction enzyme at 37 degrees Celsius for 1 hour. Now, load 20 microliters of a molecular weight ladder, 1 microgram of digested plasmid DNA, and 1 microgram of undigested plasmid DNA into consecutive wells of a 1% agarose gel containing 1 microgram per milliliter ethidium bromide. Then, run the gel for 1 hour at 95 volts. Finally, visualize the gel with a UV illuminator.
In this experiment, E. coli DH5 Alpha chemically competent cells were prepared using an adaptation of the calcium chloride procedure, and then transformed with the plasmid pUC19 to determine transformation efficiency. To calculate the transformation efficiency, use the recorded CFU counts for the 1 in 100 and 1 in 10 dilutions, and any other dilutions with CFU counts between 30 and 300. First, the recorded CFU count, 246 in this example, is divided by the amount of DNA, .0001 micrograms here, that was plated. Then, this number is divided by the dilution factor used to give the transformation efficiency in CFUs per microgram. In this example, a 1 to 10 dilution was used and 100 microliters of a 1 milliliter solution was plated, giving a final dilution factor of 0.01. In the undigested plasmid lane, the circular DNA may appear as two or three different bands of varying brightness. This is because the circular, uncut DNA may exist in several different conformation states, such as supercoiled, open circle, or more linear, and each of these move through the gel at different rates. Analysis of the recovered plasmid DNA digestion indicated that the plasmid used has an expected size of pUC19 DNA, 2,686 base pairs.
