DNA甲基化:亚硫酸氢修改和分析
Summary
DNA甲基化分析的黄金标准是亚硫酸氢转换的DNA基因组测序。 5 – 甲基胞嘧啶(5 – MEC)亚硫酸氢在酸性条件下脱氨相比,这种方法需要利用胞嘧啶的敏感性增加。后PCR扩增目标基因组DNA甲基化的胞嘧啶甲基化的胞嘧啶可以区分。
Abstract
表观遗传学描述基因功能的遗传发生的变化,独立的DNA序列。后生基因表达调控的分子基础是复杂的,但本质上涉及到的DNA本身或与DNA联营蛋白质的修改。在哺乳动物的基因组DNA表观遗传修饰的主要是甲基化的胞嘧啶核苷酸(5 – MEC)。 DNA甲基化的基因表达机械提供指令时应该表达的基因。在哺乳动物DNA甲基化的主要目标序列的CpG – 5' – 3'二核苷酸(图1)。 CpG二核苷酸并非均匀地分布在整个基因组,但都集中在重复的基因组序列的CpG的,通常与基因启动子(图1)相关的“孤岛”的区域。 DNA甲基化模式是建立在发展初期,在组织特异性分化调制,并在包括癌症在内的许多疾病状态的干扰。到Understand的生物学作用的DNA甲基化,其作用在人类疾病中,精确,高效和可重复性的方法,检测和量化个人5 MECS。
这为亚硫酸氢转换的协议是“黄金标准”DNA甲基化分析,并便于识别和量化的DNA甲基化的单核苷酸分辨率。亚硫酸氢钠的胞嘧啶脱氨的化学包括三个步骤(图2)。 (1)磺化:除了胞嘧啶(2)水解脱氨5-6双键:对胞嘧啶亚硫酸氢产生的衍生给予尿嘧啶亚硫酸氢衍生物(3)碱Desulphonation水解脱氨的亚硫酸氢去除磺酸经碱处理组,给尿嘧啶。亚硫酸氢优先deaminates胞嘧啶尿嘧啶在单链DNA,而5 – MEC,是难治性亚硫酸氢介导的脱氨。 PCR扩增后,尿嘧啶是放大胸腺嘧啶,而5 – MEC的残留物仍然为胞嘧啶,允许由来自甲基化CpGs的胞嘧啶的“C”与胸腺嘧啶“T”的残留物的存在,在测序来加以区分甲基化CpGs。
DNA的亚硫酸氢盐转化的修改是一个完善的协议,可利用DNA甲基化分析的方法很多。由于5 – MEC检测亚硫酸氢转换是首次展示由Frommer 等 1和克拉克等。2,围绕基因组DNA帐户的亚硫酸氢转化为大多数对DNA甲基化的新数据为基础的方法。可利用不同的方法后PCR分析,取决于特异性和甲基化的决议要求程度。克隆和测序仍是最容易获得的方法,可以给整个DNA分子中的甲基化单核苷酸分辨率。
Protocol
Discussion
亚硫酸氢转换的DNA基因组测序的DNA甲基化分析是一个完善的和灵活的方法,便于识别和量化DNA甲基化的单核苷酸分辨率。然而,亚硫酸氢转换和随后的分析依赖的DNA已经完全符合每一个甲基化的胞嘧啶被deaminated尿嘧啶和甲基化的胞嘧啶只,其余未反应转换的前提。如果转换是不完整的,可能会出现问题分析,因为归正的甲基化的胞嘧啶甲基化的胞嘧啶可能会错误地解释。亚硫酸氢转换可以优化在不同阶段,最大限度地转化率。对于DNA全部转为首先必须是单链,使胞嘧啶残基暴露的亚硫酸氢离子。第一步,DNA变性,是至关重要的,可以是不完整的转换的源,如果DNA是不完全变性。为了确保的DNA完全变性是很重要的反应参数,包括清除所有相关的蛋白质,适当的盐分浓度,孵化温度和时间都适合保持在单链构象的DNA。重要的是用新鲜的3M NaOH和,以便有足够的孵化时间。如果DNA是抗变性,一些协议的修改,包括孵化时间延长至30分钟或碎片的DNA变性前,可促进DNA变性。此外,单链DNA(ssDNA的)可以重新退火双双链DNA(dsDNA)的过程中的亚硫酸氢转换反应。开展定期或不断在亚硫酸氢反应在较高的温度(90-95℃)的反应,可以帮助维修单链DNA。然而,重要的是要知道,在这些较高的温度大大加速DNA的降解。各种试剂也可以添加到扰乱日重新退火发送的DNA链,如尿素。
DNA和其质量的浓度也影响亚硫酸氢反应的效率和最终的PCR扩增产量。 DNA降解的亚硫酸氢转换协议的限制。化学处理的DNA引入单链DNA的各种链断裂和完整的亚硫酸氢转换,包括亚硫酸氢浓度高,潜伏期长的时间可以加速DNA的降解所需的一些苛刻的条件。标准反应条件下,DNA降解不但一个共同的问题,如果协议的修改出台,如难治性转换的序列,如FFPE样品的质量较差或退化的DNA模板,然后DNA降解可以成为一个显著的限制。
福尔马林固定,石蜡包埋(FFPE)和退化的DNA样本,可以有效地亚硫酸氢使用此协议的转换和放大,但是建议修改,以确保DNA是不是进一步退化。 如 t RNA的载体RNA的建议使用。也糖原时,可以添加为载体DNA浓度低(<200吴)。因为从FFPE组织中分离出的DNA已经分散和进一步分散在脱氨的地方,必须小心,以尽量减少进一步恶化。不要任何进一步的片段DNA变性和限制前孵育时间为4个小时的亚硫酸氢反应。设计扩增不超过300个基点,由于分散的DNA。
另一个因素可以影响PCR扩增甲基化胞嘧啶的亚硫酸氢转换,降低DNA模板的复杂性。在4.1节中,增加引物的长度和巢式PCR所述以下的引物设计的协议都可以帮助增加特异性和PCR扩增产量。
最后,由于DNA模板改变在bisulphITE转换,放大偏置可关注。确保检查比例的甲基化和甲基化的模板的PCR扩增与控制50/50 / ü DNA混合,如在协议中所述,见图3。此外,还要确保只有转换模板的扩增,是发生使用HpaIII限制性内切酶和凝胶电泳(图3)。 PCR反应条件,可能需要不同的温度和/或镁离子浓度或延长延伸时间进行了优化。有些模板出现问题尤其严重,引物的位置可能需要调整或简并引物,可能需要使用。
下一代测序技术正在迅速发展,实现了类似的决议,以亚硫酸氢的基因组测序,第三代单分子测序的潜力,让小学的DNA甲基化的分析,而不需要为亚硫酸氢盐测序。然而,广泛的可用性和规格ificity这些技术仍然尚需时日。亚硫酸氢盐基因组测序中的甲基化分析的黄金标准,是一个强大的协议。该方法已取得进展,已解决共同的问题和局限性和应用程序从个人的基因分析扩大到高吞吐量和克拉克等人所描述的整个基因组 的分析,2006年4。表1列出故障排除指南和其他建议。这是建议的最佳方法是最初遵循的标准协议,只介绍修改,如果在出现问题时。
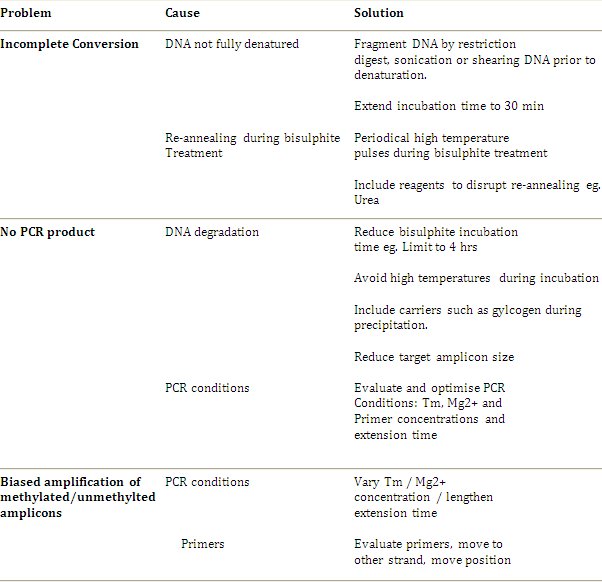
表1故障排除准则。
Declarações
The authors have nothing to disclose.
Materials
- Sodium Metabisulphite (Ajax Finechem)
- Hydroquinone (Merck)
- Wizard DNA-clean-up system (Promega)
Referências
- Frommer, M. A genomic sequencing protocol that yields a positive display of 5-methylcytosine residues in individual DNA strands. Proc. Natl. Acad. Sci. U. S. A. 89, 1827-1831 (1992).
- Clark, S. J. High sensitivity mapping of methylated cytosines. Nucleic. Acids. Res. 22, 2990-2997 (1994).
- Grunau, C., Clark, S. J., Rosenthal, A. Bisulfite genomic sequencing: systematic investigation of critical experimental parameters. Nucleic. Acids. Res. 29, E65-E65 (2001).
- Clark, S. J. DNA methylation: bisulphite modification and analysis. Nat. Protoc. 1, 2353-2364 (2006).

