从快速荧光成像在分子扩散法对活细胞的膜在商业显微镜
Summary
Spatial distribution and temporal dynamics of plasma membrane proteins and lipids is a hot topic in biology. Here this issue is addressed by a spatio-temporal image fluctuation analysis that provides conceptually the same physical quantities of single particle tracking, but it uses small molecular labels and standard microscopy setups.
Abstract
它已成为越来越明显,空间分布和膜元件,如脂质和蛋白质的运动是关键因素在许多细胞功能的调节。然而,由于快速的动力学和所涉及的微小结构,非常高的时空分辨率,需要捕捉分子的真实行为。这里,我们提出的实验方案用于研究活细胞中以高时空分辨率荧光标记的质膜蛋白质和脂质的动态。值得注意的是,这种方法并不需要跟踪每个分子,但它计算使用的所有分子在膜中的一个给定区域的人口行为。其出发点是在膜上某一区域的快速成像。之后,一个完整的时空自相关函数计算在增加的时间延迟,例如每2,3中,n次重复关联采集的图像。因此能够证明该宽度的在增加的时间延迟作为粒子运动的函数,由于扩散空间自相关函数的增加的峰值。因此,串联的自相关函数的拟合使得能够提取实际蛋白质意味着从成像(IMSD)方位移,这里提出的表观扩散系数比平均位移的形式。这就产生了单分子与纳米精度的平均动态的定量看法。通过使用转铁蛋白受体(TFR)和一个ATTO488的GFP标记的变体标记的1 -棕榈酰基-2 -羟基- SN -glycero-3-磷酸乙醇胺(PPE),可以观察到的蛋白质和脂质扩散的时空调控上微米尺寸的膜的区域中的微至毫秒的时间范围内。
Introduction
从原来的“流体镶嵌”模型歌手和尼科尔森开始,细胞质膜的画面不断在过去几十年更新,以包括细胞骨架和脂质域1,2的新兴角色。
第一观察由荧光漂白恢复(FRAP)揭开该膜蛋白的显著分数是不动的3-5后得到。这些开创性的研究,虽然非常丰富,受到FRAP设置在空间(微米)相对较差的分辨率和时间(秒)。此外,作为一个统计平均测量,FRAP缺乏使单分子行为的信息。
在这种情况下,有可能以特别标示具有非常明亮的标记(允许在扩散过程中的一个分子在一个时间的研究)的单个分子已经非常成功。特别是,通过推动单粒子追踪(SPT)的方法来微秒的时间尺度,久住,等脂质和蛋白质动力学的未知功能,极大地促进了表彰肌动蛋白为基础的膜骨架的作用在细胞膜的生理6 人获得了进入的时间分辨率,7。这些发现产生了所谓的“尖桩和栅栏”的模式,在其中的脂质和蛋白质的扩散是通过基于肌动蛋白骨架的调节。然而,为了能够访问大量的由SPT许多实验的问题所提供的信息必须得到解决。具体地讲,标记过程通常是通过许多步骤,如标记物种进入该系统的生产,纯化和介绍组成。此外,大的标签,如量子点,金属纳米颗粒,通常需要达到亚毫秒级的时间尺度与目标分子的标记的交联无法避免在许多情况下。最后,许多轨迹已被记录,以符合统计标准和伴随低密度标签的需要,以允许跟踪。
相比SPT,荧光相关光谱(FCS),克服许多这些缺点,是一种非常有前途的方法来研究分子动力学。事实上,FCS的效果很好也有暗淡而密的标签,使研究荧光蛋白标记的分子动力学瞬时转染细胞。此外,它允许达到较高的数据在有限的时间。最后,尽管标签的“高”密度FCS提供单个分子的信息。感谢所有这些特性,FCS是一个非常简单的方法,并已被广泛应用于研究脂类和蛋白质动力学无论是在模型膜和活细胞8-10。许多不同的方法已经被提出以提高FCS的揭示分子扩散的细节的能力。例如,它是sh的自己通过对不同大小的观察区域进行的FCS可以定义分子运动11,12的“FCS扩散法”的启发隐藏的功能。除了 被大小不一,重点领域也重复13,移动沿线14-20或共轭快速相机21,22空间。使用这些“时空”的相关办法,几个膜元件相关的生物学参数上都细胞膜的模型与实际的生物学者,由此制得的洞察膜空间组织进行了定量描述。
然而,在所有的FRAP和FCS应用到目前为止描述的焦点区域的大小代表了在空间分辨率的限制,无法克服。几个超分辨率成像方法近来已经开发绕过此限制。有些是基于定位的精度,如随机光学重建显微镜(STORM)<SUP> 23,24,光活化定位显微镜(PALM)25,荧光PALM(FPALM)26,和单粒子跟踪PALM(sptPALM)27:比较大的值,在每个快照需要的光子,但是,限制的时间分辨率这些方法中的至少几毫秒,从而妨碍了它们在体内的适用性。
与此相反,一个有前途的替代超分辨率成像已被打开的空间调制与受激发射损耗的方法(STED或可逆饱和光荧光跃迁(RESOLFT))28,29的荧光发射。这些方法结合起来观察体积的成形以及低于衍射极限与使用快速扫描显微镜和检测系统的可能性。在用荧光波动分析组合,STED显微镜允许直接探测脂质和p的纳米尺度的时空动力学roteins活细胞膜30,31。
相同的物理量的受激发射损耗系显微镜可以通过改性时空图像相关光谱(STICS 32,33)方法,该方法适合于荧光标记的膜蛋白和/或脂 类中的活细胞的动力学的研究得到而由商业显微镜。实验方案这里介绍的是由几个步骤组成。第一个要求在膜感兴趣区域的快速成像。然后,将得到的图像的堆栈用来计算平均值的空间 – 时间相关性的函数。通过拟合一系列的相关函数,该分子“扩散法”,可直接从成像获得的表观扩散系数(D 应用程序 )的形式- 比 -平均位移曲线图。该图的关键取决于由分子探索的环境,并允许直接识别的实际扩散模式的感兴趣的脂质/蛋白质。
更详细地,如先前所示图34中,获取的图像序列的时空自相关函数的关键取决于移动中所收集的图像序列的分子动力学(请注意,相同的推理也可以从行采集应用凡在空间只是一个维度考虑)。特别地,我们定义的相关函数为:
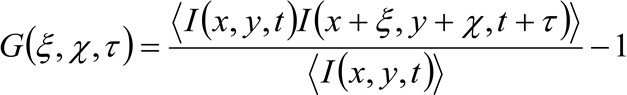 (1)
(1)
其中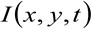 表示测得的荧光强度中的位置x,y和时间t,
表示测得的荧光强度中的位置x,y和时间t, ![]() 和
和![]() 表示在x的距离和分别为Y方向,
表示在x的距离和分别为Y方向, ![]() 表示的时间差,并
表示的时间差,并![]() 表示平均。这个函数可以表示为:
表示平均。这个函数可以表示为:
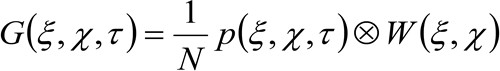 (2)
(2)
其中“N”表示在该观察区域分子的平均数目, ![]() 表示空间中的卷积运算,并
表示空间中的卷积运算,并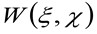 代表器乐腰部的自相关。后者可以被解释为对如何将单个发射器的光子被摊开在空间的量度,由于光/记录设置程序(所谓的点扩散函数,PSF,基因涨势以及由高斯函数近似)。最后,
代表器乐腰部的自相关。后者可以被解释为对如何将单个发射器的光子被摊开在空间的量度,由于光/记录设置程序(所谓的点扩散函数,PSF,基因涨势以及由高斯函数近似)。最后, 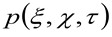 代表的概率找到一个粒子的距离
代表的概率找到一个粒子的距离![]() 和
和![]() 延时后,
延时后, ![]() 。如果我们考虑扩散动力学,粒子随机移动在各个方向和净通量是不存在的,这个功能也很好用高斯函数,其中的差异,可以认定为运动粒子的均方位移(MSD)的近似。因此,相关函数的腰部(也称为
。如果我们考虑扩散动力学,粒子随机移动在各个方向和净通量是不存在的,这个功能也很好用高斯函数,其中的差异,可以认定为运动粒子的均方位移(MSD)的近似。因此,相关函数的腰部(也称为![]() ),可以被定义为颗粒的MSD的总和和器乐腰部,并且可以通过高斯拟合来测定婷的相关函数为每个时间延迟。在测量电流I MSD可以用来计算移动分子的表观扩散系数
),可以被定义为颗粒的MSD的总和和器乐腰部,并且可以通过高斯拟合来测定婷的相关函数为每个时间延迟。在测量电流I MSD可以用来计算移动分子的表观扩散系数![]() 和平均位移
和平均位移![]() 如:
如:
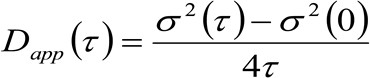 (3)
(3)
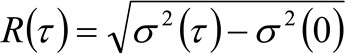 (4)
(4)
很少考虑对实验装置可以用来引导读者在以下几节。为了选择性地激发荧光团上的活细胞,我们将使用全内反射(TIR)的照明,使用市售的TIR荧光的基膜(TIRF)显微镜(细节可在材料部分中找到)。此外,为了收集日ê荧光,我们将使用高倍率物镜(100X NA 1.47,高数值孔径需要全内反射荧光照明)和EMCCD摄像头(像素的物理尺寸上的芯片16微米)。以达到100nm的像素大小,我们应用1.6X的附加放大率透镜。如下面讨论的,将需要低于1毫秒的时间分辨率来适当地描述的快速膜脂质的动力学低于100nm。为了达到这个时间分辨率,我们需要选择的感兴趣区域(ROI)的区域比相机(512×512)的整个芯片更小。在这种方式中,相机将读取线增加时间分辨率的数目可以减少。然而,在此读出机制的帧时间将通过从曝光的电荷转移到读出芯片的照相机上需要与通常以毫秒为512×512像素的EMCCD的顺序的时间限制。要打败这个限制,一个新兴的技术可以转移的投资回报率,仅行而不是整个框架,W第i个实际有效减少暴露的芯片尺寸(称为我们的EMCCD七分传感器模式)。对于这种配置是有效的,必须将ROI的外片覆盖的一对狭缝安装在光路中的。由于这种设置的时间分辨率降低到10 -4秒就可以实现。但是,请注意,这种方法可以与许多不同的实验装置,为“讨论”一节中的说明。
该方法的演示将在活细胞中提供,通过使用这两个标记的一个ATTO488 1 -棕榈酰基-2 -羟基- SN -glycero-3-磷酸乙醇胺(ATTO488-PPE)和转铁蛋白受体的GFP-标记的变体(GFP- TFR)。在ATTO488-PPE的情况下,这种方法可以成功地回收了几乎常数D 的应用程序作为平均位移表示较自由扩散的功能,如先前报道30,35。相比之下,TFR-GFP呈减少ð <suB>应用程序的平均位移的功能,提示局部密闭扩散6。此外,在后者的情况下也能够进行量化的局部扩散常数和平均禁闭面积比在膜面内的许多微米。
Protocol
Representative Results
Discussion
单粒子追踪(SPT)表示的研究分子动力学中最常见的策略之一,它具有测量粒子轨迹的显着优点。这又允许探测在一个复杂的系统,即使少量标记粒子的行为。然而,为了达到这个优点SPT通常需要将探头和非常明亮的标签的密度低。具体地讲,以获得高的时间分辨率(微秒范围)无机探针通常需要( 例如,量子点或金属纳米粒子):在这种情况下的产量,标签和插入一个复杂的过程进系统…
Declarações
The authors have nothing to disclose.
Acknowledgements
This work is supported in part by NIH-P41 P41-RRO3155 and NIH P50-GM076516 (grant to EG), and Fondazione Monte dei Paschi di Siena (grant to FB).
Materials
| Name of Material/ Equipment | Company | Catalog Number | Comments/Description |
| iXon Ultra 897 | Andor | DU-897U-CS0 | |
| Solis | Andor | ||
| CHO-K1 | ATCC | CCL-61 | |
| ATTO 488 labeled PPE | ATTO-TEC GmbH | AD 488-151 | |
| DOPE | Avanti Polar Lipids, Inc. | 850725 | |
| DOTAP | Avanti Polar Lipids, Inc. | 890890 | |
| 100X Penicillin-Streptomycin-Glutamine | Gibco | 10378-016 | |
| DMEM/F-12 | Gibco | 21331 | |
| FBS | Gibco | 10082147 | |
| HEPES | Gibco | 15630-106 | |
| PBS | Gibco | 10010-023 | |
| SimFCS 3.0 | Globals Software | the software can be downloaded here: http://www.lfd.uci.edu/globals/ | |
| DMI6000 with TIRF modulus | Leica | ||
| LAS AF | Leica | ||
| Lipofectamine 2000 | Lipofectamine | 11668019 | |
| Matlab | MathWork | ||
| Imagej | NIH | ||
| C-terminal GFP tagged Tranferrin Receptor | OriGene | RG200980 | |
| Agar | Sigma Aldrich | A5306 | |
| Chloroform | Sigma Aldrich | 528730 | |
| Latex beads, fluorescent yellow-green, 30 nm | Sigma Aldrich | L5155 | |
| SONICA Ultrasonic Cleaners | SOLTEC | ETH S3 | |
| Petri Dishes | Willco | GWSt-3522 | |
| Bio-Format importer for Matlab | http://www.openmicroscopy.org/site/support/bio-formats5/users/matlab/ | ||
| ICS-MatLab Tools | https://www.cellmigration.org/resource/imaging/software/ICSMATLAB_28-02-06.zip | ||
| Simulation by Matlab Tutorial | https://www.cellmigration.org/resource/imaging/icsmatlab/ICSTutorial.html | ||
| Simulation by SimFCS Tutorial | https://www.cellmigration.org/resource/imaging/ppt-pdf/RICS%20Simulations.ppt |
Referências
- Engelman, D. M. Membranes are more mosaic than fluid. Nature. 438 (7068), 578-580 (2005).
- Vereb, G., et al. yet structured: The cell membrane three decades after the Singer-Nicolson model. Proc. Natl. Acad. Sci. U. S. A. 100 (14), 8053-8058 (1073).
- Ishihara, A., Hou, Y., Jacobson, K. The Thy-1 antigen exhibits rapid lateral diffusion in the plasma membrane of rodent lymphoid cells and fibroblasts. 84 (5), 1290-1293 (1987).
- Axelrod, D., et al. Lateral motion of fluorescently labeled acetylcholine receptors in membranes of developing muscle fibers. Proc. Natl. Acad. Sci. U. S. A. 73 (12), 4594-4598 (1976).
- Jacobson, K., Derzko, Z., Wu, E. S., Hou, Y., Poste, G. Measurement of the lateral mobility of cell surface components in single, living cells by fluorescence recovery after photobleaching. J. Supramol. Struct. 5 (4), 10-1002 (1976).
- Kusumi, A., et al. Paradigm shift of the plasma membrane concept from the two-dimensional continuum fluid to the partitioned fluid: high-speed single-molecule tracking of membrane molecules. Annu. Rev. Biophys. Biomol. Struct. 34, 351-378 (2005).
- Kusumi, A., Ike, H., Nakada, C., Murase, K., Fujiwara, T. Single-molecule tracking of membrane molecules: plasma membrane compartmentalization and dynamic assembly of raft-philic signaling molecules. Semin. Immunol. 17 (1), 3-21 (2005).
- Schwille, P., Korlach, J., Webb, W. W. Fluorescence correlation spectroscopy with single-molecule sensitivity on cell and model membranes. Cytometry. 36, 176-182 (1999).
- Gielen, E., et al. Diffusion of sphingomyelin and myelin oligodendrocyte glycoprotein in the membrane of OLN-93 oligodendroglial cells studied by fluorescence correlation spectroscopy. C. R. Biol. 328 (12), 1057-1064 (2005).
- Weiss, M., Hashimoto, H., Nilsson, T. Anomalous protein diffusion in living cells as seen by fluorescence correlation spectroscopy. Biophys. J. 84, 4043-4052 (2003).
- Wawrezinieck, L., Rigneault, H., Marguet, D., Lenne, P. F. Fluorescence correlation spectroscopy diffusion laws to probe the submicron cell membrane organization. Biophys. J. 89 (6), 4029-4042 (2005).
- Lenne, P. F., et al. Dynamic molecular confinement in the plasma membrane by microdomains and the cytoskeleton meshwork. EMBO J. 25 (14), 3245-3256 (2006).
- Ries, J., Schwille, P. Studying slow membrane dynamics with continuous wave scanning fluorescence correlation spectroscopy. Biophys. J. 91 (5), 1915-1924 (2006).
- Ruan, Q., Cheng, M. A., Levi, M., Gratton, E., Mantulin, W. W. Spatial-temporal studies of membrane dynamics: scanning fluorescence correlation spectroscopy (SFCS). Biophys. J. 87 (2), 1260-1267 (2004).
- Berland, K. M., So, P. T., Chen, Y., Mantulin, W. W., Gratton, E. Scanning two-photon fluctuation correlation spectroscopy: particle counting measurements for detection of molecular aggregation. Biophys. J. 71, 410-420 (1996).
- Heinemann, F., Betaneli, V., Thomas, F. A., Schwille, P. Quantifying lipid diffusion by fluorescence correlation spectroscopy: a critical treatise. Langmuir. 28 (37), 13395-13404 (2012).
- Cardarelli, F., Lanzano, L., Gratton, E. Capturing directed molecular motion in the nuclear pore complex of live cells. Proc. Natl. Acad. Sci. U. S. A. 109 (25), 9863-9868 (2012).
- Sanchez, S. A., Tricerri, M. A., Gratton, E. Laurdan generalized polarization fluctuations measures membrane packing micro-heterogeneity in vivo. Proc. Natl. Acad. Sci. U. S. A. 109 (19), 7314-7319 (2012).
- Cardarelli, F., Lanzano, L., Gratton, E. Fluorescence correlation spectroscopy of intact nuclear pore complexes. Biophys. J. 101 (4), 27-29 (2012).
- Di Rienzo, C., et al. Unveiling LOX-1 receptor interplay with nanotopography: mechanotransduction and atherosclerosis onset. Sci. Rep. 3, 10-1038 (2013).
- Unruh, J. R., Gratton, E. Analysis of molecular concentration and brightness from fluorescence fluctuation data with an electron multiplied CCD camera. Biophys. J. 95 (11), 5385-5398 (2008).
- Kannan, B., et al. Electron multiplying charge-coupled device camera based fluorescence correlation spectroscopy. Anal. Chem. 78 (10), 3444-3451 (2006).
- Jones, S. A., Shim, S. H., He, J., Fast Zhuang, X. three-dimensional super-resolution imaging of live cells. Nat. Methods. 8 (6), 499-508 (2011).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy. 3 (10), 793-795 (2006).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313 (5793), 1642-1645 (2006).
- Hess, S. T., Girirajan, T. P., Mason, M. D. Ultra-high resolution imaging by fluorescence photoactivation localization microscopy. Biophys. J. 91 (11), 4258-4272 (2006).
- Manley, S., et al. High-density mapping of single-molecule trajectories with photoactivated localization microscopy. Nat. Methods. 5 (2), 155-157 (2008).
- Hell, S. W. Far-field optical nanoscopy. Science. 316 (5828), 1153-1158 (2007).
- Klar, T. A., Hell, S. W. Subdiffraction resolution in far-field fluorescence microscopy. Opt. Lett. 24 (14), 954-956 (1999).
- Eggeling, C., et al. Direct observation of the nanoscale dynamics of membrane lipids in a living cell. Nature. 457 (7233), 1159-1162 (2009).
- Hedde, P. N., et al. Stimulated emission depletion-based raster image correlation spectroscopy reveals biomolecular dynamics in live cells. Nat. Commun. 4, .
- Hebert, B., Costantino, S., Wiseman, P. W. Spatiotemporal image correlation spectroscopy (STICS) theory, verification, and application to protein velocity mapping in living CHO cells. Biophys. J. 88 (5), 3601-3614 (2005).
- Brown, C. M., et al. Probing the integrin-actin linkage using high-resolution protein velocity mapping. J. Cell Sci. 119, 5204-5214 (2006).
- Di Rienzo, C., Gratton, E., Beltram, F., Cardarelli, F. Fast spatiotemporal correlation spectroscopy to determine protein lateral diffusion laws in live cell membranes. Proc. Natl. Acad. Sci. U. S. A. 110 (30), 12307-12312 (2013).
- Mueller, V., et al. STED nanoscopy reveals molecular details of cholesterol- and cytoskeleton-modulated lipid interactions in living cells. Biophys. J. 101 (7), 1651-1660 (2011).
- Kleusch, C., Hersch, N., Hoffmann, B., Merkel, R., Csiszar, A. Fluorescent lipids: functional parts of fusogenic liposomes and tools for cell membrane labeling and visualization. Molecules. 17 (1), 1055-1073 (2012).
- Ries, J., Chiantia, S., Schwille, P. Accurate determination of membrane dynamics with line-scan FCS. Biophys. J. 96 (5), 1999-2008 (2009).
- Kolin, D. L., Wiseman, P. W. Advances in image correlation spectroscopy: measuring number densities, aggregation states, and dynamics of fluorescently labeled macromolecules in cells. Cell Biochem. Biophys. 49 (3), 141-164 (2007).
- Digman, M. A., et al. Measuring fast dynamics in solutions and cells with a laser scanning microscope. Biophys. J. 89 (2), 1317-1327 (2005).
- Ritchie, K., et al. Detection of non-Brownian diffusion in the cell membrane in single molecule tracking. Biophys. J. 88 (3), 2266-2277 (2005).
- Voie, A. H., Burns, D. H., Spelman, F. A. Orthogonal-plane fluorescence optical sectioning: three-dimensional imaging of macroscopic biological specimens. J. Microsc. 170, 229-236 (1993).
- Huisken, J., Swoger, J., Del Bene, ., Wittbrodt, F., J, E. H., Stelzer, Optical sectioning deep inside live embryos by selective plane illumination microscopy. Science. 305 (5686), 1007-1009 (2004).
- Wohland, T., Shi, X., Sankaran, J., Stelzer, E. H. Single plane illumination fluorescence correlation spectroscopy (SPIM-FCS) probes inhomogeneous three-dimensional environments. Opt. Express. 18 (10), 10627-10641 (2010).

