Von Fast Fluoreszenz-Imaging, um molekulare Diffusion Gesetz über Live Cell Membrane in einem kommerziellen Mikroskop
Summary
Spatial distribution and temporal dynamics of plasma membrane proteins and lipids is a hot topic in biology. Here this issue is addressed by a spatio-temporal image fluctuation analysis that provides conceptually the same physical quantities of single particle tracking, but it uses small molecular labels and standard microscopy setups.
Abstract
Es wurde zunehmend klar, daß die räumliche Verteilung und die Bewegung der Membran-Komponenten wie Lipide und Proteine sind Schlüsselfaktoren bei der Regulation vieler zellulärer Funktionen. Aufgrund der schnellen Dynamik und die winzigen Strukturen beteiligt sind, wird eine sehr hohe Raum-Zeit-Auflösung erforderlich ist, um das reale Verhalten von Molekülen zu fangen. Hier präsentieren wir die Versuchsprotokoll für die Untersuchung der Dynamik von fluoreszenzmarkierten Plasma-Membran-Proteine und Lipide in lebenden Zellen mit hoher örtlicher und zeitlicher Auflösung. Bemerkenswerterweise ist dieser Ansatz nicht jedes Molekül zu verfolgen, aber es Population Verhalten unter Verwendung aller Moleküle in einer bestimmten Region der Membran berechnet. Der Ausgangspunkt ist eine schnelle Bebilderung einer bestimmten Region auf der Membran. Danach wird eine komplette räumlich-zeitlichen Autokorrelationsfunktion korreliert erfassten Bilder mit zunehmender Zeitverzögerungen, beispielsweise alle 2, 3, n Wiederholungen berechnet. Es ist möglich zu zeigen, dass die Breiteder Peak der räumlichen Autokorrelationsfunktion nimmt bei zunehmender Zeitverzögerung in Abhängigkeit von der Partikelbewegung aufgrund von Diffusion. Daher, passend der Serie von Autokorrelationsfunktionen ermöglicht, extrahieren die tatsächliche Protein mittlere quadratische Verschiebung von Bildgebung (IMSD), hier in der Form des scheinbaren Diffusions vs mittlere Verschiebung vorgestellt. Dies ergibt eine quantitative Blick auf die durchschnittliche Dynamik einzelner Moleküle mit Nanometer-Genauigkeit. Durch die Verwendung eines GFP-markierten Variante des Transferrinrezeptors (TfR) und einer ATTO488 Bezeichnung 1-Palmitoyl-2-hydroxy-sn-glycero-3-phosphoethanolamin (PPE) ist es möglich, die Raumzeit-Regulierung von Protein-und Lipid-Diffusion beobachten um große Membranregionen der Mikro zu-Milli zweiten Zeitbereich.
Introduction
Ausgehend von der ursprünglichen "fluid mosaic"-Modell von Singer und Nicolson, das Bild der zellulären Plasmamembran kontinuierlich in den letzten Jahrzehnten um die Rolle der Schwellen Zytoskelett-und Lipid-Domänen enthalten 1,2 aktualisiert.
Die ersten Beobachtungen wurden durch Fluoreszenzerholung nach Photobleichen (FRAP) enthüllt, dass ein signifikanter Anteil von Membranproteinen unbeweglich 3-5 erhalten. Diese bahnbrechenden Studien, wenn auch sehr informativ, von der relativ schlechte Auflösung im Raum (Mikrometer) und die Zeit (in Sekunden) von FRAP-Setups gelitten. Auch als ein Ensemble Mittelung Messung, fehlt in FRAP mit Informationen über Einzelmolekülverhalten.
In diesem Zusammenhang ist die Möglichkeit, spezifisch zu markieren ein einzelnes Molekül mit sehr hellen Tags (die die Untersuchung des Diffusionsprozesses ein Molekül zu einem Zeitpunkt) ist sehr erfolgreich gewesen. Besonders, indem Sie dieZeitauflösung der Single Particle Tracking (SPT) Ansatz für die Mikrosekunden-Zeitskala, Kusumi et erhielten Zugang zu unbekannten Funktionen von Lipid-und Proteindynamik, die sich stark auf die Anerkennung der Rolle der Aktin-basierten Membranskelett in Membranphysiologie 6 beigetragen al. , 7. Diese Ergebnisse erzeugt die so genannte die "Lattenzaun und" Modell, in dem Lipid-und Proteindiffusion durch Aktin-Skelett-basierte geregelt. , Um den Zugriff auf die riesige Menge von Informationen, die von SPT viele experimentelle Fragen gestellt haben müssen jedoch angesprochen werden. Insbesondere wird das Markierungsverfahren typischerweise durch viele Schritte wie Herstellung, Reinigung und die Einführung der markierten Spezies in das System besteht. Weiterhin großen Labels, wie Quantenpunkte oder Metall-Nanopartikel, sind oft erforderlich, um die Sub-Millisekunden-Zeitskala und die Vernetzung der Zielmoleküle durch das Label nicht in vielen Fällen vermieden werden, zu erreichen. Schließlich sind viele Bahnenhaben aufzuzeichnenden statistischen Kriterien passen und damit eine niedrige Dichte des Etiketts erforderlich ist, um Tracking zu ermöglichen.
Im Vergleich zu SPT, Fluoreszenz-Korrelations-Spektroskopie (FCS), die Überwindung viele dieser Nachteile stellt einen vielversprechenden Ansatz, um Molekulardynamik zu studieren. Tatsächlich funktioniert FCS gut auch mit dim und dichte Etiketten, so dass die Dynamik des fluoreszierenden Protein-markierte Moleküle in transient transfizierten Zellen zu studieren. Außerdem ermöglicht es erreichte hohe Statistiken in einem begrenzten Zeitraum. Schließlich trotz der "hohen" Dichte von Etiketten FCS bietet einzelne Moleküle Informationen. Dank all dieser Eigenschaften stellt FCS einen sehr einfachen Ansatz und wurde ausgiebig angewendet, um Lipid und Proteindynamik sowohl in Modellmembranen und im Live-Zellen 8-10 studieren. Viele verschiedene Ansätze vorgeschlagen worden, um die Fähigkeit von FCS auf die Details der molekularen Diffusion zeigen erhöhen. Zum Beispiel war es sheigenen, die durch Ausführen FCS auf unterschiedlich großen Beobachtungsgebiete kann man ein "FCS Diffusionsgesetz" aufschlussreich versteckt Merkmale des Molekularbewegung 11,12 definieren. Neben der Verwendung in der Größe variiert wurde die Schwerpunktbereich auch dupliziert 13, bewegt im Raum entlang der Linien 14-20 bzw. konjugiert mit schnellen Kameras 21,22. Mit Hilfe dieser "Raum-Zeit-'Korrelation Ansätze wurden relevante biologische Parameter von mehreren Membrankomponenten quantitativ auf beiden Modellmembranen und der tatsächlichen biologischen diejenigen, wodurch man einen Einblick in die räumliche Organisation Membran beschrieben.
Jedoch in aller FRAP und FCS Anwendungen bisher beschriebenen die Größe der Fokusfläche eine Grenze der räumlichen Auflösung, die nicht überwunden werden können. Mehrere Super-Resolution-bildgebenden Verfahren wurden kürzlich entwickelt, um diese Grenze zu umgehen. Einige sind auf Lokalisierungsgenauigkeit basiert, wie stochastische optische Rekonstruktion Mikroskopie (STORM) <sup> 23,24, Photoaktivierung Lokalisationsmikroskopie (PALM) 25, Fluoreszenz PALM (FPALM) 26 und single-particle tracking PALM (sptPALM) 27: Die relativ große Menge an Photonen bei jeder Momentaufnahme erforderlich ist, begrenzt jedoch die Zeitauflösung Diese Verfahren, um mindestens einige Millisekunden, wodurch ihre Anwendbarkeit in vivo hemmt.
Im Gegensatz dazu haben eine vielversprechende Alternative zur Super-Resolution Imaging von räumlichen Modulation der Fluoreszenzemission mit Stimulated Emission Depletion-Methoden (STED oder reversible optische Sättigungsfluoreszenzübergänge (RESOLFT)) 28,29 geöffnet. Diese Ansätze kombinieren die Gestaltung der Beobachtungsvolumen deutlich unter der Beugungsgrenze mit der Möglichkeit, schnelle Scan-Mikroskope und Detektionssysteme zu verwenden. In Kombination mit Fluoreszenzfluktuationsanalyse, erlaubt der STED-Mikroskopie, um direkt Sonde die raumzeitliche Dynamik von nanoskaligen Lipide und proteins live Zellmembranen 30,31.
Die gleichen physikalischen Größen STED-Mikroskopie anhand kann durch eine modifizierte raumzeitlichen Bildkorrelationsspektroskopie (Stics 32,33) Verfahren, die für die Untersuchung der Dynamik von fluoreszenzmarkierten Membranproteine und / oder Lipiden in lebenden Zellen erhalten werden und von einem kommerziellen Mikroskop. Das experimentelle Protokoll hier präsentiert wird nur von wenigen Schritten zusammen. Die erste erfordert eine schnelle Abbildung des Bereichs von Interesse auf der Membran. Dann wird der resultierende Stapel von Bildern verwendet, um die durchschnittlichen räumlichen-zeitlichen Korrelationsfunktionen zu berechnen. Vs -Mittlere Verschiebung Grundstück – von der Montage der Serie von Korrelationsfunktionen, die molekulare 'Diffusionsgesetz "kann direkt von der Bildgebung in Form einer scheinbaren Diffusionskoeffizienten (D app) erhalten werden. Dieses Grundstück kritisch hängt von der Umgebung durch die Moleküle erforscht und ermöglicht direkt die Anerkennung der tatsächlichen Diffusionsartender Lipid / Protein von Interesse.
In mehr Details, wie zuvor gezeigt, 34, die räumlich-zeitliche Autokorrelationsfunktion des erworbenen Bild-Serie hängt entscheidend von der Dynamik der Moleküle sich in den gesammelten Bild-Serie (bitte beachten Sie, dass die gleiche Argumentation kann in einer Linie Erwerb angewendet werden wo nur eine Dimension im Raum wird berücksichtigt). Insbesondere definieren wir die Korrelationsfunktion als:
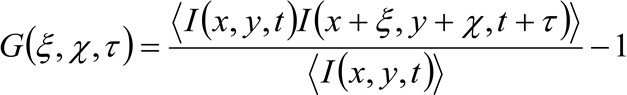 (1)
(1)
wo 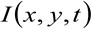 die gemessene Fluoreszenzintensität an der Position x, y, und zum Zeitpunkt t,
die gemessene Fluoreszenzintensität an der Position x, y, und zum Zeitpunkt t, ![]() und
und ![]() stellt den Abstand in der x-undy-Richtung jeweils
stellt den Abstand in der x-undy-Richtung jeweils ![]() repräsentiert die Zeitverzögerung, und
repräsentiert die Zeitverzögerung, und ![]() stellt den Mittelwert. Diese Funktion kann wie folgt ausgedrückt werden:
stellt den Mittelwert. Diese Funktion kann wie folgt ausgedrückt werden:
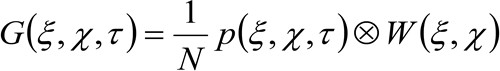 (2)
(2)
wobei 'n' die durchschnittliche Anzahl der Moleküle in dem Überwachungsbereich, ![]() stellt die Faltungsoperation im Raum, und
stellt die Faltungsoperation im Raum, und 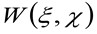 stellt die Autokorrelation der Instrumental Taille. Diese letztere kann als Maß dafür, wie die Photonen eines einzelnen Emitter sind verstreut im Raum aufgrund der optischen / Recording Setup interpretiert werden (die so genannte Point Spread Function, PSF, Gen-Kundgebung gut durch eine Gauß-Funktion angenähert). Schließlich
stellt die Autokorrelation der Instrumental Taille. Diese letztere kann als Maß dafür, wie die Photonen eines einzelnen Emitter sind verstreut im Raum aufgrund der optischen / Recording Setup interpretiert werden (die so genannte Point Spread Function, PSF, Gen-Kundgebung gut durch eine Gauß-Funktion angenähert). Schließlich 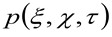 die Wahrscheinlichkeit, ein Teilchen in einem Abstand zu finden
die Wahrscheinlichkeit, ein Teilchen in einem Abstand zu finden ![]() und
und ![]() nach einer Zeitverzögerung
nach einer Zeitverzögerung ![]() . Wenn wir ein Diffusionsdynamik, in denen Teilchen bewegen sich zufällig in alle Richtungen und Nettoflüsse vorhanden sind nicht der Ansicht, diese Funktion ist auch gut mit einer Gauß-Funktion, bei der die Varianz kann als der mittlere quadratische Verschiebung (MSD) des beweglichen Teilchen identifiziert werden angenähert . Somit ist die Taille der Korrelationsfunktion (auch bezeichnet als
. Wenn wir ein Diffusionsdynamik, in denen Teilchen bewegen sich zufällig in alle Richtungen und Nettoflüsse vorhanden sind nicht der Ansicht, diese Funktion ist auch gut mit einer Gauß-Funktion, bei der die Varianz kann als der mittlere quadratische Verschiebung (MSD) des beweglichen Teilchen identifiziert werden angenähert . Somit ist die Taille der Korrelationsfunktion (auch bezeichnet als ![]() ), Kann als die Summe der Partikel MSDS und Instrumental Taille festgelegt und kann durch eine Gaußsche Form gemessen werdenlung der Korrelationsfunktion für jede Zeitverzögerung. Die gemessene I MSD verwendet, um eine scheinbare Diffusivität der beweglichen Moleküle zu berechnen
), Kann als die Summe der Partikel MSDS und Instrumental Taille festgelegt und kann durch eine Gaußsche Form gemessen werdenlung der Korrelationsfunktion für jede Zeitverzögerung. Die gemessene I MSD verwendet, um eine scheinbare Diffusivität der beweglichen Moleküle zu berechnen ![]() und einen mittleren Verschiebungs
und einen mittleren Verschiebungs ![]() Beispiel:
Beispiel:
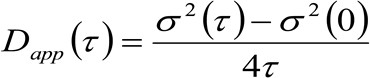 (3)
(3)
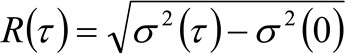 (4)
(4)
Paar Überlegungen über die verwendeten Versuchsaufbau können die Leser in den folgenden Abschnitten zu führen. Um die Fluorophore auf der Basalmembran von lebenden Zellen werden wir eine Totalreflexion (TIR) Beleuchtung zu verwenden, mit einem handelsüblichen TIR Fluoreszenz selektiv anzuregen (TIRF)-Mikroskop (Details finden Sie in der Materialabschnitt zu finden). Darüber hinaus, um zu sammeln the Fluoreszenz wir eine hohe Vergrößerung Ziel verwenden und ein EMCCD Kamera (physikalische Größe der Pixel auf dem Chip 16 um) (100X NA 1,47, wird eine hohe numerische Apertur für TIRF-Beleuchtung erforderlich). Um eine Pixelgröße von 100 nm zu erreichen wenden wir eine zusätzliche Vergrößerungslinse von 1.6X. Wie nachstehend erörtert, würde eine Zeitauflösung von weniger als 1 msec benötigt, um richtig beschreiben die Dynamik schneller Membranlipide unter 100 nm ist. Um diese zeitliche Auflösung zu erreichen müssen wir eine Region of Interest (ROI) wählen kleiner als der ganze Chip der Kamera (512 x 512). Auf diese Weise wird die Kamera eine verringerte Anzahl von Leitungen die Erhöhung der zeitlichen Auflösung zu lesen. Jedoch wird in diesem Auslese Regime die Rahmenzeit würde durch die erforderliche Zeit, um die Ladungen von dem Kontakt mit dem Auslesechip der Kamera zu verschieben und in der Regel in der Größenordnung von Millisekunden für 512 x 512 Pixel EMCCD- begrenzt. Um diese Grenze zu schlagen, ermöglicht eine neue Technologie Verschieben der den gesamten Rahmen ROI-Linien nur statt, wit einem praktischen effektive Reduktion des freigelegten Chipgröße (genannt Gestellte Sensor-Modus in unserem EM-CCD). Für diese Konfiguration wirksam zu sein, muss der Chip außerhalb der ROI von einem Paar von Schlitzen in dem optischen Weg angeordnet abgedeckt werden. Dank dieser Einrichtung eine Zeitauflösung von bis zu 10 -4 Sekunden erreicht werden kann. Bitte beachten Sie jedoch, dass dieser Ansatz mit vielen verschiedenen Versuchsanordnungen gekoppelt werden, wie im Abschnitt "Diskussion", erklärte.
Demonstration des Verfahrens wird in lebenden Zellen bereitgestellt werden, indem sowohl ein ATTO488 Bezeichnung 1-Palmitoyl-2-hydroxy-sn-glycero-3-phosphoethanolamin (ATTO488-PPE) und ein GFP-markierten Variante des Transferrinrezeptors (GFP TFR). Im Fall von ATTO488-PPE kann dieser Ansatz erfolgreich eine nahezu konstante D app als Funktion der durchschnittlichen Verschiebung angibt, dass sie eine freie Diffusion zu erholen, wie zuvor berichtet 30,35. Dagegen TfR-GFP zeigt eine abnehmende D <sub> App als Funktion der durchschnittliche Hubraum, was darauf hindeutet, teilweise beschränkt Diffusions 6. Darüber hinaus wird in dem letzteren Fall ist es möglich, das lokale Diffusionskonstante und der mittleren Einschlussbereich über viele Mikrometer auf der Membranebene zu quantifizieren.
Protocol
Representative Results
Discussion
Einzelpartikelverfolgung (SPT) ist eine der häufigsten Strategien zur Molekulardynamik zu studieren, und es hat den großen Vorteil der Messung Partikelbahnen. Dies wiederum ermöglicht Sondieren das Verhalten noch wenige markierten Partikel in einem komplexen System. Um jedoch diesen Vorteil SPT muss in der Regel eine geringe Dichte der Sonde und sehr hell Etiketten zu erreichen. In diesem Fall ein komplexes Verfahren der Produktion, die Kennzeichnung und das Einsetzen in das System notwendig ist: insbesondere auf hoh…
Declarações
The authors have nothing to disclose.
Acknowledgements
This work is supported in part by NIH-P41 P41-RRO3155 and NIH P50-GM076516 (grant to EG), and Fondazione Monte dei Paschi di Siena (grant to FB).
Materials
| Name of Material/ Equipment | Company | Catalog Number | Comments/Description |
| iXon Ultra 897 | Andor | DU-897U-CS0 | |
| Solis | Andor | ||
| CHO-K1 | ATCC | CCL-61 | |
| ATTO 488 labeled PPE | ATTO-TEC GmbH | AD 488-151 | |
| DOPE | Avanti Polar Lipids, Inc. | 850725 | |
| DOTAP | Avanti Polar Lipids, Inc. | 890890 | |
| 100X Penicillin-Streptomycin-Glutamine | Gibco | 10378-016 | |
| DMEM/F-12 | Gibco | 21331 | |
| FBS | Gibco | 10082147 | |
| HEPES | Gibco | 15630-106 | |
| PBS | Gibco | 10010-023 | |
| SimFCS 3.0 | Globals Software | the software can be downloaded here: http://www.lfd.uci.edu/globals/ | |
| DMI6000 with TIRF modulus | Leica | ||
| LAS AF | Leica | ||
| Lipofectamine 2000 | Lipofectamine | 11668019 | |
| Matlab | MathWork | ||
| Imagej | NIH | ||
| C-terminal GFP tagged Tranferrin Receptor | OriGene | RG200980 | |
| Agar | Sigma Aldrich | A5306 | |
| Chloroform | Sigma Aldrich | 528730 | |
| Latex beads, fluorescent yellow-green, 30 nm | Sigma Aldrich | L5155 | |
| SONICA Ultrasonic Cleaners | SOLTEC | ETH S3 | |
| Petri Dishes | Willco | GWSt-3522 | |
| Bio-Format importer for Matlab | http://www.openmicroscopy.org/site/support/bio-formats5/users/matlab/ | ||
| ICS-MatLab Tools | https://www.cellmigration.org/resource/imaging/software/ICSMATLAB_28-02-06.zip | ||
| Simulation by Matlab Tutorial | https://www.cellmigration.org/resource/imaging/icsmatlab/ICSTutorial.html | ||
| Simulation by SimFCS Tutorial | https://www.cellmigration.org/resource/imaging/ppt-pdf/RICS%20Simulations.ppt |
Referências
- Engelman, D. M. Membranes are more mosaic than fluid. Nature. 438 (7068), 578-580 (2005).
- Vereb, G., et al. yet structured: The cell membrane three decades after the Singer-Nicolson model. Proc. Natl. Acad. Sci. U. S. A. 100 (14), 8053-8058 (1073).
- Ishihara, A., Hou, Y., Jacobson, K. The Thy-1 antigen exhibits rapid lateral diffusion in the plasma membrane of rodent lymphoid cells and fibroblasts. 84 (5), 1290-1293 (1987).
- Axelrod, D., et al. Lateral motion of fluorescently labeled acetylcholine receptors in membranes of developing muscle fibers. Proc. Natl. Acad. Sci. U. S. A. 73 (12), 4594-4598 (1976).
- Jacobson, K., Derzko, Z., Wu, E. S., Hou, Y., Poste, G. Measurement of the lateral mobility of cell surface components in single, living cells by fluorescence recovery after photobleaching. J. Supramol. Struct. 5 (4), 10-1002 (1976).
- Kusumi, A., et al. Paradigm shift of the plasma membrane concept from the two-dimensional continuum fluid to the partitioned fluid: high-speed single-molecule tracking of membrane molecules. Annu. Rev. Biophys. Biomol. Struct. 34, 351-378 (2005).
- Kusumi, A., Ike, H., Nakada, C., Murase, K., Fujiwara, T. Single-molecule tracking of membrane molecules: plasma membrane compartmentalization and dynamic assembly of raft-philic signaling molecules. Semin. Immunol. 17 (1), 3-21 (2005).
- Schwille, P., Korlach, J., Webb, W. W. Fluorescence correlation spectroscopy with single-molecule sensitivity on cell and model membranes. Cytometry. 36, 176-182 (1999).
- Gielen, E., et al. Diffusion of sphingomyelin and myelin oligodendrocyte glycoprotein in the membrane of OLN-93 oligodendroglial cells studied by fluorescence correlation spectroscopy. C. R. Biol. 328 (12), 1057-1064 (2005).
- Weiss, M., Hashimoto, H., Nilsson, T. Anomalous protein diffusion in living cells as seen by fluorescence correlation spectroscopy. Biophys. J. 84, 4043-4052 (2003).
- Wawrezinieck, L., Rigneault, H., Marguet, D., Lenne, P. F. Fluorescence correlation spectroscopy diffusion laws to probe the submicron cell membrane organization. Biophys. J. 89 (6), 4029-4042 (2005).
- Lenne, P. F., et al. Dynamic molecular confinement in the plasma membrane by microdomains and the cytoskeleton meshwork. EMBO J. 25 (14), 3245-3256 (2006).
- Ries, J., Schwille, P. Studying slow membrane dynamics with continuous wave scanning fluorescence correlation spectroscopy. Biophys. J. 91 (5), 1915-1924 (2006).
- Ruan, Q., Cheng, M. A., Levi, M., Gratton, E., Mantulin, W. W. Spatial-temporal studies of membrane dynamics: scanning fluorescence correlation spectroscopy (SFCS). Biophys. J. 87 (2), 1260-1267 (2004).
- Berland, K. M., So, P. T., Chen, Y., Mantulin, W. W., Gratton, E. Scanning two-photon fluctuation correlation spectroscopy: particle counting measurements for detection of molecular aggregation. Biophys. J. 71, 410-420 (1996).
- Heinemann, F., Betaneli, V., Thomas, F. A., Schwille, P. Quantifying lipid diffusion by fluorescence correlation spectroscopy: a critical treatise. Langmuir. 28 (37), 13395-13404 (2012).
- Cardarelli, F., Lanzano, L., Gratton, E. Capturing directed molecular motion in the nuclear pore complex of live cells. Proc. Natl. Acad. Sci. U. S. A. 109 (25), 9863-9868 (2012).
- Sanchez, S. A., Tricerri, M. A., Gratton, E. Laurdan generalized polarization fluctuations measures membrane packing micro-heterogeneity in vivo. Proc. Natl. Acad. Sci. U. S. A. 109 (19), 7314-7319 (2012).
- Cardarelli, F., Lanzano, L., Gratton, E. Fluorescence correlation spectroscopy of intact nuclear pore complexes. Biophys. J. 101 (4), 27-29 (2012).
- Di Rienzo, C., et al. Unveiling LOX-1 receptor interplay with nanotopography: mechanotransduction and atherosclerosis onset. Sci. Rep. 3, 10-1038 (2013).
- Unruh, J. R., Gratton, E. Analysis of molecular concentration and brightness from fluorescence fluctuation data with an electron multiplied CCD camera. Biophys. J. 95 (11), 5385-5398 (2008).
- Kannan, B., et al. Electron multiplying charge-coupled device camera based fluorescence correlation spectroscopy. Anal. Chem. 78 (10), 3444-3451 (2006).
- Jones, S. A., Shim, S. H., He, J., Fast Zhuang, X. three-dimensional super-resolution imaging of live cells. Nat. Methods. 8 (6), 499-508 (2011).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy. 3 (10), 793-795 (2006).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313 (5793), 1642-1645 (2006).
- Hess, S. T., Girirajan, T. P., Mason, M. D. Ultra-high resolution imaging by fluorescence photoactivation localization microscopy. Biophys. J. 91 (11), 4258-4272 (2006).
- Manley, S., et al. High-density mapping of single-molecule trajectories with photoactivated localization microscopy. Nat. Methods. 5 (2), 155-157 (2008).
- Hell, S. W. Far-field optical nanoscopy. Science. 316 (5828), 1153-1158 (2007).
- Klar, T. A., Hell, S. W. Subdiffraction resolution in far-field fluorescence microscopy. Opt. Lett. 24 (14), 954-956 (1999).
- Eggeling, C., et al. Direct observation of the nanoscale dynamics of membrane lipids in a living cell. Nature. 457 (7233), 1159-1162 (2009).
- Hedde, P. N., et al. Stimulated emission depletion-based raster image correlation spectroscopy reveals biomolecular dynamics in live cells. Nat. Commun. 4, .
- Hebert, B., Costantino, S., Wiseman, P. W. Spatiotemporal image correlation spectroscopy (STICS) theory, verification, and application to protein velocity mapping in living CHO cells. Biophys. J. 88 (5), 3601-3614 (2005).
- Brown, C. M., et al. Probing the integrin-actin linkage using high-resolution protein velocity mapping. J. Cell Sci. 119, 5204-5214 (2006).
- Di Rienzo, C., Gratton, E., Beltram, F., Cardarelli, F. Fast spatiotemporal correlation spectroscopy to determine protein lateral diffusion laws in live cell membranes. Proc. Natl. Acad. Sci. U. S. A. 110 (30), 12307-12312 (2013).
- Mueller, V., et al. STED nanoscopy reveals molecular details of cholesterol- and cytoskeleton-modulated lipid interactions in living cells. Biophys. J. 101 (7), 1651-1660 (2011).
- Kleusch, C., Hersch, N., Hoffmann, B., Merkel, R., Csiszar, A. Fluorescent lipids: functional parts of fusogenic liposomes and tools for cell membrane labeling and visualization. Molecules. 17 (1), 1055-1073 (2012).
- Ries, J., Chiantia, S., Schwille, P. Accurate determination of membrane dynamics with line-scan FCS. Biophys. J. 96 (5), 1999-2008 (2009).
- Kolin, D. L., Wiseman, P. W. Advances in image correlation spectroscopy: measuring number densities, aggregation states, and dynamics of fluorescently labeled macromolecules in cells. Cell Biochem. Biophys. 49 (3), 141-164 (2007).
- Digman, M. A., et al. Measuring fast dynamics in solutions and cells with a laser scanning microscope. Biophys. J. 89 (2), 1317-1327 (2005).
- Ritchie, K., et al. Detection of non-Brownian diffusion in the cell membrane in single molecule tracking. Biophys. J. 88 (3), 2266-2277 (2005).
- Voie, A. H., Burns, D. H., Spelman, F. A. Orthogonal-plane fluorescence optical sectioning: three-dimensional imaging of macroscopic biological specimens. J. Microsc. 170, 229-236 (1993).
- Huisken, J., Swoger, J., Del Bene, ., Wittbrodt, F., J, E. H., Stelzer, Optical sectioning deep inside live embryos by selective plane illumination microscopy. Science. 305 (5686), 1007-1009 (2004).
- Wohland, T., Shi, X., Sankaran, J., Stelzer, E. H. Single plane illumination fluorescence correlation spectroscopy (SPIM-FCS) probes inhomogeneous three-dimensional environments. Opt. Express. 18 (10), 10627-10641 (2010).

