Screening for Funksjonell Non-koding genetiske varianter Bruke Elektro Mobility Skift Assay (EMSA) og DNA-affinitet Nedbør analyse (DAPA)
Summary
We present a strategic plan and protocol for identifying non-coding genetic variants affecting transcription factor (TF) DNA binding. A detailed experimental protocol is provided for electrophoretic mobility shift assay (EMSA) and DNA affinity precipitation assay (DAPA) analysis of genotype-dependent TF DNA binding.
Abstract
Population and family-based genetic studies typically result in the identification of genetic variants that are statistically associated with a clinical disease or phenotype. For many diseases and traits, most variants are non-coding, and are thus likely to act by impacting subtle, comparatively hard to predict mechanisms controlling gene expression. Here, we describe a general strategic approach to prioritize non-coding variants, and screen them for their function. This approach involves computational prioritization using functional genomic databases followed by experimental analysis of differential binding of transcription factors (TFs) to risk and non-risk alleles. For both electrophoretic mobility shift assay (EMSA) and DNA affinity precipitation assay (DAPA) analysis of genetic variants, a synthetic DNA oligonucleotide (oligo) is used to identify factors in the nuclear lysate of disease or phenotype-relevant cells. For EMSA, the oligonucleotides with or without bound nuclear factors (often TFs) are analyzed by non-denaturing electrophoresis on a tris-borate-EDTA (TBE) polyacrylamide gel. For DAPA, the oligonucleotides are bound to a magnetic column and the nuclear factors that specifically bind the DNA sequence are eluted and analyzed through mass spectrometry or with a reducing sodium dodecyl sulfate polyacrylamide gel electrophoresis (SDS-PAGE) followed by Western blot analysis. This general approach can be widely used to study the function of non-coding genetic variants associated with any disease, trait, or phenotype.
Introduction
Sekvensering og genotyping baserte studier, inkludert Genome-Wide Association Studies (GWAS), kandidat locus studier, og dyp-sekvense studier har identifisert mange genetiske varianter som er statistisk assosiert med en sykdom, trekk, eller fenotype. I motsetning til tidligere prediksjoner, er de fleste av disse variantene (85-93%) lokalisert i ikke-kodende regioner og endrer ikke aminosyresekvensen til proteiner 1,2. Tolking av funksjonen av disse ikke-kodende varianter og å bestemme de biologiske mekanismer som forbinder dem til den tilhørende sykdom, har egenskap, eller fenotype vist seg vanskelig 3-6. Vi har utviklet en generell strategi for å identifisere de molekylære mekanismene som lenker varianter til en viktig mellom fenotype – genuttrykk. Denne rørledningen er spesielt utviklet for å identifisere modulering av TF bindende av genetiske varianter. Denne strategien kombinerer beregnings tilnærminger og molekylærbiologiske teknikker som mål å forutsibiologiske effekter av kandidat varianter i silico og bekrefte disse spådommene empirisk (figur 1).
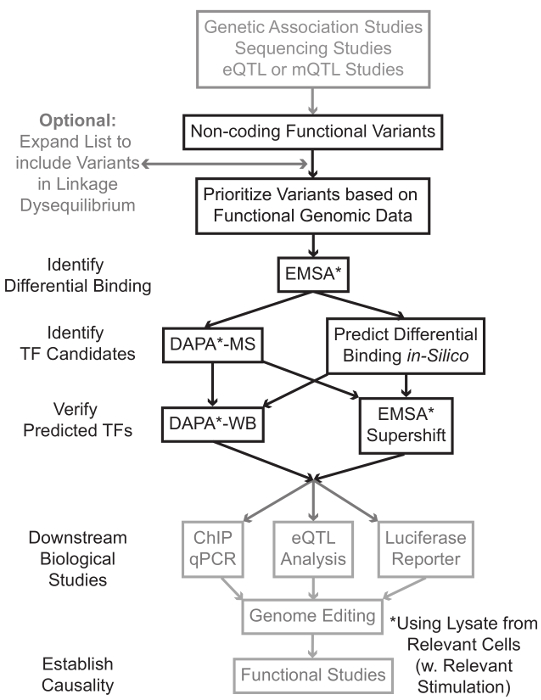
Figur 1:.. Strategisk tilnærming for analyse av ikke-kodende genetiske varianter trinn som ikke er inkludert i detaljert protokoll i forbindelse med dette manuskriptet er skyggelagt i grått Klikk her for å se en større versjon av dette tallet.
I mange tilfeller er det viktig å begynne med å utvide listen over varianter for å inkludere alle de i høy linkage-ulikevekt (LD) med hver statistisk tilhørende variant. LD er et mål på ikke-tilfeldig forening av alleler ved to forskjellige kromosomale stillinger, som kan måles ved R 2 7. R2 er et mål på linKage ulikevekt mellom to varianter, med en r 2 = 1 betegner perfekt kobling mellom to varianter. Alleler i høy LD er funnet å co-skille på kromosomet over forfedrenes populasjoner. Nåværende genotyping arrays omfatter ikke alle kjente varianter i det menneskelige genom. I stedet, de utnytte LD innenfor det menneskelige genom, og inkluderer en undergruppe av de kjente variantene som fungerer som stedfortredere for andre varianter innenfor en bestemt region av LD 8. Således kan en variant uten noe biologisk konsekvens være assosiert med en spesiell sykdom fordi det er i LD med den kausale variant-varianten med en meningsfull biologisk virkning. Prosedyremessig anbefales det å konvertere den nyeste utgaven av 1000 genomer projisere 9 variant samtalefiler (vcf) i binærfiler som er kompatible med plink 10,11, en åpen kildekode-verktøy for hele genomet forening analyse. Deretter alle andre genetiske varianter med LD r 2> 0,8 med hver inngang genetisk vaRiant kan identifiseres som kandidater. Det er viktig å bruke riktig referansegruppen for dette trinn for eksempel hvis en variant ble identifisert i fagene europeisk opphav, data fra personer av samme herkomst bør brukes for LD ekspansjon.
LD ekspansjon resulterer ofte i en rekke kandidat varianter, og det er sannsynlig at bare en liten brøkdel av disse bidrar til sykdom mekanisme. Ofte er det umulig å eksperimentelt undersøke hver av disse variantene individuelt. Det er derfor nyttig å utnytte de tusenvis av offentlig tilgjengelige funksjonell genomikk datasett som et filter for å prioritere varianter. For eksempel har KODE konsortiet 12 utført tusenvis av chip-seq eksperimenter som beskriver binding av TFS og co-faktorer, og histone karakterer i et bredt spekter av sammenhenger, sammen med kromatin tilgjengelighets data fra teknologier som DNase-seq 13, ATAC -seq 14, og FAIRE-seq 15. Databases og webservere som UCSC Genome Browser 16, Roadmap Epigenomics 17, Blueprint epigenome 18, Cistrome 19, og tilordne 20 gir gratis tilgang til data som produseres av disse og andre eksperimentelle teknikker over et bredt spekter av celletyper og vilkår. Når det er for mange varianter å undersøke eksperimentelt, kan disse dataene brukes til å prioritere de som ligger innenfor sannsynlig regulatoriske regioner i relevante celle- og vevstyper. Videre, i tilfeller der en variant er innenfor en chip-seq topp for et bestemt protein, kan disse dataene gir potensielle kunder med hensyn til spesifikke TF (e) eller co-faktorer som bindende kan påvirke.
Deretter blir de resulterende prioritert variantene vist eksperimentelt å validere spådd genotype-avhengige proteinbinding ved hjelp av EMSA 21,22. EMSA måler endringen i migreringen av oligo på en ikke-reduserende TBE gel. Fluorescensmerkede oligo inkuberes med denatom lysat, og binding av nukleære faktorer vil retardere bevegelsen av oligo på gelen. På denne måte oligo som har bundet flere nukleære faktorer vil presentere som en sterkere fluorescerende signal på skanning. Spesielt, har EMSA ikke krever spådommer om spesifikke proteiner som binding vil bli berørt.
Når varianter er identifisert som befinner seg innenfor forutsagte regulatoriske områder og er i stand til differensielt bindende nukleære faktorer, blir beregningsmetoder anvendt for å forutsi den aktuelle TF (e) hvis binding de kan påvirke. Vi foretrekker å bruke cis-BP 23,24, RegulomeDB 25, UniProbe 26, og Jaspar 27. Når kandidaten TFS er identifisert, kan disse spådommene være spesielt testet ved hjelp av antistoffer mot disse TFS (EMSA-supershifts og DAPA-Westerns). En EMSA-supershift involverer tilsetning av et TF-spesifikt antistoff til det nukleære lysatet og oligo. Et positivt resultat i en EMSA-supershift er represented som en ytterligere forskyvning i EMSA band, eller et tap av bandet (anmeldt i referanse 28). I den komplementære DAPA, er en 5'-biotinylert oligo duplex inneholder variant og 20 basepar flankerer nukleotider inkubert med atom lysat fra relevant celletype (r) for å fange opp eventuelle atom faktorer spesifikt binde oligos. Oligo duplex-nukleær faktor komplekset er immobilisert streptavidin av mikroperler i en magnetisk kolonne. De bundet kjernefysiske faktorer samles direkte gjennom eluering 29,48 -kan. Bindings forutsigelser kan deretter bli vurdert av en Western blot ved anvendelse av antistoffer som er spesifikke for proteinet. I tilfeller der det er ingen åpenbare forutsigelser, eller for mange spådommer, de elueringer fra variant pull-downs av DAPA eksperimenter kan sendes til en proteomikk kjerne for å identifisere kandidat TFS hjelp massespektrometri, som senere kan bli validert ved hjelp av disse tidligere beskrevet metoder.
I det gjenværende av article er detaljert protokoll for EMSA og DAPA analyse av genetiske varianter forutsatt.
Protocol
Representative Results
Discussion
Selv om fremskritt i sekvensering og genotyping teknologi har kraftig forbedret vår evne til å identifisere genetiske varianter assosiert med sykdom, er vår evne til å forstå de funksjonelle mekanismer som påvirkes av disse variantene lagging. En hovedkilde til problemet er at mange sykdomsassosierte variantene er lokalisert i n on-kodende regioner av genomet, som sannsynligvis påvirke vanskeligere å forutsi mekanismene som kontrollerer genekspresjon. Her presenterer vi en protokoll basert på EMSA og DAPA…
Declarações
The authors have nothing to disclose.
Acknowledgements
We thank Erin Zoller, Jessica Bene, and Lindsey Hays for input and direction in protocol development. MTW was supported in part by NIH R21 HG008186 and a Trustee Award grant from the Cincinnati Children’s Hospital Research Foundation. ZHP was supported in part by T32 GM063483-13.
Materials
| Custom DNA Oligonucleotides | Integrated DNA Technologies | http://www.idtdna.com/site/order/oligoentry | |
| Potassium Chloride | Fisher Scientific | BP366-500 | KCl, for CE buffer |
| HEPES (1M) | Fisher Scientific | 15630-080 | For CE and NE buffer |
| EDTA (0.5M), pH 8.0 | Life Technologies | R1021 | For CE, NE, and annealing buffer |
| Sodium Chloride | Fisher Scientific | BP358-1 | NaCl, for NE buffer |
| Tris-HCl (1M), pH 8.0 | Invitrogen | BP1756-100 | For annealing buffer |
| Phosphate Buffered Saline (1X) | Fisher Scientific | MT21040CM | PBS, for cell wash |
| DL-Dithiothreitol solution (1M) | Sigma | 646563 | Reducing agent |
| PMSF | Thermo Scientific | 36978 | Protease Inhibitor |
| Phosphatase Inhibitor Cocktail | Thermo Scientific | 78420 | Prevents dephosphorylation of TFs |
| Nonidet P-40 Substitute | IBI Scientific | IB01140 | NP-40, for nuclear extraction |
| BCA Protein Assay Kit | Thermo Scientific | 23225 | For measuring protein concentration |
| Odyssey EMSA Buffer Kit | Licor | 829-07910 | Contains all necessary EMSA buffers |
| TBE Gels, 6%, 12 Wells | Invitrogen | EC6265BOX | For EMSA |
| TBE Buffer (10X) | Thermo Scientific | B52 | For EMSA |
| FactorFinder Starting Kit | Miltenyi Biotec | 130-092-318 | Contains all necessary DAPA buffers |
| Licor Odyssey CLx | Licor | Recommended scanner for DAPA/EMSA | |
| Antibiotic-Antimycotic | Gibco | 15240-062 | Contains 10,000 units/mL of penicillin, 10,000 µg/mL of streptomycin, and 25 µg/mL of Fungizone® Antimycotic |
| Fetal Bovine Serum | Gibco | 26140-079 | FBS, for culture media |
| RPMI 1640 Medium | Gibco | 22400-071 | Contains L-glutamine and 25mM HEPES |
Referências
- Hindorff, L. A., et al. Potential etiologic and functional implications of genome-wide association loci for human diseases and traits. Proc Natl Acad Sci U S A. 106 (23), 9362-9367 (2009).
- Maurano, M. T., et al. Systematic localization of common disease-associated variation in regulatory DNA. Science. 337 (6099), 1190-1195 (2012).
- Ward, L. D., Kellis, M. Interpreting noncoding genetic variation in complex traits and human disease. Nat Biotechnol. 30 (11), 1095-1106 (2012).
- Paul, D. S., Soranzo, N., Beck, S. Functional interpretation of non-coding sequence variation: concepts and challenges. Bioessays. 36 (2), 191-199 (2014).
- Zhang, F., Lupski, J. R. Non-coding genetic variants in human disease. Hum Mol Genet. , (2015).
- Lee, T. I., Young, R. A. Transcriptional regulation and its misregulation in disease. Cell. 152 (6), 1237-1251 (2013).
- Slatkin, M. Linkage disequilibrium–understanding the evolutionary past and mapping the medical future. Nat Rev Genet. 9 (6), 477-485 (2008).
- Bush, W. S., Moore, J. H. Chapter 11: Genome-wide association studies. PLoS Comput Biol. 8 (12), e1002822 (2012).
- 1000 Genomes Project Consortium. An integrated map of genetic variation from 1,092 human genomes. Nature. 491 (7422), 56-65 (2012).
- Chang, C. C., et al. Second-generation PLINK: rising to the challenge of larger and richer datasets. Gigascience. 4, 7 (2015).
- Purcell, S., et al. PLINK: a tool set for whole-genome association and population-based linkage analyses. Am J Hum Genet. 81 (3), 559-575 (2007).
- ENCODE Project Consortium. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Crawford, G. E., et al. Genome-wide mapping of DNase hypersensitive sites using massively parallel signature sequencing (MPSS). Genome Res. 16 (1), 123-131 (2006).
- Buenrostro, J. D., Giresi, P. G., Zaba, L. C., Chang, H. Y., Greenleaf, W. J. Transposition of native chromatin for fast and sensitive epigenomic profiling of open chromatin, DNA-binding proteins and nucleosome position. Nat Methods. 10 (12), 1213-1218 (2013).
- Giresi, P. G., Kim, J., McDaniell, R. M., Iyer, V. R., Lieb, J. D. FAIRE Formaldehyde-Assisted Isolation of Regulatory Elements) isolates active regulatory elements from human chromatin. Genome Res. 17 (6), 877-885 (2007).
- Kent, W. J., et al. The human genome browser at UCSC. Genome Res. 12 (6), 996-1006 (2002).
- Roadmap Epigenomics Consortium. Integrative analysis of 111 reference human epigenomes. Nature. 518 (7539), 317-330 (2015).
- Martens, J. H., Stunnenberg, H. G. BLUEPRINT: mapping human blood cell epigenomes. Haematologica. 98 (10), 1487-1489 (2013).
- Liu, T., et al. Cistrome: an integrative platform for transcriptional regulation studies. Genome Biol. 12 (8), R83 (2011).
- Griffon, A., et al. Integrative analysis of public ChIP-seq experiments reveals a complex multi-cell regulatory landscape. Nucleic Acids Res. 43 (4), e27 (2015).
- Staudt, L. M., et al. A lymphoid-specific protein binding to the octamer motif of immunoglobulin genes. Nature. 323 (6089), 640-643 (1986).
- Singh, H., Sen, R., Baltimore, D., Sharp, P. A. A nuclear factor that binds to a conserved sequence motif in transcriptional control elements of immunoglobulin genes. Nature. 319 (6049), 154-158 (1986).
- Weirauch, M. T., et al. Determination and inference of eukaryotic transcription factor sequence specificity. Cell. 158 (6), 1431-1443 (2014).
- Ward, L. D., Kellis, M. HaploReg: a resource for exploring chromatin states, conservation, and regulatory motif alterations within sets of genetically linked variants. Nucleic Acids Res. 40 (Database issue), D930-D934 (2012).
- Boyle, A. P., et al. Annotation of functional variation in personal genomes using RegulomeDB. Genome Res. 22 (9), 1790-1797 (2012).
- Hume, M. A., Barrera, L. A., Gisselbrecht, S. S., Bulyk, M. L. UniPROBE, update 2015: new tools and content for the online database of protein-binding microarray data on protein-DNA interactions. Nucleic Acids Res. 43 (Database issue), D117-D122 (2015).
- Mathelier, A., et al. JASPAR 2014: an extensively expanded and updated open-access database of transcription factor binding profiles. Nucleic Acids Res. 42 (Database issue), 142-147 (2014).
- Smith, M. F., Delbary-Gossart, S. Electrophoretic Mobility Shift Assay (EMSA). Methods Mol Med. 50, 249-257 (2001).
- Franza, B. R., Josephs, S. F., Gilman, M. Z., Ryan, W., Clarkson, B. Characterization of cellular proteins recognizing the HIV enhancer using a microscale DNA-affinity precipitation assay. Nature. 330 (6146), 391-395 (1987).
- . BCA Protein Assay Kit: User Guide Available from: https://tools.thermofisher.com/content/sfs/manuals/MAN0011430_Pierce_BCA_Protein_Asy_UG.pdf (2014)
- Wijeratne, A. B., et al. Phosphopeptide separation using radially aligned titania nanotubes on titanium wire. ACS Appl Mater Interfaces. 7 (21), 11155-11164 (2015).
- Silva, J. M., McMahon, M. The Fastest Western in Town: A Contemporary Twist on the Classic Western Blot Analysis. J. Vis. Exp. (84), (2014).
- Lu, X., et al. Lupus Risk Variant Increases pSTAT1 Binding and Decreases ETS1 Expression. Am J Hum Genet. 96 (5), 731-739 (2015).
- Ramana, C. V., Chatterjee-Kishore, M., Nguyen, H., Stark, G. R. Complex roles of Stat1 in regulating gene expression. Oncogene. 19 (21), 2619-2627 (2000).
- Fillebeen, C., Wilkinson, N., Pantopoulos, K. Electrophoretic Mobility Shift Assay (EMSA) for the Study of RNA-Protein Interactions: The IRE/IRP Example. J. Vis. Exp. (94), e52230 (2014).
- Heng, T. S., Painter, M. W. Immunological Genome Project, C. The Immunological Genome Project: networks of gene expression in immune cells. Nat Immunol. 9 (10), 1091-1094 (2008).
- Wu, C., et al. BioGPS: an extensible and customizable portal for querying and organizing gene annotation resources. Genome Biol. 10 (11), R130 (2009).
- Wu, C., Macleod, I., Su, A. I. BioGPS and MyGene.info: organizing online, gene-centric information. Nucleic Acids Res. 41 (Database issue), D561-D565 (2013).
- Wang, J., et al. Sequence features and chromatin structure around the genomic regions bound by 119 human transcription factors. Genome Res. 22 (9), 1798-1812 (2012).
- Holden, N. S., Tacon, C. E. Principles and problems of the electrophoretic mobility shift assay. J Pharmacol Toxicol Methods. 63 (1), 7-14 (2011).
- Xu, J., Liu, H., Park, J. S., Lan, Y., Jiang, R. Osr1 acts downstream of and interacts synergistically with Six2 to maintain nephron progenitor cells during kidney organogenesis. Development. 141 (7), 1442-1452 (2014).
- Yang, T. -. P., et al. Genevar: a database and Java application for the analysis and visualization of SNP-gene associations in eQTL studies. Bioinformatics. 26 (19), 2474-2476 (2010).
- Fort, A., et al. A liver enhancer in the fibrinogen gene cluster. Blood. 117 (1), 276-282 (2011).
- Solberg, N., Krauss, S. Luciferase assay to study the activity of a cloned promoter DNA fragment. Methods Mol Biol. 977, 65-78 (2013).
- Rahman, M., et al. A repressor element in the 5′-untranslated region of human Pax5 exon 1A. Gene. 263 (1-2), 59-66 (2001).
- Mali, P., et al. RNA-Guided Human Genome Engineering via Cas9. Science. 339 (6121), 823-826 (2013).

