양육 및 숨기기 비틀에 RNA 매개 유전자 최저를 두 번 좌초,<em> Dermestes maculatus</em
Summary
여기, 우리는 실험실에서 중간 – 세균 딱정벌레, Dermestes maculatus (D. maculatus)를 양육하기위한 프로토콜을 제시한다. 우리는 또한이 종에서 유전자 기능을 연구하기 위해 배아 표현형을 분석 배아 및 부모의 RNAi를위한 프로토콜과 방법을 공유 할 수 있습니다.
Abstract
Advances in genomics have raised the possibility of probing biodiversity at an unprecedented scale. However, sequence alone will not be informative without tools to study gene function. The development and sharing of detailed protocols for the establishment of new model systems in laboratories, and for tools to carry out functional studies, is thus crucial for leveraging the power of genomics. Coleoptera (beetles) are the largest clade of insects and occupy virtually all types of habitats on the planet. In addition to providing ideal models for fundamental research, studies of beetles can have impacts on pest control as they are often pests of households, agriculture, and food industries. Detailed protocols for rearing and maintenance of D. maculatus laboratory colonies and for carrying out dsRNA-mediated interference in D. maculatus are presented. Both embryonic and parental RNAi procedures-including apparatus set up, preparation, injection, and post-injection recovery-are described. Methods are also presented for analyzing embryonic phenotypes, including viability, patterning defects in hatched larvae, and cuticle preparations for unhatched larvae. These assays, together with in situ hybridization and immunostaining for molecular markers, make D. maculatus an accessible model system for basic and applied research. They further provide useful information for establishing procedures in other emerging insect model systems.
Introduction
1998 년 화재와 멜로는 이중 가닥 RNA (dsRNA를이) 예쁜 꼬마 선충 1 유전자 기능의 억제를 유도 할 수 있다고보고했다. dsRNA에 의해 유발이 반응은 RNA 간섭 (RNAi의) 명명하고, 이러한 RNAi를 매개 된 유전자 사일런 싱은 동물, 식물, 균류 및 2-7에서 보존되는 것으로보고되었다. 일부 식물 및 동물에서의 RNAi 기능 전신적 의미 효과 (8-10에서 검토) dsRNA를 직접 도입되지 않은 다른 세포 / 조직으로 확산 될 수있다. 과학자하여 직접 (11-14에서 검토) 게놈을 조작없이 유전자 기능을 쓰러 뜨린, 관심의 유전자를 대상으로 dsRNA에를 설계하여이 내생 세포의 RNAi 응답의 사용을 만들었습니다.
RNAi의는 다음과 같은 장점으로 인해 기능 연구를위한 강력한 도구입니다. 먼저도 최소한의 유전자 서열 정보하는 유전자의 RNAi를 사용하여 타겟팅 될 수있다. 이 일에 특히 중요하다게놈 또는 transcriptomic 데이터가없는 비 모델 생물의 udies. 둘째, RNAi의 응답이 견고 전신 인 유기체에서 RNAi를 매개하는 유전자 녹다운 거의 모든 발육 단계에서 수행 될 수있다. 이 기능은다면 발현 유전자의 기능을 연구하는데 매우 유용하다. 셋째, 어떤 경우에는, RNAi의 효과 표현형 자손 15,16 관찰되도록 상기 생식선 및 자손에 퍼졌다. 단일 주입 부모에 의해 생성 된 수많은 자손 계란을 직접 조작하지 않고 검사 할 수있는 부모의 RNAi (pRNAi)로 알려진이 현상은,,, 배아 발달에 영향을 미치는 유전자에 특히 유리하다. 이러한 이유로, pRNAi는 선택의 방법입니다. pRNAi 비효율적 인 경우, oogenesis 필요한 유전자, 예를 들어 다음의 RNAi 배아 (eRNAi)이 사용되어야한다. 넷째, RNAi의 강력한 결함 약한 생산하는 dsRNA의 양은 범위에 걸쳐 변화 될 수있는 전달하는 대립 시리즈의 등가를 생성하는데 사용될 수있다. 표현형 그러한 계조 유전자가 치명적인 복잡한 프로세스 및 / 또는 기능을 완전히 상실 포함될 때 유전자 기능을 이해하는데 도움이 될 수있다. 다섯째, dsRNA를 전달 특히 강력한 조직의 RNAi 반응을 나타내는 동물, 일반적으로 쉽게 가능하다. dsRNA를이 미세 주입 1,5, 공급 / 섭취 17, 18, 침지, 19, 20 및 바이러스 / 박테리아 매개 배달 (21, 22)에 의해 도입 될 수있다. 여섯째, 일부 유전자 타겟팅 / 편집 방법과 달리, 돌연변이를 운반 생물을 선별하거나 RNAi의 사용시 동형 접합체를 생성하기 위해 유전 교배를 실시 할 필요가 없다. 따라서, 유전자 기능을 연구하기위한 다양한 다른 기술에 비해 저렴 RNAi를 빠르게하고, 대형 화면 (23-25)에 적용될 수있다.
의 RNAi의 다양한 유틸리티 beyon 연구 가능한 종의 범위를 확대하고, 생물의 광범위한 기능적 연구를 수행하는 방법을 제공한다유전 적 도구가 개발되어있는 기존의 모델 시스템 거라고. 예를 들어, 비 – 모델 시스템을 사용하여 연구가 다른 개발 모드를 표시하거나 별개의 형태 적 특징 26-29을 나타내는 종 orthologs의 기능을 비교하여 유전자 및 유전자 네트워크의 발전에 대한 통찰력을 제공해야한다. 연구 이러한 유형의 응용 및 기본적인 두 연구의 영향이 생물학적 다양성의 더 나은 이해를 제공 할 것이다.
지구상에서 가장 큰 동물 그룹이기 때문에, 곤충 메커니즘을 기본 다양성을 탐험 할 수있는 좋은 기회를 제공합니다. 또한, 곤충은 일반적으로 작고 짧은 수명주기, 높은 생산력을 가지고 있고, 실험실에서 뒤쪽으로 쉽습니다. 지난 20 년에, RNAi의 성공적 파리목 (사실 파리) 5, 나비목 (나비와 나방) (30)를 포함하여 주문에 걸쳐 곤충 적용되어, 딱정벌레 목 (딱정벌레) 16,31, 벌목 (sawf거짓말, 말벌, 개미와 꿀벌) (32), 매미목 (사실 버그), Isoptera (흰개미) (34), Blattodea (바퀴벌레) (35), 메뚜기 (귀뚜라미, 메뚜기, 메뚜기, 그리고 katydids) 36 Phthiraptera (이가) 37. RNAi를 성공적으로 응용 프로그램, 에크 디손 (42) 신호, 초기 배아에서 패턴의 연구 기능 데이터 (전후방 축 (32), 지느러미 – 복부 축 (28), 세그멘테이션 26,38), 성별 결정 39, 40, 키틴 / 표피의 생합성 (41)를 제공하고있다 사회적 행동 (43), 그리고 더. 다른 곤충 종 개발의 RNAi 방법은 (44-46 검토) 방제에 유용 할 가능성이 있다는 점에서 추가적인 이점이 될 수있다. RNAi의 효과는 비 보존 지역을 타겟팅 선택되는만큼, 유전자 특이뿐만 아니라 종 특이 할 것이다. 의 생존을위한 필수적인 유전자를 대상으로 꿀벌과 누에와 같은 유익한 곤충 종에 대한감염을 제어하는 바이러스 또는 기생충이 종 (47, 48)을 보호하기위한 새로운 전략을 제공 할 수있다.
Dermestes maculatus (D. maculatus), 일반 이름 숨기기 딱정벌레, 남극 대륙을 제외하고 전 세계적으로 배포됩니다. holometabolous 곤충으로, D. maculatus 수명주기는 배아, 애벌레, 번데기 및 성인 단계 (그림 1)을 포함한다. 그것은 육체에 공급하기 때문에, D. maculatus는 죽은 동물을 해골로하는 박물관으로 사용되며, 법의학 곤충 학자 죽음 49,50의 시간을 예상하는 데 사용할 수 있습니다. D. maculatus는 시체, 말린 고기, 치즈, 그리고 번데기 / 다른 곤충의 고치를 포함한 동물 제품을 공급함으로써 가정의 손상, 저장 음식, 그리고 실크, 치즈, 육류 산업 51,52됩니다. 이 딱정벌레에서의 RNAi를 적용하면 경제에 미치는 영향을 최소화하기 위해 효율적이고 환경 친화적 인 방법을 제공 할 수있다. 우리 연구소는 새로운 m로 D. maculatus을 사용하고있다ODEL 곤충 세그먼트 (53)를 연구한다. 그것은 단기 및 장기 생식 개발 사이의 전환을 공부하고 그것에게 유용한 종을, 중간 – 생식 개발자로 실험실 양육 의무가있을뿐 아니라, D. maculatus는 기초 연구에 대한 관심이다.
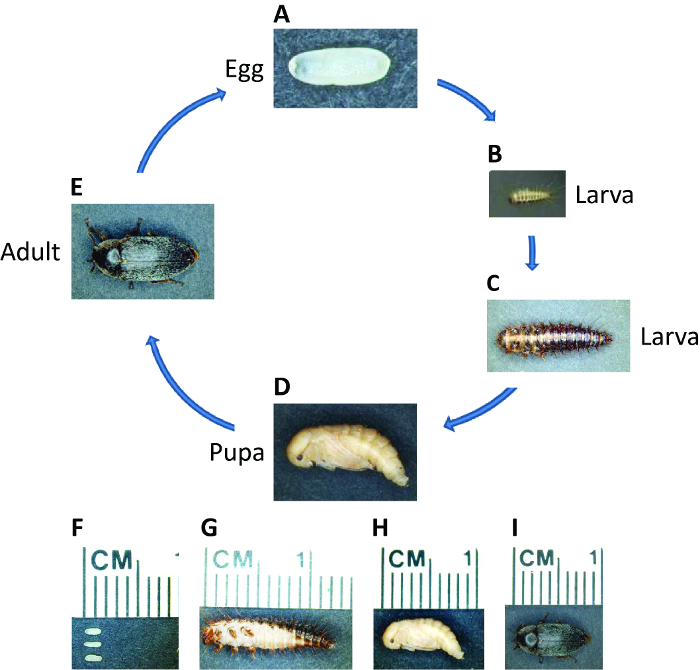
그림 1 : D. maculatus의 수명주기. 나타낸 바와 같이, 서로 다른 삶의 단계에서 D. maculatus의 사진. 성인에 계란에서 라이프 사이클은 낮은 온도에서 세 30 ° C에서 주하지만 더 오래 걸립니다. (A, F) 갓 배치 배아의 길이는 약 1.5 mm, 밝은 노란색 타원형에 흰색입니다. 배아는 ~ 30 ° C에서 55 시간이 걸립니다. (B, C와 G) 애벌레 어두운 색소 줄무늬가 있고 강모로 덮여있다. 애벌레는 1 이상 cm까지 확장 할 수있는 환경과 길이에 따라 여러 령충을 통해 이동합니다. (D, H) </str옹> 젊은 번데기는 황색이다. 30 ° C에서 칠일 – 번데기는 5 ~합니다. 곧 우화 후 (E, I)는, 어두운 색소 침착은 성인 딱정벌레 몸에 나타납니다. 성인은 몇 개월까지 살 수있는 한 여성은 그녀의 일생을 통해 배아의 수백을 배치 할 수 있습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
이전에, 우리는 RNAi의는 D. maculatus (53)에 유전자 기능을 쓰러 뜨린에 효과가 있음을 보여 주었다. 다음은 실험실에서 D. maculatus 식민지를 양육 경험은 배아와 부모 모두 RNAi의 설정, 사출, 포스트 분사 관리 및 표현형 분석을위한 단계별 프로토콜과 함께 공유됩니다. 여기에 소개 된 dsRNA를 매개 유전자 최저 및 분석 방법 D. maculatus에 문제를 해결에 대한 자세한 정보를 제공 할뿐만 아니라, FO 잠재적 인 의미가 아니라R 아닌 다른 모델 딱정벌레 / 곤충 종의 RNAi를 적용.
Protocol
Representative Results
Discussion
정교한 모델 시스템 (마우스, 날아 벌레)의 소수는 20 세기 동안 개발 동안, 21 세기 새로운 동물 시스템의 물결이 전 세계에 걸쳐 연구실에서 개발중인 보았다. 이 새로운 시스템은 과학자 만 '표준'모델 시스템을 사용하여 탐색 할 수 없습니다 비교, 진화의 문제를 해결 할 수 있습니다. 새로운 모델이 배포 실험실 배양, 유전자 식별 및 새로운 종의 기능적 접근 방법의 급속?…
Declarações
The authors have nothing to disclose.
Acknowledgements
We thank Drs. Alison Heffer and Yong Lu for setting up the microinjection apparatus and sharing their invaluable knowledge and experience with insect RNAi. This work was supported by the National Institutes of Health (R01GM113230 to L.P.).
Materials
| Dermestes maculatus live beetles | Our lab or Carolina Biological Supply | #144168 | Our lab strain was verified by COI barcoding; strain variation from Carolina cannot be ruled out |
| Wet cat food | Fancy Feast | Chunks of meat with gravy. Can buy at most pet food and grocery stores | |
| Dry dog food | Purina Puppy Chow | Can buy at most pet food and grocery stores | |
| Insect cage (size medium, 30.5x19x20.3 cm) | Exo Terra | PT2260 | For colony maintenance. Can use larger cage if needed |
| Insect cage (size mini, 17.8×10.2×12.7 cm) | Exo Terra | PT2250 | For embryo collection |
| Petri dish | VWR | 89038-968 | |
| Cotton ball | Fisher | 22-456-883 | |
| Megascript T7 transcription kit | Fisher | AM1334 | For 40 reactions |
| Pneumatic pump | WPI | PV830 | |
| Capillary holder | WPI | ||
| Micromanipulator | NARISHIGE | MN-151 | |
| Black filter paper (90 mm) | VWR | 28342-010 | |
| Food coloring (green) | McCormick | ||
| Borosilicate glass capillary | Hilgenberg | 1406119 | |
| Needle puller (micropipette puller) | Sutter Instrument Co. | P-97 | |
| Microscope glass slide | WorldWide Life Sciences Division | 41351157 | |
| Sealing film (Parafilm M) | Fisher | 13-374-12 | |
| Model 801 Syringe (10 µl ) | Hamilton | 7642-01 | |
| Needle (32-gauge) | Hamilton | 7762-05 | |
| Fixation Solution (Pampel's) | BioQuip Products, Inc. | 1184C | Toxic, needs to be handled in fume hood |
| Forcep (DUMONT #5) | Fine Science Tools | 11252-30 | |
| Cover slip (24X50 mm, No. 1.5) | Globe Scientific | 1415-15 | |
| Eppendorf Femtotips Microloader pipette tip | Fisher | E5242956003 | |
| Dissecting microscopy for embryo injection | Leica | M420 | |
| Dissecting microscopy for larval phenotypic visualization | Zeiss | SteREO Discover. V12 | |
| DIC microscopy | Zeiss | AXIO Imager. M1 | |
Referências
- Fire, A., et al. Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans. Nature. 391, 806-811 (1998).
- Wianny, F., Zernicka-Goetz, M. Specific interference with gene function by double-stranded RNA in early mouse development. Nat Cell Biol. 2, 70-75 (2000).
- Svoboda, P., Stein, P., Hayashi, H., Schultz, R. M. Selective reduction of dormant maternal mRNAs in mouse oocytes by RNA interference. Development. 127, 4147-4156 (2000).
- Zimmermann, T. S., et al. RNAi-mediated gene silencing in non-human primates. Nature. 441, 111-114 (2006).
- Kennerdell, J. R., Carthew, R. W. Use of dsRNA-mediated genetic interference to demonstrate that frizzled and frizzled 2 act in the wingless pathway. Cell. 95, 1017-1026 (1998).
- Cogoni, C., et al. Transgene silencing of the al-1 gene in vegetative cells of Neurospora is mediated by a cytoplasmic effector and does not depend on DNA-DNA interactions or DNA methylation. EMBO J. 15, 3153-3163 (1996).
- Napoli, C., Lemieux, C., Jorgensen, R. Introduction of a Chimeric Chalcone Synthase Gene into Petunia Results in Reversible Co-Suppression of Homologous Genes in trans. Plant Cell. 2, 279-289 (1990).
- van Roessel, P., Brand, A. H. Spreading silence with Sid. Genome Biol. 5, 208 (2004).
- Grishok, A. RNAi mechanisms in Caenorhabditis elegans. FEBS Lett. 579, 5932-5939 (2005).
- Jose, A. M., Hunter, C. P. Transport of sequence-specific RNA interference information between cells. Annu Rev Genet. 41, 305-330 (2007).
- Hannon, G. J. RNA interference. Nature. 418, 244-251 (2002).
- Hammond, S. M., Caudy, A. A., Hannon, G. J. Post-transcriptional gene silencing by double-stranded RNA. Nat Rev Genet. 2, 110-119 (2001).
- Dorsett, Y., Tuschl, T. siRNAs: applications in functional genomics and potential as therapeutics. Nat Rev Drug Discov. 3, 318-329 (2004).
- Agrawal, N., et al. RNA interference: biology, mechanism, and applications. Microbiol Mol Biol Rev. 67, 657-685 (2003).
- Grishok, A., Tabara, H., Mello, C. C. Genetic requirements for inheritance of RNAi in C. elegans. Science. 287, 2494-2497 (2000).
- Bucher, G., Scholten, J., Klingler, M. Parental RNAi in Tribolium (Coleoptera). Curr Biol. 12, 85-86 (2002).
- Timmons, L., Fire, A. Specific interference by ingested dsRNA. Nature. 395, 854 (1998).
- Turner, C. T., et al. RNA interference in the light brown apple moth, Epiphyas postvittana (Walker) induced by double-stranded RNA feeding. Insect Mol Biol. 15, 383-391 (2006).
- Tabara, H., Grishok, A., Mello, C. C. RNAi in C. elegans: soaking in the genome sequence. Science. 282, 430-431 (1998).
- Eaton, B. A., Fetter, R. D., Davis, G. W. Dynactin is necessary for synapse stabilization. Neuron. 34, 729-741 (2002).
- Travanty, E. A., et al. Using RNA interference to develop dengue virus resistance in genetically modified Aedes aegypti. Insect Biochem Mol Biol. 34, 607-613 (2004).
- Whitten, M. M., et al. Symbiont-mediated RNA interference in insects. Proc Biol Sci. 283, (2016).
- Schmitt-Engel, C., et al. The iBeetle large-scale RNAi screen reveals gene functions for insect development and physiology. Nat Commun. 6, 7822 (2015).
- Dönitz, J., et al. iBeetle-Base: a database for RNAi phenotypes in the red flour beetle Tribolium castaneum. Nucleic Acids Res. 43, 720-725 (2015).
- Ulrich, J., et al. Large scale RNAi screen in Tribolium reveals novel target genes for pest control and the proteasome as prime target. BMC Genomics. 16, 674 (2015).
- Choe, C. P., Miller, S. C., Brown, S. J. A pair-rule gene circuit defines segments sequentially in the short-germ insect Tribolium castaneum. Proc. Natl. Acad. Sci. U. S. A. 103, 6560-6564 (2006).
- Angelini, D. R., Kaufman, T. C. Functional analyses in the hemipteran Oncopeltus fasciatus reveal conserved and derived aspects of appendage patterning in insects. Dev Biol. 271, 306-321 (2004).
- Lynch, J. A., Peel, A. D., Drechsler, A., Averof, M., Roth, S. EGF signaling and the origin of axial polarity among the insects. Curr Biol. 20, 1042-1047 (2010).
- Tenlen, J. R., McCaskill, S., Goldstein, B. RNA interference can be used to disrupt gene function in tardigrades. Dev Genes Evol. 223, 171-181 (2013).
- Quan, G. X., Kanda, T., Tamura, T. Induction of the white egg 3 mutant phenotype by injection of the double-stranded RNA of the silkworm white gene. Insect Mol Biol. 11, 217-222 (2002).
- Brown, S. J., Mahaffey, J. P., Lorenzen, M. D., Denell, R. E., Mahaffey, J. W. Using RNAi to investigate orthologous homeotic gene function during development of distantly related insects. Evol Dev. 1, 11-15 (1999).
- Lynch, J. A., Brent, A. E., Leaf, D. S., Pultz, M. A., Desplan, C. Localized maternal orthodenticle patterns anterior and posterior in the long germ wasp Nasonia. Nature. 439, 728-732 (2006).
- Liu, P. Z., Kaufman, T. C. hunchback is required for suppression of abdominal identity, and for proper germband growth and segmentation in the intermediate germband insect Oncopeltus fasciatus. Development. 131, 1515-1527 (2004).
- Zhou, X., Wheeler, M. M., Oi, F. M., Scharf, M. E. RNA interference in the termite Reticulitermes flavipes through ingestion of double-stranded RNA. Insect Biochem Mol Biol. 38, 805-815 (2008).
- Ciudad, L., Piulachs, M. D., Bellés, X. Systemic RNAi of the cockroach vitellogenin receptor results in a phenotype similar to that of the Drosophila yolkless mutant. FEBS J. 273, 325-335 (2006).
- Mito, T., et al. Non-canonical functions of hunchback in segment patterning of the intermediate germ cricket Gryllus bimaculatus. Development. 132, 2069-2079 (2005).
- Yoon, K. S., et al. Brief exposures of human body lice to sublethal amounts of ivermectin over-transcribes detoxification genes involved in tolerance. Insect Mol Biol. 20, 687-699 (2011).
- Rosenberg, M. I., Brent, A. E., Payre, F., Desplan, C. Dual mode of embryonic development is highlighted by expression and function of Nasonia pair-rule genes. Elife. 3, 01440 (2014).
- Hasselmann, M., et al. Evidence for the evolutionary nascence of a novel sex determination pathway in honeybees. Nature. 454, 519-522 (2008).
- Shukla, J. N., Palli, S. R. Sex determination in beetles: production of all male progeny by parental RNAi knockdown of transformer. Sci Rep. 2, 602 (2012).
- Arakane, Y., et al. The Tribolium chitin synthase genes TcCHS1 and TcCHS2 are specialized for synthesis of epidermal cuticle and midgut peritrophic matrix. Insect Mol Biol. 14, 453-463 (2005).
- Cruz, J., Mané-Padròs, D., Bellés, X., Martìn, D. Functions of the ecdysone receptor isoform-A in the hemimetabolous insect Blattella germanica revealed by systemic RNAi in vivo. Dev Biol. 297, 158-171 (2006).
- Guidugli, K. R., et al. Vitellogenin regulates hormonal dynamics in the worker caste of a eusocial insect. FEBS Lett. 579, 4961-4965 (2005).
- Zhang, H., Li, H. C., Miao, X. X. Feasibility, limitation and possible solutions of RNAi-based technology for insect pest control. Insect Sci. 20, 15-30 (2013).
- Huvenne, H., Smagghe, G. Mechanisms of dsRNA uptake in insects and potential of RNAi for pest control: a review. J Insect Physiol. 56, 227-235 (2010).
- Price, D. R., Gatehouse, J. A. RNAi-mediated crop protection against insects. Trends Biotechnol. 26, 393-400 (2008).
- Paldi, N., et al. Effective gene silencing in a microsporidian parasite associated with honeybee (Apis mellifera) colony declines. Appl Environ Microbiol. 76, 5960-5964 (2010).
- Kanginakudru, S., et al. Targeting ie-1 gene by RNAi induces baculoviral resistance in lepidopteran cell lines and in transgenic silkworms. Insect Mol Biol. 16, 635-644 (2007).
- Magni, P. A., Voss, S. C., Testi, R., Borrini, M., Dadour, I. R. A Biological and Procedural Review of Forensically Significant Dermestes Species (Coleoptera: Dermestidae). J Med Entomol. 52, 755-769 (2015).
- Zanetti, N. I., Visciarelli, E. C., Centeno, N. D. The Effect of Temperature and Laboratory Rearing Conditions on the Development of Dermestes maculatus (Coleoptera: Dermestidae). J Forensic Sci. , (2015).
- Veer, V., Negi, B. K., Rao, K. M. Dermestid beetles and some other insect pests associated with stored silkworm cocoons in India, including a world list of dermestid species found attacking this commodity. Journal of Stored Products Research. 32, 69-89 (1996).
- Xiang, J., Forrest, I. S., Pick, L. Dermestes maculatus: an intermediate-germ beetle model system for evo-devo. Evodevo. 6, 32 (2015).
- Fontenot, E. A., Arthur, F. H., Hartzer, K. L. Oviposition of Dermestes maculatus DeGeer, the hide beetle, as affected by biological and environmental conditions. Journal of Stored Products Research. 64, 154-159 (2015).
- Heffer, A., Grubbs, N., Mahaffey, J., Pick, L. The evolving role of the orphan nuclear receptor ftz-f1, a pair-rule segmentation gene. Evol Dev. 15, 406-417 (2013).
- Heffer, A., Shultz, J. W., Pick, L. Surprising flexibility in a conserved Hox transcription factor over 550 million years of evolution. Proc. Natl. Acad. Sci. U. S. A. 107, 18040-18045 (2010).
- Wilson, M. J., Dearden, P. K. Pair-rule gene orthologues have unexpected maternal roles in the honeybee (Apis mellifera). PLoS One. 7, 46490 (2012).
- Dawes, R., Dawson, I., Falciani, F., Tear, G., Akam, M. Dax, a locust Hox gene related to fushi-tarazu but showing no pair-rule expression. Development. 120, 1561-1572 (1994).
- Erezyilmaz, D. F., Kelstrup, H. C., Riddiford, L. M. The nuclear receptor E75A has a novel pair-rule-like function in patterning the milkweed bug, Oncopeltus fasciatus. Dev Biol. 334, 300-310 (2009).
- Stuart, J. J., Brown, S. J., Beeman, R. W., Denell, R. E. A deficiency of the homeotic complex of the beetle Tribolium. Nature. 350, 72-74 (1991).
- Aranda, M., Marques-Souza, H., Bayer, T., Tautz, D. The role of the segmentation gene hairy in Tribolium. Dev Genes Evol. 218, 465-477 (2008).
- Mito, T., et al. even-skipped has gap-like, pair-rule-like, and segmental functions in the cricket Gryllus bimaculatus, a basal, intermediate germ insect (Orthoptera). Dev Biol. 303, 202-213 (2007).
- Patel, N. H., Ball, E. E., Goodman, C. S. Changing role of even-skipped during the evolution of insect pattern formation. Nature. 357, 339-342 (1992).
- Nüsslein-Volhard, C., Wieschaus, E. Mutations affecting segment number and polarity in Drosophila. Nature. 287, 795-801 (1980).
- Jürgens, G., Wieschaus, E., Nüsslein-Volhard, C., Kluding, H. Mutations affecting the pattern of the larval cuticle in Drosophila melanogaster. II. Zygotic loci on the third chromosome. Wilhelm Roux’s archives of developmental biology. 193, 283-295 (1984).
- Wakimoto, B. T., Kaufman, T. C. Analysis of larval segmentation in lethal genotypes associated with the Aantennapedia gene complex in Drosophila melanogaster. Dev. Biol. 81, 51-64 (1981).
- Yu, Y., et al. The nuclear hormone receptor Ftz-F1 is a cofactor for the Drosophila homeodomain protein Ftz. Nature. 385, 552-555 (1997).
- Nüsslein-Volhard, C., Wieschaus, E., Kluding, H. Mutations affecting the pattern of the larval cuticle in Drosophila melanogaster. I.Zygotic loci on the second chromosome. Wilhelm Roux’s archives of developmental biology. 193, 267-282 (1984).
- Wieschaus, E., Nüsslein-Volhard, C., Jürgens, G. Mutations affecting the pattern of the larval cuticle in Drosophila melanogaster. III.Zygotic loci on the X-chromosome and fourth chromosome. ‘Wilhelm Roux’s archives of developmental biology. 193, 296-307 (1984).
- Guichet, A., et al. The nuclear receptor homologue Ftz-F1 and the homeodomain protein Ftz are mutually dependent cofactors. Nature. 385, 548-552 (1997).
- Fontenot, E. A., Arthur, F. H., Hartzer, K. L. Effect of diet and refugia on development of Dermestes maculatus DeGeer reared in a laboratory. J Pest Sci. 88, 113-119 (2014).
- Yang, Y., et al. Biodegradation and Mineralization of Polystyrene by Plastic-Eating Mealworms: Part 1. Chemical and Physical Characterization and Isotopic Tests. Environ Sci Technol. 49, 12080-12086 (2015).
- Kitzmann, P., Schwirz, J., Schmitt-Engel, C., Bucher, G. RNAi phenotypes are influenced by the genetic background of the injected strain. BMC Genomics. 14, 5 (2013).
- Chandler, C. H., Chari, S., Tack, D., Dworkin, I. Causes and consequences of genetic background effects illuminated by integrative genomic analysis. Genética. 196, 1321-1336 (2014).
- Montagutelli, X. Effect of the genetic background on the phenotype of mouse mutations. J Am Soc Nephrol. 11, 101-105 (2000).
- Doetschman, T. Influence of genetic background on genetically engineered mouse phenotypes. Methods Mol Biol. 530, 423-433 (2009).

