Die Erzeugung von Fluorescent Protein Fusions in<em> Candida</em> Arten
Summary
PCR-vermittelte Gen – Modifikation kann verwendet werden , fluoreszierendes Protein – Fusionen in Candida – Spezies zu erzeugen, die Visualisierung und Quantifizierung von Hefezellen und Proteinen erleichtert. Hier präsentieren wir eine Strategie für ein fluoreszierendes Protein – Fusion (ENO1-FP) in Candida parapsilosis zu konstruieren.
Abstract
Candida – Spezies, weit verbreitete Kolonisatoren der Darm- und Urogenitaltrakts, sind die Ursache für die Mehrzahl der invasiven Pilzinfektionen beim Menschen. So werden molekulare und genetische Werkzeuge benötigt, um die Untersuchung ihrer Pathogenese-Mechanismen zu erleichtern. PCR-vermittelte Genveränderung ist eine einfache und schnelle Ansatz Epitop-markierte Proteine zu erzeugen, deren Nachweis zu erleichtern. Insbesondere sind fluoreszierendes Protein (FP) Fusionen leistungsfähige Werkzeuge, die Visualisierung und Quantifizierung von beiden Hefezellen und Proteine, die durch Fluoreszenzmikroskopie und Immunoblotting ermöglichen, respectively. Plasmide enthalten FP – codierenden Sequenzen, zusammen mit Nahrungsmarkergene, die die Transformation von Candida – Arten zu erleichtern, wurden zum Zwecke der FP Konstruktion und Expression in Candida erzeugt wird . Hier präsentieren wir eine Strategie für eine FP – Fusion in einer Candida – Spezies zu konstruieren. Plasmide, die die Nourseothricin resista enthältnce Transformationsmarker – Gen (NAT1) zusammen mit Sequenzen für entweder grün, gelb oder Kirsche FPs (GFP, YFP, mCherry) zusammen mit Primer verwendet , die Gen-spezifische Sequenzen in einer Polymerase – Kettenreaktion (PCR) eine FP – Kassette zu erzeugen , . Diese genspezifischen Kassette hat die Fähigkeit, in das 3'-Ende des entsprechenden Genlocus durch homologe Rekombination zu integrieren. Erfolgreich in-frame-Fusion der FP-Sequenz in das Gen-Locus von Interesse genetisch überprüft, gefolgt von einer Analyse der Fusionsprotein-Expression mittels Mikroskopie und / oder Immundetektionsverfahren. Darüber hinaus ist für den Fall von stark exprimierten Proteine können erfolgreiche Fusionen für primär durch Fluoreszenz-Bildgebungstechniken gescreent werden.
Introduction
Candida – Spezies sind symbiotischer Pilze , die die Darm- und Urogenitaltrakts aller Menschen besiedeln. Unter den Bedingungen der Immunschwäche, wie das mit Frühgeburt auftreten oder immunsuppressive Wirkung von Behandlungen für Krebs, kann Candida – Spezies opportunistischen Erregern werden. Von den Candida – Spezies, ist Candida albicans die häufigste Pilz Kolonisator und bewirkt , dass die Mehrheit der invasiven Pilzinfektionen. Andere Candida – Spezies wie C. glabrata, C. parapsilosis, C. tropicalis und C. kruseii auch schwere Infektionen bei immunsupprimierten Patienten verursachen, mit einigen ausstellenden intrinsische Resistenz gegen häufig Anti-Pilz – Antibiotika wie Fluconazol und Amphotericin B. Daher verwendet, Infektionen mit einigen dieser Arten werden immer häufiger, insbesondere bei Patienten beobachtet prophylaktisch mit Anti-Pilz-Mittel behandelt werden. Auch bei angemessene und rechtzeitige einNTI-Pilz – Behandlung, invasive Candida – Infektionen weiterhin mit einer signifikanten Morbidität und Mortalität 1 verbunden zu werden. Wegen der Bedeutung von Candida – Spezies in die menschliche Gesundheit besteht ein Bedarf an leicht verfügbaren molekularen Werkzeugen, die die Studie und Aufklärung ihrer Pathogenese – Mechanismen ermöglichen.
Ein wichtiges Instrument, das Forscher ermöglicht die Visualisierung und mikrobiellen Zellen und die Proteine zu quantifizieren, die sie zum Ausdruck bringen ist FP-Fusion-Technologie. Polymerase – Kettenreaktion (PCR) -vermittelte Modifikation Gen, wie in diesem Dokument beschrieben ist , ermöglicht die Konstruktion von Fusionen zwischen FP – Sequenzen und ein Candida Protein von Interesse an seiner genomischen Locus codierende Sequenz. Stabile Integration des Konstrukts erleichtert Analyse der Proteinexpression sowie Proteinlokalisierung Dynamik. Plasmide enthalten FP – Sequenzen, die für die Expression in Candida albicans optimiert und das kann in der PCR-vermittelten g verwendet werdenene Modifikationsstrategie wurden zuvor konstruiert 2, 3, 4, 5. Plasmide enthalten FP transformation "Kassetten": eine Sequenz FP auf einen Ernährungsmarker – Gen verknüpft, die die Transformation von C. albicans und C. parapsilosis 2, 3, 4, 5, 6, 7 erleichtert. Derzeit verfügbare Plasmide enthalten eine Vielzahl von selektierbaren Markergenen Nahrungs (URA3, HIS1, ARG4) für die Transformation von auxotrophen Stämmen sowie eine dominant Arzneimittelresistenzmarker (NAT1), die Auxotrophien fehlt Transformation von klinischen Stämmen ermöglicht. Darüber hinaus enthalten die Plasmide Optionen für bis zu vier verschiedenen FP-Sequenzen (grün [GFP], yellow [YFP], cyan [CFP] und Kirsche [mCherry]) und entweder eine ADH1 Terminierungssequenz für den Bau von Carboxy-terminus Protein – Fusionen oder einer Promotorsequenz für den Bau von Amino-Terminus Protein – Fusionen. Primer werden mit Homologie zu der Plasmid-DNA rund um die FP-Kassette entwickelt. Außerdem enthalten die Primer auch 5'-Verlängerungssequenzen eine Homologie zu dem Hefe – Gen von Interesse trägt markiert werden, der Integration der Kassette in den genomischen Locus durch homologe Rekombination (Figur 1) erleichtert. Genspezifischen FP Kassetten werden durch PCR erzeugt und dann umgewandelt in Candida – Zellen kompetent gemacht für die Aufnahme von DNA durch Behandlung mit Lithiumacetat.
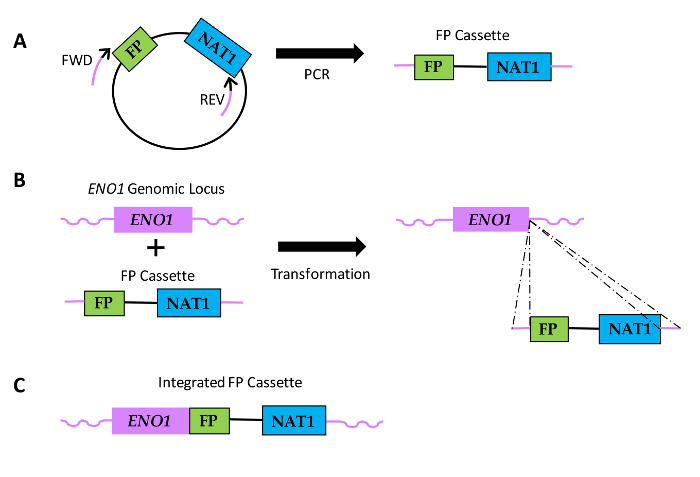
Abbildung 1: Diagramm der wie FP – Sequenz – Fusionen in Candida – Spezies erzeugt werden. (A) Plasmid DNA includes eine FP – Sequenz und eine Sequenz , die Nourseothricin Widerstand (NAT1). Relativen Positionen Vorwärts (FWD) und rückwärts (REV) Primer gezeigt sind, mit schwarzen Abschnitte der Primer die Region der Homologie zu dem Plasmid-Sequenz anzeigt, und die purpurrote Bereiche bezeichnet die genspezifischen Region Homologie oder Primerverlängerung. (B) FP – Kassetten werden in Candida umgewandelt und zu integrieren in der ENO1 genomischen Locus durch homologe Rekombination (gestrichelte Linien). (C) Resultierende FP Fusionssequenz am 3' – Ende von ENO1. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
Hier stellen wir ein Beispiel für Protein – Fusion (ENO1-FP) Konstruktionen in Candida – Spezies. Wir verwenden Tagging Plasmide , die die NAT1 Transformation Markergen zusammen mit Sequenzen , die für GFP, YFP enthält, odermCherry (Abbildung 2). Diese Plasmide werden zusammen mit den Primern in PCR verwendet , um genspezifische Kassetten erzeugen , die Fusion von FPs an das 3'-Ende ENO1 erleichtern, was zu einer Expression von ENO1 fusioniert an FPs an seinem Carboxy-Terminus.
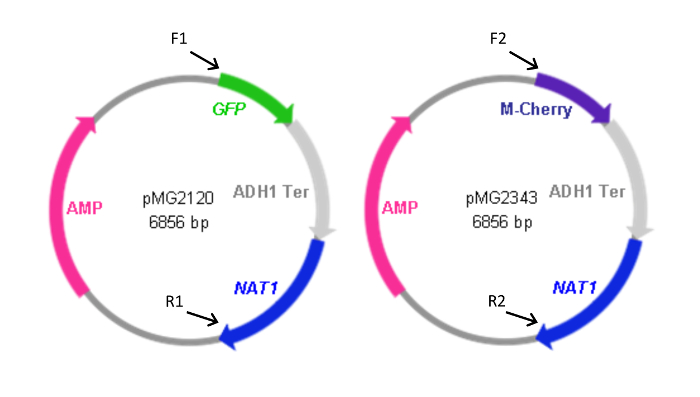
Abbildung 2: Karten von FP – Kassette enthaltenden Plasmide. Vorwärts (F) und rückwärts (R) Primer verwendet, um die Kassetten aus den Plasmiden zu erzeugen, zusammen mit der relativen Lage ihrer Homologie zu den Plasmiden angegeben. Primersequenzen sind in Tabelle 1 aufgeführt. F1 und R1 wurden auch zur Erzeugung der pYFP- NAT1 Kassette verwendet. Das Plasmid , das das YFP- NAT1 Kassette (pMG2263) enthält , ist identisch mit pMG2120 mit Ausnahme von YFP anstelle des GFP – Sequenz. Kassettengrößen: GFP-NAT1, 3,7 kbp; mCherry- NAT1, 3,2 kbp; YFP- NAT1, 3.7 kbp. Diese Zahl wurde von Gerami-Nejad modifiziert, et al. 4 Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
Protocol
Representative Results
Discussion
Konstruktion von Epitop – markierten Sequenzen in Candida Spezies die PCR-vermittelte Gen – Modifikationsstrategie oben beschrieben verwendet , kann als ein dreistufiges Verfahren zusammengefaßt werden. Zunächst wird eine Kassette durch PCR hergestellt, die die Sequenz homolog zur Integration und Regionen gewünschten codiert sowohl auf den Ort des Einfügens in das Hefegenom. Zweitens werden die Hefezellen transformiert werden, mit der Kassette chemisch kompetente mit Lithiumacetat und co-inkubiert hergestel…
Declarações
The authors have nothing to disclose.
Acknowledgements
Wir danken N. Dean für die ursprüngliche mCherry FP-Sequenz bietet, M. Gerami-Nejad für den Bau von Plasmiden, B. Larson für die technische Unterstützung und T. Heisel für hilfreiche Ratschläge bei der Entwicklung dieses Projektes. JB wurde vom European Research Council Erweiterte Auszeichnung 340.087 (RAPLODAPT) unterstützt. Mikroskopie und Imaging-Systeme wurden von der University of Minnesota Pediatrics Foundation und der University of Minnesota Imaging Center zur Verfügung gestellt.
Materials
| 100W mercury lamp | CHIU Technical Corporation | M-100T | |
| 95% Ethanol | Any | NA | |
| Adenine | Any | NA | |
| Ampicillin | Any | NA | |
| Carrier DNA | Ambion | AM9680 | Sheared Salmon Sperm DNA 10 mg/ml |
| CCD Camera | Photometrics | CoolSNAP HQ | |
| Conical Tube | Corning | 430828 | 50ml |
| Culture Tube Rotator | New Brunswick | 2013923 | TC-8, or Any Culture Tube Rotator |
| Deoxynucleotides (dNTP) PCR Grade | Any | NA | |
| Eppendorf Tubes | Eppendorf | 022363719, 022363212 | 0.5ml, 1.5ml |
| Erlenmeyer Flask | Fisher Scientific | 7250089 | 125ml |
| Ethylenediaminetetraacetic Acid (EDTA) | Any | NA | |
| Freezer (-80 °C ) | Thermo Electron Corporation | ULT-1386-9-V | Revco Ultima II |
| GFP, YFP and Texas Red Filter Sets | Chroma Technology Corporation | 49002, 86004v2, 49008 | |
| Glass culture tubes | Fisher Scientific | 1496126 | 75mm |
| HRP goat anti-mouse antibody | Santa Cruz Biotechnology | SC-2005 | |
| HRP goat anti-rabbit antibody | Santa Cruz Biotechnology | SC-2301 | |
| Incubator (30 °C ) | Any | NA | |
| Lithium Acetate | Any | NA | |
| Lysogeny Broth (LB) Media | Any | NA | |
| Magnesium Chloride | Any | NA | |
| Microcentrifuge | Eppendorf | 5415 D | |
| Microscope | Nikon | E600 | Nikon Eclipse E600 |
| Microscope Image Analysis Software | Universal Imaging Corporation | 6.3r7 | MetaMorph Software Series 6.3r7 |
| Mouse anti-GFP antibody | Roche | 11814460001 | |
| Nourseothricin | Fisher Scientific | 50997939 | |
| PCR Thermocycler | Applied Biosystems | 9700 | GeneAmp PCR System |
| PCR tubes | BioExpress, GeneMate | T-3035-1 | 0.2ml |
| Polyethylene Glycol 3350 | Any | NA | |
| Potassium Chloride | Any | NA | |
| Rabbit anti-mCherry antibody | BioVision | 5993-100 | |
| Refrigerator (4°C) | Any | NA | |
| Sodium Acetate | Any | NA | |
| Stereomicroscope | Nikon | SMZ1500 | |
| Table Top Centrifuge | Labnet | Z 400 | Hermle Z 400 |
| Taq DNA Polymerase | Any | NA | |
| Tris(hydroxymethyl)aminomethane (Tris) | Any | NA | |
| Vortex Mixer | Scientific Industries | SI-0236 | Vortex Genie 2 |
| Yeast Extract Peptone Dextrose (YPD) Media | Any | NA |
Referências
- Bendel, C. M. Colonization and epithelial adhesion in the pathogenesis of neonatal candidiasis. Semin. Perinatol. 27 (5), 357-364 (2003).
- Gerami-Nejad, M., Berman, J., Gale, C. A. Cassettes for PCR-mediated construction of green, yellow, and cyan fluorescent protein fusions in Candida albicans. Yeast. 18 (9), 859-864 (2001).
- Gerami-Nejad, M., Dulmage, K., Berman, J. Additional cassettes for epitope and fluorescent fusion proteins in Candida albicans. Yeast. 26 (7), 399-406 (2009).
- Gerami-Nejad, M., Forche, A., McClellan, M., Berman, J. Analysis of protein function in clinical C. albicans isolates. Yeast. 29 (8), 303-309 (2012).
- Gerami-Nejad, M., Hausauer, D., McClellan, M., Berman, J., Gale, C. Cassettes for the PCR-mediated construction of regulatable alleles in Candida albicans. Yeast. 21 (5), 429-436 (2004).
- Gonia, S., Larson, B., Gale, C. A. PCR-mediated gene modification strategy for construction of fluorescent protein fusions in Candida parapsilosis. Yeast. 33 (2), 63-69 (2016).
- Milne, S. W., Cheetham, J., Lloyd, D., Aves, S., Bates, S. Cassettes for PCR- mediated gene tagging in Candida albicans utilizing nourseothricin resistance. Yeast. 28 (12), 833-841 (2011).
- Ausubel, F. M., et al. . Current Protocols in Molecular Biology. , (1995).
- Wilson, R. B., Davis, D., Mitchell, A. P. Rapid hypothesis testing with Candida albicans through gene disruption with short homology regions. J. Bacteriol. 181 (6), 1868-1874 (1999).
- Pulver, R., et al. Rsr1 focuses Cdc42 activity at hyphal tips and promotes maintenance of hyphal development in Candida albicans. Eukaryotic Cell. 12 (4), 482-495 (2013).
- Falgier, C., et al. Candida species differ in their interactions with immature human gastrointestinal epithelial cells. Pediatr. Res. 69 (5), 384-389 (2011).
- Nosek, J., et al. Genetic manipulation of the pathogenic yeast Candida parapsilosis. Curr. Genet. 42 (1), 27-35 (2002).
- Zemanova, J., Nosek, J., Tomaska, L. High-efficiency transformation of the pathogenic yeast Candida parapsilosis. Curr. Genet. 45 (3), 183-186 (2004).
- Benjamin, D. K., et al. Neonatal candidiasis among extremely low birth weight infants: risk factors, mortality rates, and neurodevelopmental outcomes at 18 to 22 months. Pediatrics. 117 (1), 84-92 (2006).
- Kullberg, B. J., Arendrup, M. C. Invasive Candidiasis. N Engl J Med. 373 (15), 1445-1456 (2015).

