Generering av Fluorescent Protein Fusions i<em> Candida</em> Arter
Summary
PCR-mediert gen modifikasjon kan brukes til å generere fluorescerende proteinfusjoner i Candida art, som muliggjør visualisering og kvantifisering av gjærceller og proteiner. Heri presenterer vi en strategi for å konstruere et fluorescerende protein fusjon (Eno1-FP) i Candida parapsilosis.
Abstract
Candida-arter, utbredt kolonisatorer intestinal og urin traktater, er årsaken til de fleste av invasive soppinfeksjoner hos mennesker. Dermed er molekylære og genetiske verktøy for å legge til rette studiet av deres patogeneserelaterte mekanismer. PCR-mediert genmodifisering er en enkel og rask måte å generere epitop-merket proteiner for å lette deres oppdagelse. Spesielt fluorescerende protein (FP)-fusjoner er kraftige verktøy som tillater visualisering og kvantifisering av både gjærceller og proteiner ved fluorescens mikroskopi og immunblotting, respektivt. Plasmider inneholdende FP kodende sekvenser, sammen med nærings-markørgener som letter transformasjon av Candida arter, er blitt generert i den hensikt å FP konstruksjon og ekspresjon i Candida. Heri presenterer vi en strategi for å bygge en FP fusjon i en Candida-arter. Plasmider som inneholder nourseothricin sveisince transformasjon markør-gen (Nat1) sammen med sekvenser for enten grønn, gul, eller kirsebær FPS (GFP, YFP, mCherry) brukes sammen med primere som inneholder gen-spesifikke sekvenser i en polymerase kjedereaksjon (PCR) for å generere en FP kassett . Dette genspesifikke kassett har evnen til å integrere seg i 3'-enden av det tilsvarende gen locus via homolog rekombinasjon. Vellykket i-ramme fusjon av FP sekvens i genet locus av interesse blir verifisert genetisk, etterfulgt av analyse av fusjonsprotein-ekspresjon ved mikroskopi og / eller immuno-deteksjonsmetoder. I tillegg, i tilfellet av høyt uttrykte proteiner, vellykkede fusjoner kan screenes for i første rekke ved fluorescens bildeteknikker.
Introduction
Candida-arter er commensal sopp som kolonisere tarm og urogenitale områder av alle mennesker. Under tilstander med immunsvikt, så som det som forekommer med prematur fødsel eller immunsuppressive effekter av behandlinger for kreft, kan Candida-arter bli opportunistiske patogener. Av de Candida-arter, er Candida albicans den mest utbredte sopp kolonisator og forårsaker de fleste av invasive soppinfeksjoner. Andre Candida-arter som C. glabrata, C. parapsilosis, C. tropicalis og C. kruseii også forårsake alvorlige infeksjoner hos immunkompromitterte pasienter, med noen oppviser iboende motstand mot brukte anti-fungal antibiotika slik som fluconazol og amfotericin B. Derfor infeksjoner med noen av disse artene blir observert oftere, spesielt hos pasienter som behandles profylaktisk med antisoppmidler. Selv med riktig og rettidig enNTI-soppbehandling, invasive Candida-infeksjoner fortsette å være assosiert med signifikant morbiditet og mortalitet 1. På grunn av betydningen av Candida-arter i menneskers helse, er det et behov for lett tilgjengelige molekylære verktøy som tillater studiet og klarlegging av deres patogenese mekanismer.
Et viktig verktøy som lar forskere å visualisere og kvantifisere mikrobielle celler og proteiner som de uttrykker er FP fusion teknologi. Polymerase kjedereaksjon (PCR) -mediert gen modifikasjon, slik det er beskrevet i denne artikkelen, tillater bygging av fusjoner mellom FP sekvenser og en Candida-protein-kodende sekvens av interesse ved dens genomiske locus. Stabil integrering av konstruksjonen forenkler analysen av protein ekspresjon, så vel som proteinlokaliserings dynamikk. Plasmider inneholdende sekvenser som FP, optimalisert for ekspresjon i Candida albicans, og som kan brukes i PCR-mediert gene modifikasjon strategi, er tidligere bygget 2, 3, 4, 5. Plasmider som inneholder FP transformasjon "kassetter": et FP-sekvens bundet til et ernæringsmessig markørgen som muliggjør transformasjon av C. albicans og C. parapsilosis 2, 3, 4, 5, 6, 7. For tiden tilgjengelige plasmider inneholder en rekke valgbare markørgener ernærings (URA3, HIS1, ARG4) for omforming av auxotrofe stammer, så vel som en dominant medikamentresistensmarkør (Nat1), som muliggjør transformasjon av kliniske stammer mangler auxotrophies. I tillegg plasmider inneholder muligheter for opptil fire forskjellige FP sekvenser (grønn [GFP], yellow [YFP], cyan [CFP] og kirsebær [mCherry]) og enten en ADH1 termineringssekvens for bygging av carboxyenden proteinfusjoner, eller en promotorsekvens for bygging av amino-terminale proteinfusjoner. Primerne er utformet med homologi med plasmid-DNA som omgir FP kassetten. I tillegg primerne inneholder også 5'-forlengelse sekvenser som bærer homologi til gjærgenet av interesse å bli merket, noe som letter integrering av kassetten i det genomiske lokuset via homolog rekombinasjon (figur 1). Genspesifikke FP-kassetter er generert ved PCR og deretter transformert inn i Candida-celler som er gjort kompetente for opptak av DNA ved behandling med litium-acetat.
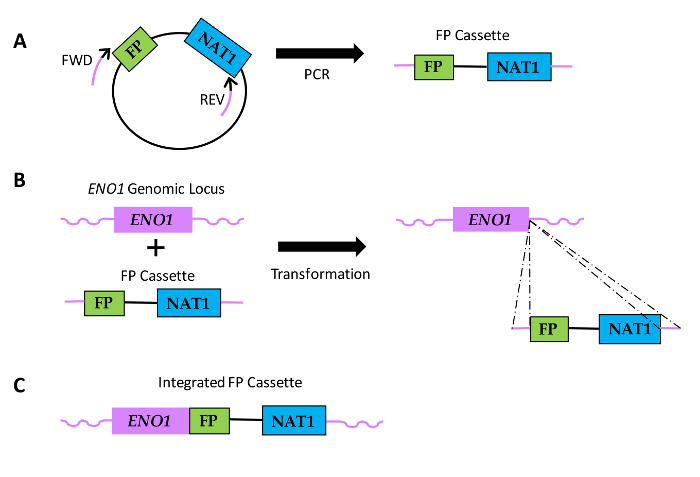
Figur 1: Diagram av hvordan FP sekvens fusjoner blir generert i Candida-arter. (A) Plasmid-DNA inklues en FP sekvens og en sekvens som koder nourseothricin motstand (Nat1). Relative steder av Forward (FWD) og revers (REV) primere er vist med svarte deler av primere som indikerer regionen av homologi til plasmidet sekvens og lilla deler betegner genet spesifikke homologi region eller primer extension. (B) FP kassetter er forvandlet til Candida og integrere i ENO1 genomiske locus via homolog rekombinasjon (stiplede linjer). (C) Resulterer FP fusjon sekvens på 3'end av ENO1. Klikk her for å se en større versjon av dette tallet.
Heri presenterer vi et eksempel på proteinfusjon (Eno1-FP) konstruksjoner i Candida-arter. Vi bruker tagging plasmider som inneholder Nat1 transformasjon markørgenet sammen med sekvenser som koder GFP, YFP, ellermCherry (figur 2). Disse plasmidene brukes sammen med primere i PCR for å generere genspesifikke kassetter som lette fusjon av fps til 3'-enden av ENO1, noe som resulterer i ekspresjon av Eno1 kondensert til fps ved sin karboksy-terminus.
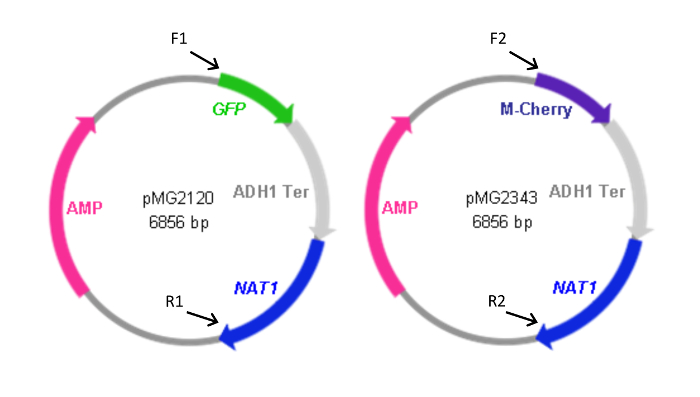
Figur 2: Kart over FP kassett som inneholder plasmider. Forover (F) og omvendt (R) primere som brukes for å generere kassettene fra plasmidene er angitt sammen med den relative plasseringen av deres homologi til plasmidene. Primer sekvenser er som angitt i tabell 1. F1 og R1 ble også brukt for å generere pYFP- Nat1 kassetten. Plasmidet inneholdende YFP- Nat1 kassetten (pMG2263) er identisk med pMG2120 med unntak av YFP i stedet for GFP-sekvensen. Kassett størrelser: GFP-Nat1, 3,7 KBP; mCherry- Nat1, 3,2 kbp; YFP- Nat1, tre.7 kbp. Dette tallet har blitt forandret fra Gerami-Nejad, et al. 4 Klikk her for å se en større versjon av dette tallet.
Protocol
Representative Results
Discussion
Bygging av epitop tagget sekvenser i Candida-arter ved hjelp av PCR-mediert genmodifisering strategi beskrevet ovenfor kan oppsummeres som en tre-trinns prosess. For det første er en kassett laget ved PCR som koder for både den ønskede for integrering og regioner homologe til det geometriske sted for innføring i gjærgenomsekvens. For det andre, blir gjærcellene som skal transformeres gjøres kjemisk kompetent med litiumacetat og ko-inkubert med kassetten. For det tredje, blir cellene sådd ut på selektiv…
Declarações
The authors have nothing to disclose.
Acknowledgements
Vi takker N. Dean for å gi den opprinnelige mCherry FP sekvens, M. Gerami-Nejad for bygging av plasmider, B. Larson for teknisk assistanse, og T. Heisel for nyttige råd under utviklingen av dette prosjektet. JB ble støttet av European Research Council Advanced Award 340087 (RAPLODAPT). Mikroskopi og bildesystemer ble gitt av University of Minnesota Pediatrics Foundation og University of Minnesota Imaging Center.
Materials
| 100W mercury lamp | CHIU Technical Corporation | M-100T | |
| 95% Ethanol | Any | NA | |
| Adenine | Any | NA | |
| Ampicillin | Any | NA | |
| Carrier DNA | Ambion | AM9680 | Sheared Salmon Sperm DNA 10 mg/ml |
| CCD Camera | Photometrics | CoolSNAP HQ | |
| Conical Tube | Corning | 430828 | 50ml |
| Culture Tube Rotator | New Brunswick | 2013923 | TC-8, or Any Culture Tube Rotator |
| Deoxynucleotides (dNTP) PCR Grade | Any | NA | |
| Eppendorf Tubes | Eppendorf | 022363719, 022363212 | 0.5ml, 1.5ml |
| Erlenmeyer Flask | Fisher Scientific | 7250089 | 125ml |
| Ethylenediaminetetraacetic Acid (EDTA) | Any | NA | |
| Freezer (-80 °C ) | Thermo Electron Corporation | ULT-1386-9-V | Revco Ultima II |
| GFP, YFP and Texas Red Filter Sets | Chroma Technology Corporation | 49002, 86004v2, 49008 | |
| Glass culture tubes | Fisher Scientific | 1496126 | 75mm |
| HRP goat anti-mouse antibody | Santa Cruz Biotechnology | SC-2005 | |
| HRP goat anti-rabbit antibody | Santa Cruz Biotechnology | SC-2301 | |
| Incubator (30 °C ) | Any | NA | |
| Lithium Acetate | Any | NA | |
| Lysogeny Broth (LB) Media | Any | NA | |
| Magnesium Chloride | Any | NA | |
| Microcentrifuge | Eppendorf | 5415 D | |
| Microscope | Nikon | E600 | Nikon Eclipse E600 |
| Microscope Image Analysis Software | Universal Imaging Corporation | 6.3r7 | MetaMorph Software Series 6.3r7 |
| Mouse anti-GFP antibody | Roche | 11814460001 | |
| Nourseothricin | Fisher Scientific | 50997939 | |
| PCR Thermocycler | Applied Biosystems | 9700 | GeneAmp PCR System |
| PCR tubes | BioExpress, GeneMate | T-3035-1 | 0.2ml |
| Polyethylene Glycol 3350 | Any | NA | |
| Potassium Chloride | Any | NA | |
| Rabbit anti-mCherry antibody | BioVision | 5993-100 | |
| Refrigerator (4°C) | Any | NA | |
| Sodium Acetate | Any | NA | |
| Stereomicroscope | Nikon | SMZ1500 | |
| Table Top Centrifuge | Labnet | Z 400 | Hermle Z 400 |
| Taq DNA Polymerase | Any | NA | |
| Tris(hydroxymethyl)aminomethane (Tris) | Any | NA | |
| Vortex Mixer | Scientific Industries | SI-0236 | Vortex Genie 2 |
| Yeast Extract Peptone Dextrose (YPD) Media | Any | NA |
Referências
- Bendel, C. M. Colonization and epithelial adhesion in the pathogenesis of neonatal candidiasis. Semin. Perinatol. 27 (5), 357-364 (2003).
- Gerami-Nejad, M., Berman, J., Gale, C. A. Cassettes for PCR-mediated construction of green, yellow, and cyan fluorescent protein fusions in Candida albicans. Yeast. 18 (9), 859-864 (2001).
- Gerami-Nejad, M., Dulmage, K., Berman, J. Additional cassettes for epitope and fluorescent fusion proteins in Candida albicans. Yeast. 26 (7), 399-406 (2009).
- Gerami-Nejad, M., Forche, A., McClellan, M., Berman, J. Analysis of protein function in clinical C. albicans isolates. Yeast. 29 (8), 303-309 (2012).
- Gerami-Nejad, M., Hausauer, D., McClellan, M., Berman, J., Gale, C. Cassettes for the PCR-mediated construction of regulatable alleles in Candida albicans. Yeast. 21 (5), 429-436 (2004).
- Gonia, S., Larson, B., Gale, C. A. PCR-mediated gene modification strategy for construction of fluorescent protein fusions in Candida parapsilosis. Yeast. 33 (2), 63-69 (2016).
- Milne, S. W., Cheetham, J., Lloyd, D., Aves, S., Bates, S. Cassettes for PCR- mediated gene tagging in Candida albicans utilizing nourseothricin resistance. Yeast. 28 (12), 833-841 (2011).
- Ausubel, F. M., et al. . Current Protocols in Molecular Biology. , (1995).
- Wilson, R. B., Davis, D., Mitchell, A. P. Rapid hypothesis testing with Candida albicans through gene disruption with short homology regions. J. Bacteriol. 181 (6), 1868-1874 (1999).
- Pulver, R., et al. Rsr1 focuses Cdc42 activity at hyphal tips and promotes maintenance of hyphal development in Candida albicans. Eukaryotic Cell. 12 (4), 482-495 (2013).
- Falgier, C., et al. Candida species differ in their interactions with immature human gastrointestinal epithelial cells. Pediatr. Res. 69 (5), 384-389 (2011).
- Nosek, J., et al. Genetic manipulation of the pathogenic yeast Candida parapsilosis. Curr. Genet. 42 (1), 27-35 (2002).
- Zemanova, J., Nosek, J., Tomaska, L. High-efficiency transformation of the pathogenic yeast Candida parapsilosis. Curr. Genet. 45 (3), 183-186 (2004).
- Benjamin, D. K., et al. Neonatal candidiasis among extremely low birth weight infants: risk factors, mortality rates, and neurodevelopmental outcomes at 18 to 22 months. Pediatrics. 117 (1), 84-92 (2006).
- Kullberg, B. J., Arendrup, M. C. Invasive Candidiasis. N Engl J Med. 373 (15), 1445-1456 (2015).

