Et grafisk brugergrænseflade til softwareassisteret sporing af proteinkoncentration i dynamiske cellulære protrusioner
Summary
Vi præsenterer en software løsning til semi-automatiseret sporing af relativ protein koncentration langs længden af dynamiske cellulære fremspring.
Abstract
Filopodier er dynamiske, fingerlignende cellulære fremspring forbundet med migration og cellecellekommunikation. For bedre at forstå de komplekse signalmekanismer, der ligger til grund for filopodial initiering, forlængelse og efterfølgende stabilisering eller tilbagetrækning, er det afgørende at bestemme den spatio-temporale proteinaktivitet i disse dynamiske strukturer. For at analysere proteinfunktion i filopodi udviklede vi for nylig en halvautomatiseret sporingsalgoritme, der tilpasser sig filopodiale formændringer, hvilket muliggør parallelanalyse af fremspringsdynamik og relativ proteinkoncentration langs hele filopodiallængden. Her præsenterer vi en detaljeret trin-for-trin protokol for optimeret cellehåndtering, billedoptagelse og softwareanalyse. Vi giver yderligere anvisninger om brugen af valgfrie funktioner under billedanalyse og datarrepræsentation samt retningslinjer for fejlfinding for alle kritiske trin undervejs. Endelig indeholder vi også en sammenligning af dSkræddersyet billedanalysesoftware med andre programmer til filopodi-kvantificering. Sammen giver den præsenterede protokol en ramme for nøjagtig analyse af proteindynamik i filopodiale fremspring ved hjælp af billedanalysesoftware.
Introduction
Spatio-temporal kontrol af actin regulatoriske proteiner er forbundet med filopodiumdynamik 1 , 2 . Sporing af rumligt løst proteinkoncentration langs hele filopodiallængden gennem tiden er således afgørende for at fremme vores forståelse af mekanismerne underliggende initiation, forlængelse, stabilisering eller sammenbrud af disse dynamiske strukturer 3 , 4 . I modsætning til proteinanalyse i cytosol, hvor mange celleformændringer forekommer i større målestok, er filopodier dynamiske mikrostrukturer, der konstant spænder 5 og bøjer og dermed udelukker analyse ved hjælp af en simpel tilgang, såsom en linjeskanning.
Forskellige software løsninger til sporing af filopodial form er tilgængelige 6 , 7 , 8 , 9 . LikewIse, software til ratiometrisk sporing af proteindynamik i cellekroppen er blevet udviklet 10 , 11 . For at kombinere automatiseret sporing af filopodial form og spatio-temporal proteinanalyse har vi for nylig udviklet en billedanalysesoftware baseret på den konvekse skrogalgoritme 12 . Denne nye analysemetode, der drives via en grafisk brugergrænseflade (GUI), kombinerer for første gang den relative proteinkoncentration langs filopodiallængden og væksthastigheden, hvilket tillader den nøjagtige måling af spatio-temporal proteinfordeling uafhængig af bevægelsen af disse Dynamiske strukturer 12 .
Ideen bag softwaren (kildekoden er frit tilgængelig, se nedenfor) er, at en af det konvekse skrogs hjørner vil falde sammen med toppen af filopodiet ( figur 1A ). Ved at se i den efterfølgende ramme foR det nærmeste hjørne af det konvekse skrog, kan den bevægelige spids spores gennem hele filmen. Når spidsen er detekteret i hver ramme, bruges dens position til at tegne en akse ved at forbinde spidsen med et referencepunkt ved bunden af filopodiet ( figur 1B ). Endelig anvendes der ved anvendelse af ækvivalente knudepunkter, hvis positioner bestemmes af median pixel med maksimal intensitet langs linien ortogonale til aksen, til at bestemme en rygrad, som følger filopodial form. Udnyttelse af denne adaptive rygrad genereres en kymografi for at spore filopodial vækst og proteinkoncentrationer for op til tre kanaler langs filopodiallængden ( figur 1C ).
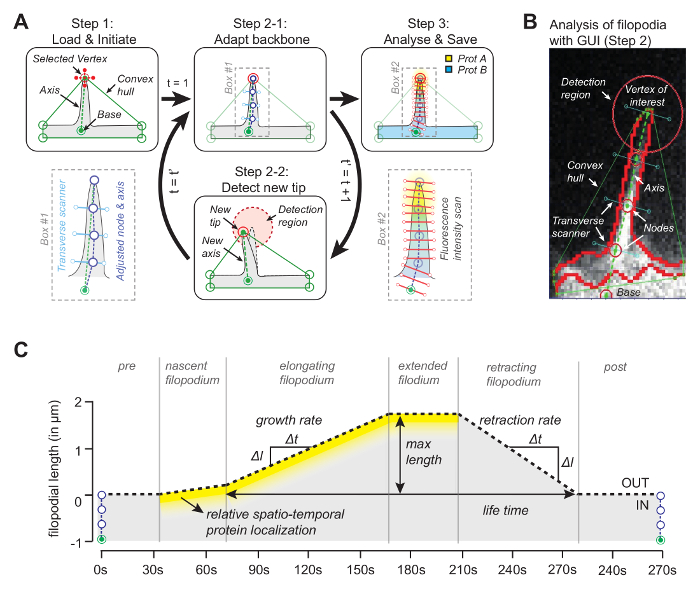
Figur 1: Arbejdsprincip for billedanalysesoftwaren. ( A ) Algoritmen bagvedSoftwaren. I trin 1 specificerer brugeren referencen (basen) og toppunktet (spidsen) af filopodiet. I trin 2-1 opnås filopodiumets rygrad ved hjælp af median pixel med maksimal intensitetsværdi. I trin 3 anvendes rygraden til rumlig proteinintensitetsprofil. I trin 2-2 sporer softwaren automatisk spidsen i den efterfølgende ramme. Hele proceduren gentager. ( B ) Snapshot af algoritmen med ægte filopodium, der introducerer vigtige elementer som det konvekse skrog, der bruges til sporing. ( C ) Oversigt over parametre, som kan måles med algoritmen. Dette tal er blevet ændret fra reference 12 . Klik her for at se en større version af denne figur.
Billedanalyseprogrammet betjenes i Matlab (benævnt programmeringssoftware) via en grafisk brugR interface. For at maksimere fleksibilitet og robusthed til den pågældende eksperimentelle indstilling kan brugeren justere en række sporingsparametre ( f.eks. Tilladt bøjningsvinkel og mellemrammebevægelse) og også foretage nogle korrektioner i filmene ( f.eks. Beskæring, rotation, fjernelse af uønskede objekter) ( Figur 2A og tabel 1) .
| GUI | Ingen. | Obligatorisk | Beskrivelse | Navn (i GUI) | ||||
| # 1 | 1a | Y | Indlæsning af stablet .tiff-fil, der repræsenterer cellelegemet (med boks tjekket ind) eller oprettet overlejret cellelegeme fra kanaler | CellBody | ||||
| # 1 | 1b | Y | <td colspan="5"> Ilægning af stablet billedfil svarende til protein 1Protein 1 | |||||
| # 1 | 1c | Y | Indlæser stablet billedfil svarende til protein 2 | Protein 2 | ||||
| # 1 | 1d | Y | Ilægning af stablet billedfil svarende til protein 3 | Protein 3 | ||||
| # 1 | 1e | N | Nulstiller alt til forudindlæste stablede billedfiler | Nulstille | ||||
| # 1 | 2a | Y | Rulestang for at bestemme den indledende ramme til analyse i GUI-vindue nr. 2 | NA | ||||
| # 1 | 2b | Y | Rulestang for at bestemme den endelige ramme til analyse i GUI vindue nr. 2 | NA | ||||
| # 1 | 2c | Y | Rullefelt repræsenterer curreNt ramme | NA | ||||
| # 1 | 2d | N | Den grå værdi af pixlerne nedenfor, hvor alle pixels vil blive indstillet til nul | NA | ||||
| # 1 | 2e | N | Den grå værdi af pixlerne over hvilke alle pixels vil blive indstillet til maksimale værdier | NA | ||||
| # 1 | 2f | N | Indstil intensitetsværdierne for de pixels, der er angivet af <2e> & <2f> | Sæt | ||||
| # 1 | 2g | N | Afspil den intensitetsjusterede film | Spille | ||||
| # 1 | 2h | N | Beskær billede | Afgrøde | ||||
| # 1 | 2i | N | Rotér billede | Rotere | ||||
| # 1 | 2j | N | Slet regioner i hele stakken | Slet regioner | ||||
| # 1 | 3a | Y | Klik for at åbne 'Analysevindue' (GUI vindue nr. 2) | Tracking Window | ||||
| # 1 | 3b | Y | Indtast størrelsen på en pixel i mikroner | Indtast pixelstørrelse | ||||
| # 2 | 4 | Y | Klik for at generere grænsen / kantbilledet af den overlejrede cellekrop | Grænse | ||||
| # 2 | 5 | Y | Klik for at vælge base og tip af filopodien | Referernce | ||||
| # 2 | 6a | Y | Indtast antallet af segmenter eller noder | Ingen af segmenter | ||||
| # 2 | 6b | Y | Indtast scanningslængden (vinkelret på akse) | Scanbredde | ||||
| # 2 | 6c | Y | Indtast længden over hvilken filopodia begynder at bøje | Nøjagtige mål efter | ||||
| # 2 | 6d | Y | Indtast radius af tipdetektionscirklen (dvs. område, hvor vertexet kan lokaliseres i den næste ramme) | Radius af spidsopdagelse | ||||
| # 2 | 6e | Y | Indtast den maksimale vinkel, som filopodiet kan bøjes fra den vertikale akse | Vinkelgrænse | ||||
| # 2 | 6f | N | Tilføj referencepunkter for base og tip til den specifikke ramme | Vælg reference | ||||
| # 2 | 6g | N | Indtast længden over hvilken filopodia begynder at bøje for den pågældende ramme | Nøjagtige mål efter | ||||
| # 2 | <td> 6hN | Indtast radius for detektionscirkel for den pågældende ramme | Radius af spidsopdagelse | |||||
| # 2 | 6i | N | Efter indtastning af alle parametrene for den specifikke ramme klik for at gemme værdierne til hukommelse og fil for yderligere reference | Tilføje | ||||
| # 2 | 6j | N | Klik for at slette de indstillede manuelt parametre for den pågældende ramme | Slet | ||||
| # 2 | 6k | N | Klik for at slette alle parametre, der er gemt manuelt, ved hjælp af panelet "valgfrit funktioner" for alle rammer | Nulstille | ||||
| # 2 | 6l | N | Check ind før sporing for at gemme alle sporingsresultater i hukommelsen for fremtidig reference | Historie spor | ||||
| # 2 | 7 | Y | Klik for at starte sporing | Spor & Analyse | ||||
| # 2 | 8a | N | Klik for at begynde at spore proteinkanalintensitet | Analyser proteinintensiteter | ||||
| # 2 | 8b | N | Check ind for at spore proteinkanalintensitet langs filopodiallængden | Hele filopodier | ||||
| # 2 | 8c | N | Check ind for at spore referenceproteinet eller proteinet A | protein A | ||||
| # 2 | 8d | N | Check ind for at spore protein B | ProteinB | ||||
| # 2 | 8e | N | Check ind for at spore proteinet C | ProteinC | ||||
| # 2 | 8f | N | Check ind for at spore gennemsnitlig proteinintensitet i thE tip | Ledende tip | ||||
| # 2 | 8g | N | Indtast spidsens længde | Tip længde | ||||
| # 2 | 8h | N | Indtast den minimale afstand fra bunden, over hvilken spidsen begynder at danne | Grænseværdi | ||||
| # 2 | 8i | N | Klik for at gemme ledende tipanalyseresultater til filen | Trykknap | ||||
| # 2 | 9a | N | Klik for at indlede ratiometrisk proteinanalyse | Sammenligne | ||||
| # 2 | 9b | N | Check ind for at sammenligne protein B med hensyn til A | log10 (B / A) | ||||
| # 2 | 9c | N | Check ind for at sammenligne protein C med hensyn til A | log10 (C / A) | ||||
| # 2 | <td> 9dN | Check ind for at sammenligne protein B med hensyn til A på spidsen | log10 (B / A) | |||||
| # 2 | 9e | N | Check ind for at sammenligne protein C med hensyn til A på spidsen | log10 (C / A) | ||||
| # 2 | 10a | N | Vælg andet farvekort (standard: Jetplot) | Farvekort | ||||
| # 2 | 10b | N | Rediger colormap | Rediger colormap | ||||
Tabel 1: Oversigt over alle funktioner, der findes i GUI Windows # 1 og # 2.
Når dette er opnået, skaber programmet et konvekst skrog og sporer automatisk spidsen i hele filmen. Parametre uddraget fra filmen, såsom en ratiometrisk kymografi, væksthastighed og filopodial længde aVises igen og gemmes også i arbejdsmappen som billeder og som datafiler. Andre parametre såsom filopodial levetid, væksthastighed og tilbagetrækningsgrad kan derefter ekstraheres og analyseres yderligere fra de lagrede datafiler ( figur 2B ).
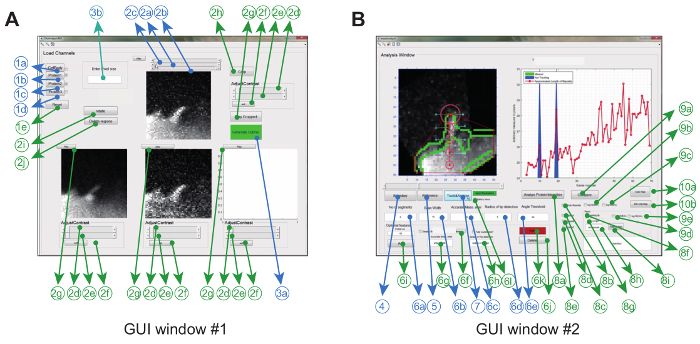
Figur 2: Grafisk brugergrænseflade til brug af Image Analysis Software. ( A ) GUI Window # 1 bruges til at indlæse og behandle billeder. Programmet kan indlæse op til 3 proteinkanaler, hvorved 2 kanaler sammenlignes parvis. Vinduet leveres med obligatorisk (blå) og valgfri egenskaber (grøn) til forbehandling af billederne forud for sporing ( B ) GUI Window # 2 bruges til at spore filopodium samt spatio-temporal og ratio-metrisk proteinanalyse. Igen er valgfrie funktioner markeret i grønt. Dette tal er blevet ændretUdgår fra reference 12 . Klik her for at se en større version af denne figur.
Her præsenterer vi en detaljeret protokol til prøveudarbejdelse og softwarehåndtering. Vi starter med detaljerede instruktioner om dyrkning af celler og erhvervelse af film optimeret til billedanalyse. Dette afsnit om dataindsamling efterfølges af en detaljeret beskrivelse for drift af billedanalysesoftwaren. Gennem protokollen introducerer vi kritiske trin og valgfrie funktioner, der bør overvejes, når data indsamles og behandles. Endelig analyserer vi filopodier fra forskellige modelsystemer med billedanalyseprogrammet inden lukning med en sammenligning af den beskrevne billedanalysesoftware med andre programmer til filopodial kvantificering og en diskussion om begrænsninger og fremtidig retning.
Protocol
Representative Results
Discussion
Her præsenterer vi en detaljeret protokol til sporing af filopodial vækstdynamik og analyse af relative proteinkoncentrationer i disse dynamiske strukturer via den konvekse skrogalgoritme. Ved hjælp af softwaren kan op til 3 kanaler sammenlignes parvis i et enkelt løb, hvorved de relative koncentrationer af to kanaler ( dvs. proteiner) bestemmes gennem hele forlængelses- / tilbagekredscyklusen og opbevares som billed- og datafiler i separate mapper. Udover de rutinemæssige operationer indeholder softwaren…
Declarações
The authors have nothing to disclose.
Acknowledgements
Forfatterne anerkender finansiering fra DFG (EXC-1003 til MG).
Materials
| DMEM | Life Technologies | 31966-021 | |
| 10% Fetal bovine serum | Biochrom AG | L11-044 | |
| Lipofectamine 2000 | Life Technologies | 11668-027 | |
| 1% penicillin/streptomycin | Biochrom AG | 12212 | |
| Neurobasal Medium | Life Technologies | 21103-049 | |
| B27 | Life Technologies | 17504-044 | |
| HEPES (1M stock solution) | Life Technologies | 15630 | |
| Citrine-N1 | Addgene | 54593 | |
| Labtech | Thermo | 155411 | |
| Glutamax-I | Thermo | 35050-061 | |
| Hela | Leibniz Institute DSMZ | ACC-57 | |
| COS 7 | Leibniz Institute DSMZ | ACC-60 | |
| 3T3 cells | Leibniz Institute DSMZ | ACC-59 | |
| Microscope | Nicon Eclipse | ||
| Camera | Andor | DU888 Ultra | |
| Confocal Unit | Yokagawa | CSU-X1 | |
| Pyruvate | Gibco | 31966-021 |
Referências
- Dunaevsky, A., Tashiro, A., Majewska, A., Mason, C., Yuste, R. Developmental regulation of spine motility in the mammalian central nervous system. Proc Natl Acad Sci U S A. 96 (23), 13438-13443 (1999).
- Matus, A., Brinkhaus, H., Wagner, U. Actin dynamics in dendritic spines: a form of regulated plasticity at excitatory synapses. Hippocampus. 10 (5), 555-560 (2000).
- Galic, M., et al. Dynamic recruitment of the curvature-sensitive protein ArhGAP44 to nanoscale membrane deformations limits exploratory filopodia initiation in neurons. Elife. 3, e03116 (2014).
- Hotulainen, P., et al. Defining mechanisms of actin polymerization and depolymerization during dendritic spine morphogenesis. J Cell Biol. 185 (2), 323-339 (2009).
- Leijnse, N., Oddershede, L. B., Bendix, P. M. Helical buckling of actin inside filopodia generates traction. Proc Natl Acad Sci U S A. 112 (1), 136-141 (2015).
- Tsygankov, D., et al. CellGeo: a computational platform for the analysis of shape changes in cells with complex geometries. J Cell Biol. 204 (3), 443-460 (2014).
- Xiong, Y., et al. Automated characterization of cell shape changes during amoeboid motility by skeletonization. BMC Syst Biol. 4, 33 (2010).
- Styner, M., Gerig, G., Lieberman, J., Jones, D., Weinberger, D. Statistical shape analysis of neuroanatomical structures based on medial models. Med Image Anal. 7 (3), 207-220 (2003).
- Blum, H., Wathen-Dunn, W. A transformation for extracting new descriptors of shape. Models for the Perception of Speech and Visual Form: Proceedings of a Symposium. , 362-380 (1967).
- Barry, D. J., Durkin, C. H., Abella, J. V., Way, M. Open source software for quantification of cell migration, protrusions, and fluorescence intensities. J Cell Biol. 209 (1), 163-180 (2015).
- Machacek, M., et al. Coordination of Rho GTPase activities during cell protrusion. Nature. 461 (7260), 99-103 (2009).
- Saha, T., et al. Automated analysis of filopodial length and spatially resolved protein concentration via adaptive shape tracking. Mol Biol Cell. 27 (22), 3616-3626 (2016).
- Argiro, V., Bunge, M. B., Johnson, M. I. A quantitative study of growth cone filopodial extension. J Neurosci Res. 13 (1-2), 149-162 (1985).
- Mogilner, A., Rubinstein, B. The physics of filopodial protrusion. Biophys J. 89 (2), 782-795 (2005).
- Shaner, N. C., Steinbach, P. A., Tsien, R. Y. A guide to choosing fluorescent proteins. Nat Methods. 2 (12), 905-909 (2005).
- Abramoff, M. D., Magalhaes, P. J., Ram, S. J. Image Processing with ImageJ. Biophotonics International. 11, 36-42 (2004).
- Courtney, J., Woods, E., Scholz, D., Hall, W. W., Gautier, V. W. MATtrack: A MATLAB-Based Quantitative Image Analysis Platform for Investigating Real-Time Photo-Converted Fluorescent Signals in Live Cells. PLoS One. 10 (10), e0140209 (2015).
- Schell, M. J., Erneux, C., Irvine, R. F. Inositol 1,4,5-trisphosphate 3-kinase A associates with F-actin and dendritic spines via its N terminus. J Biol Chem. 276 (40), 37537-37546 (2001).
- Korobova, F., Svitkina, T. Molecular architecture of synaptic actin cytoskeleton in hippocampal neurons reveals a mechanism of dendritic spine morphogenesis. Mol Biol Cell. 21 (1), 165-176 (2010).
- Cheadle, L., Biederer, T. The novel synaptogenic protein Farp1 links postsynaptic cytoskeletal dynamics and transsynaptic organization. J Cell Biol. 199 (6), 985-1001 (2012).
- Tarnok, K., et al. A new tool for the quantitative analysis of dendritic filopodial motility. Cytometry A. 87 (1), 89-96 (2015).
- Hendricusdottir, R., Bergmann, J. H. F-dynamics: automated quantification of dendrite filopodia dynamics in living neurons. J Neurosci Methods. 236, 148-156 (2014).
- Fanti, Z., Martinez-Perez, M. E., De-Miguel, F. F. NeuronGrowth, a software for automatic quantification of neurite and filopodial dynamics from time-lapse sequences of digital images. Dev Neurobiol. 71 (10), 870-881 (2011).
- Costantino, S., et al. Semi-automated quantification of filopodial dynamics. J Neurosci Methods. 171 (1), 165-173 (2008).
- Nilufar, S., Morrow, A. A., Lee, J. M., Perkins, T. J. FiloDetect: automatic detection of filopodia from fluorescence microscopy images. BMC Syst Biol. 7, 66 (2013).

