Un'interfaccia utente grafica per il monitoraggio assistito della concentrazione di proteine in protrusioni cellulari dinamiche
Summary
Presentiamo una soluzione software per il monitoraggio semi-automatico della concentrazione proteica relativa lungo la lunghezza delle protuberanze cellulari dinamiche.
Abstract
Le filopodie sono dinamiche, protrusioni cellulari simili a dito associate alla migrazione e alla comunicazione cellulare. Per comprendere meglio i complessi meccanismi di segnalazione sottostanti all'iniziazione filosofica, all'allungamento e alla successiva stabilizzazione o ritrazione, è fondamentale determinare l'attività proteica spazi-temporale in queste strutture dinamiche. Per analizzare la funzione proteica in filopodi, abbiamo recentemente sviluppato un algoritmo semi-automatizzato di tracciamento che si adatta a cambiamenti di forma filopodiale consentendo così un'analisi parallela delle dinamiche di protrusione e della concentrazione di proteine relative lungo tutta la lunghezza filopodiale. Qui presentiamo un dettagliato protocollo dettagliato per la gestione delle celle ottimizzate, l'acquisizione di immagini e l'analisi del software. Inoltre forniamo istruzioni per l'utilizzo delle funzioni opzionali durante l'analisi delle immagini e la rappresentazione dei dati, nonché le linee guida per la risoluzione dei problemi per tutti i passaggi critici lungo la strada. Infine, includiamo anche un confronto del dSoftware di analisi di immagini escribed con altri programmi disponibili per la quantificazione filopodia. Insieme, il protocollo presentato fornisce un quadro per un'analisi accurata delle dinamiche proteiche nelle protuberanze filopodiali usando il software di analisi delle immagini.
Introduction
Il controllo spazio-temporale delle proteine regolatrici dell'atina è associato alla dinamica dei filopodium 1 , 2 . Il monitoraggio della concentrazione proteica spazialmente risolto lungo tutta la lunghezza filopodiale nel tempo è quindi cruciale per favorire la comprensione dei meccanismi che sottendono l'iniziazione, l'allungamento, la stabilizzazione o il crollo di queste strutture dinamiche 3 , 4 . A differenza dell'analisi proteica nel citosolo, in cui si verificano molteplici cambiamenti di forma cellulare in una scala più ampia, le filopodie sono microstrutture dinamiche che costantemente stringono 5 e si curvano, evitando così l'analisi utilizzando un approccio semplice come una scansione di linea.
Diverse soluzioni software per il monitoraggio della forma filopodiale sono disponibili 6 , 7 , 8 , 9 . LikewÈ stato sviluppato software per il tracking ratiometrico delle dinamiche proteiche all'interno del corpo cellulare 10 , 11 . Per combinare il monitoraggio automatizzato della forma filopodiale e dell'analisi proteica spaziometrica, abbiamo recentemente sviluppato un software di analisi delle immagini basato sull'algoritmo convesso 12 . Questo nuovo metodo di analisi, gestito tramite un'interfaccia grafica (GUI), combina per la prima volta la concentrazione di proteine relativa lungo la lunghezza filopodiale e la velocità di crescita, consentendo così la misurazione accurata della distribuzione proteica spazi-temporale indipendente dal movimento di queste Strutture dinamiche 12 .
L'idea dietro il software (codice sorgente è liberamente disponibile, vedi sotto) è che uno dei vertici dello scafo convesso coinciderà con la punta del filopodium ( Figura 1A ). Guardando nella cornice successivaR il vertice più vicino dello scafo convesso, la punta in movimento può essere tracciata in tutto il filmato. Una volta che la punta viene rilevata in ogni telaio, la sua posizione viene usata per disegnare un asse unendosi alla punta con un punto di riferimento alla base del filopodium ( figura 1B ). Infine, usando punti nodali equidistanti, le cui posizioni sono determinate dal pixel mediano con intensità massima lungo la linea ortogonale all'asse, vengono utilizzati per determinare una spina dorsale che segue la forma filopodiale. Approfittando di questa base adattativa, viene generato un kymograph per tracciare la crescita filopodiale e le concentrazioni proteiche per un massimo di tre canali lungo la lunghezza filopodiale ( Figura 1C ).
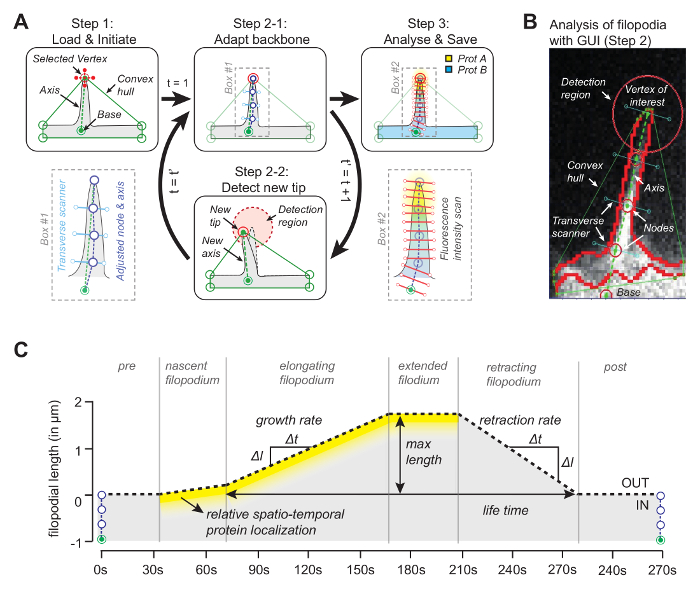
Figura 1: Principio di funzionamento del software di analisi delle immagini. ( A ) L'algoritmo dietroil software. In Step1 l'utente specifica il riferimento (base) e il vertice (la punta) del filopodium. Nel passaggio 2-1 si ottiene la spina dorsale del filopodium usando il pixel mediano con il valore massimo di intensità. Nel passaggio 3 la spina dorsale è utilizzata per il profilo di intensità proteica spaziale. Nel passaggio 2-2 il software segue automaticamente la punta nel fotogramma successivo. L'intera procedura si estende. ( B ) Snapshot dell'algoritmo con filopodium reale introducendo elementi importanti come lo scafo convesso che viene utilizzato per il tracciamento. ( C ) Panoramica dei parametri che possono essere misurati con l'algoritmo. Questa cifra è stata modificata dal riferimento 12 . Clicca qui per visualizzare una versione più grande di questa figura.
Il software di analisi delle immagini viene utilizzato in Matlab (indicato come software di programmazione) tramite un uso graficoR interfaccia. Per massimizzare la flessibilità e la robustezza per la particolare impostazione sperimentale, l'utente può regolare una serie di parametri di tracciamento ( ad esempio l'angolo di flusso consentito e il movimento inter-frame) e anche fare alcune correzioni ai filmati ( ad es. Ritaglio, rotazione, rimozione di oggetti indesiderati) ( Figura 2A e Tabella 1) .
| GUI | No. | Obbligatorio | Descrizione | Nome (in GUI) | ||||
| # 1 | 1a | Y | Caricare un file .tiff impilato che rappresenta il corpo cellulare (con casella controllata) o crea un corpo cellulare sovrapposto dai canali | Corpo cellulare | ||||
| # 1 | 1b | Y | <td colspan="5"> Caricamento di file di immagini sovrapposte che corrispondono alla proteina 1Proteina 1 | |||||
| # 1 | 1c | Y | Caricamento di un file di immagine impilato corrispondente alla proteina 2 | Proteina 2 | ||||
| # 1 | 1d | Y | Caricare il file di immagine sovrapposto alla proteina 3 | Proteina 3 | ||||
| # 1 | 1e | N | Ripristina tutti i file di immagine impilati precaricati | Reset | ||||
| # 1 | 2a | Y | Barra di scorrimento per determinare il frame iniziale per l'analisi nella finestra GUI # 2 | N / A | ||||
| # 1 | 2b | Y | Barra di scorrimento per determinare il frame finale per l'analisi nella finestra GUI # 2 | N / A | ||||
| # 1 | 2c | Y | Barra di scorrimento che rappresenta la curraNt frame | N / A | ||||
| # 1 | 2d | N | Il valore grigio dei pixel al di sotto di cui tutti i pixel saranno impostati su zero | N / A | ||||
| # 1 | 2e | N | Il valore grigio dei pixel al di sopra del quale tutti i pixel saranno impostati sui valori massimi | N / A | ||||
| # 1 | 2f | N | Impostare i valori di intensità dei pixel specificati da <2e> e <2f> | Impostato | ||||
| # 1 | 2g | N | Riprodurre il filmato regolato dall'intensità | Giocare | ||||
| # 1 | 2h | N | Ritaglia l'immagine | raccolto | ||||
| # 1 | 2i | N | Ruota l'immagine | Ruotare | ||||
| # 1 | 2J | N | Cancellare le regioni in tutta la pila | Elimina regioni | ||||
| # 1 | 3a | Y | Fare clic per aprire la finestra di analisi (finestra GUI # 2) | Finestra di monitoraggio | ||||
| # 1 | 3b | Y | Immettere le dimensioni di un pixel in micron | Inserisci dimensione pixel | ||||
| # 2 | 4 | Y | Fare clic per generare l'immagine del bordo / bordo del corpo cellulare sovrapposto | Confine | ||||
| # 2 | 5 | Y | Clicca per selezionare la base e la punta della filopodia | Referernce | ||||
| # 2 | 6a | Y | Inserisci il numero di segmenti o nodi | No di segmenti | ||||
| # 2 | 6b | Y | Inserisci la lunghezza della scansione (perpendicolare all'asse) | Larghezza di scansione | ||||
| # 2 | 6c | Y | Immettere la lunghezza sopra la quale filopodia inizia a piegarsi | Accurate misura dopo | ||||
| # 2 | 6d | Y | Inserisci il raggio del cerchio di rilevamento della punta (ovvero l'area in cui il vertice può essere localizzato nel fotogramma successivo) | Raggio di rilevamento delle punte | ||||
| # 2 | 6e | Y | Inserisci l'angolo massimo che il filopodium può piegarsi dall'asse verticale | Soglia di angolo | ||||
| # 2 | 6f | N | Aggiungere punti di riferimento per la base e la punta per quella specifica cornice | Seleziona il riferimento | ||||
| # 2 | 6g | N | Immettere la lunghezza sopra la quale filopodia inizia a piegarsi per quel particolare fotogramma | Accurate misura dopo | ||||
| # 2 | <td> 6hN | Inserisci il raggio del cerchio di rilevamento per quel fotogramma specifico | Raggio di rilevamento delle punte | |||||
| # 2 | 6i | N | Dopo aver inserito tutti i parametri per il frame specifico cliccare per memorizzare i valori in memoria e file per ulteriori riferimenti | Inserisci | ||||
| # 2 | 6j | N | Fare clic per eliminare i parametri impostati manualmente per quel fotogramma | Elimina | ||||
| # 2 | 6K | N | Fare clic per eliminare tutti i parametri memorizzati manualmente utilizzando il pannello delle funzioni opzionali per tutti i fotogrammi | Reset | ||||
| # 2 | 6L | N | Accedere prima del monitoraggio per memorizzare tutti i risultati di monitoraggio in memoria per futuri riferimenti | Traccia di storia | ||||
| # 2 | 7 | Y | Fai clic per avviare il tracciamento | Traccia e Analisi | ||||
| # 2 | 8a | N | Fai clic per iniziare a monitorare l'intensità del canale proteico | Analizzare le intensità proteiche | ||||
| # 2 | 8b | N | Entra per monitorare l'intensità del canale proteico lungo la lunghezza filopodiale | Tutta la filopodia | ||||
| # 2 | 8c | N | Entra per il monitoraggio della proteina o della proteina di riferimento A | proteína | ||||
| # 2 | 8d | N | Entra per il monitoraggio della proteina B | ProteinB | ||||
| # 2 | 8e | N | Entra per il monitoraggio della proteina C | ProteinC | ||||
| # 2 | 8f | N | Entra per tenere traccia dell'intensità media della proteina in thE punta | Punta dirigente | ||||
| # 2 | 8g | N | Inserire la lunghezza della punta | Tip Lunghezza | ||||
| # 2 | 8h | N | Immettere la distanza minima dalla base sopra la quale la punta comincia a formarsi | soglia | ||||
| # 2 | 8i | N | Fare clic per salvare i risultati principali dell'analisi dei suggerimenti in file | Premi il bottone | ||||
| # 2 | 9a | N | Fare clic per avviare analisi proteometrica delle proteine | Confrontare | ||||
| # 2 | 9b | N | Controlla per confrontare la proteina B rispetto a A | log10 (B / A) | ||||
| # 2 | 9c | N | Controlla per confrontare la proteina C rispetto a A | log10 (C / A) | ||||
| # 2 | <td> 9dN | Controlla per confrontare la proteina B rispetto a A alla punta | log10 (B / A) | |||||
| # 2 | 9e | N | Controlla per confrontare la proteina C rispetto a A alla punta | log10 (C / A) | ||||
| # 2 | 10a | N | Scegli un'altra mappa a colori (impostazione predefinita: Jetplot) | Mappa del colore | ||||
| # 2 | 10b | N | Modifica la colormap | Modifica colormap | ||||
Tabella 1: Panoramica di tutte le funzioni presenti nella GUI Windows # 1 e # 2.
Una volta raggiunto questo obiettivo, il programma crea uno scafo convesso e traccia automaticamente la punta in tutto il filmato. Parametri estratti dal film, come un kymograph ratiometrico, velocità di crescita e lunghezza filopodiale aVengono visualizzati e memorizzati nella cartella di lavoro come immagini e come file di dati. Possono poi essere estratti altri parametri, come la durata della vita filopodiale, la velocità di crescita e la frequenza di ritiro e vengono ulteriormente analizzati dai file di dati memorizzati ( Figura 2B ).
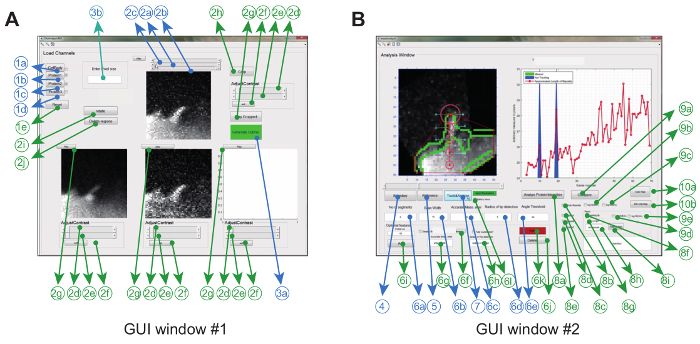
Figura 2: Interfaccia grafica utente per l'utilizzo del software di analisi delle immagini. ( A ) La finestra # 1 della GUI viene utilizzata per il caricamento e l'elaborazione di immagini. Il programma può caricare fino a 3 canali proteici, per cui 2 canali vengono confrontati in coppia. La finestra viene fornita con le funzioni obbligatorie (blu) e facoltative (verde) per la pre-elaborazione delle immagini prima della tracciabilità ( B ) La finestra # 2 della GUI viene utilizzata per il monitoraggio dell'analisi della proteina filopodica e spaziometrica e rapporto-metrica. Ancora una volta, le caratteristiche facoltative sono contrassegnate in verde. Questa cifra è stata modificataDal riferimento 12 . Clicca qui per visualizzare una versione più grande di questa figura.
Qui presentiamo un protocollo dettagliato per la preparazione del campione e la gestione del software. Iniziamo con istruzioni dettagliate sulle cellule di coltura e l'acquisizione di filmati ottimizzati per l'analisi delle immagini. Questa sezione relativa all'acquisizione dati è seguita da una descrizione dettagliata per l'utilizzo del software di analisi delle immagini. Durante tutto il protocollo, vengono introdotti passi critici e funzionalità opzionali che dovrebbero essere considerate durante la raccolta e l'elaborazione dei dati. Infine, analizziamo filopodi da diversi sistemi di modello con il software di analisi delle immagini, prima di chiudere con un confronto del software di analisi delle immagini descritto con altri programmi disponibili per la quantificazione filopodiale e una discussione sulle limitazioni e la direzione futura.
Protocol
Representative Results
Discussion
Qui presentiamo un protocollo dettagliato per il monitoraggio delle dinamiche di crescita filopodale e l'analisi di concentrazioni di proteine relative in queste strutture dinamiche attraverso l'algoritmo convesso. Utilizzando il software, fino a 3 canali possono essere confrontati in coppia in una singola esecuzione, per cui le relative concentrazioni di due canali ( cioè le proteine) vengono determinate durante il ciclo di estensione / ritrazione e memorizzate come file immagine e dati in carte…
Declarações
The authors have nothing to disclose.
Acknowledgements
Gli autori riconoscono i finanziamenti del DFG (EXC-1003 a MG).
Materials
| DMEM | Life Technologies | 31966-021 | |
| 10% Fetal bovine serum | Biochrom AG | L11-044 | |
| Lipofectamine 2000 | Life Technologies | 11668-027 | |
| 1% penicillin/streptomycin | Biochrom AG | 12212 | |
| Neurobasal Medium | Life Technologies | 21103-049 | |
| B27 | Life Technologies | 17504-044 | |
| HEPES (1M stock solution) | Life Technologies | 15630 | |
| Citrine-N1 | Addgene | 54593 | |
| Labtech | Thermo | 155411 | |
| Glutamax-I | Thermo | 35050-061 | |
| Hela | Leibniz Institute DSMZ | ACC-57 | |
| COS 7 | Leibniz Institute DSMZ | ACC-60 | |
| 3T3 cells | Leibniz Institute DSMZ | ACC-59 | |
| Microscope | Nicon Eclipse | ||
| Camera | Andor | DU888 Ultra | |
| Confocal Unit | Yokagawa | CSU-X1 | |
| Pyruvate | Gibco | 31966-021 |
Referências
- Dunaevsky, A., Tashiro, A., Majewska, A., Mason, C., Yuste, R. Developmental regulation of spine motility in the mammalian central nervous system. Proc Natl Acad Sci U S A. 96 (23), 13438-13443 (1999).
- Matus, A., Brinkhaus, H., Wagner, U. Actin dynamics in dendritic spines: a form of regulated plasticity at excitatory synapses. Hippocampus. 10 (5), 555-560 (2000).
- Galic, M., et al. Dynamic recruitment of the curvature-sensitive protein ArhGAP44 to nanoscale membrane deformations limits exploratory filopodia initiation in neurons. Elife. 3, e03116 (2014).
- Hotulainen, P., et al. Defining mechanisms of actin polymerization and depolymerization during dendritic spine morphogenesis. J Cell Biol. 185 (2), 323-339 (2009).
- Leijnse, N., Oddershede, L. B., Bendix, P. M. Helical buckling of actin inside filopodia generates traction. Proc Natl Acad Sci U S A. 112 (1), 136-141 (2015).
- Tsygankov, D., et al. CellGeo: a computational platform for the analysis of shape changes in cells with complex geometries. J Cell Biol. 204 (3), 443-460 (2014).
- Xiong, Y., et al. Automated characterization of cell shape changes during amoeboid motility by skeletonization. BMC Syst Biol. 4, 33 (2010).
- Styner, M., Gerig, G., Lieberman, J., Jones, D., Weinberger, D. Statistical shape analysis of neuroanatomical structures based on medial models. Med Image Anal. 7 (3), 207-220 (2003).
- Blum, H., Wathen-Dunn, W. A transformation for extracting new descriptors of shape. Models for the Perception of Speech and Visual Form: Proceedings of a Symposium. , 362-380 (1967).
- Barry, D. J., Durkin, C. H., Abella, J. V., Way, M. Open source software for quantification of cell migration, protrusions, and fluorescence intensities. J Cell Biol. 209 (1), 163-180 (2015).
- Machacek, M., et al. Coordination of Rho GTPase activities during cell protrusion. Nature. 461 (7260), 99-103 (2009).
- Saha, T., et al. Automated analysis of filopodial length and spatially resolved protein concentration via adaptive shape tracking. Mol Biol Cell. 27 (22), 3616-3626 (2016).
- Argiro, V., Bunge, M. B., Johnson, M. I. A quantitative study of growth cone filopodial extension. J Neurosci Res. 13 (1-2), 149-162 (1985).
- Mogilner, A., Rubinstein, B. The physics of filopodial protrusion. Biophys J. 89 (2), 782-795 (2005).
- Shaner, N. C., Steinbach, P. A., Tsien, R. Y. A guide to choosing fluorescent proteins. Nat Methods. 2 (12), 905-909 (2005).
- Abramoff, M. D., Magalhaes, P. J., Ram, S. J. Image Processing with ImageJ. Biophotonics International. 11, 36-42 (2004).
- Courtney, J., Woods, E., Scholz, D., Hall, W. W., Gautier, V. W. MATtrack: A MATLAB-Based Quantitative Image Analysis Platform for Investigating Real-Time Photo-Converted Fluorescent Signals in Live Cells. PLoS One. 10 (10), e0140209 (2015).
- Schell, M. J., Erneux, C., Irvine, R. F. Inositol 1,4,5-trisphosphate 3-kinase A associates with F-actin and dendritic spines via its N terminus. J Biol Chem. 276 (40), 37537-37546 (2001).
- Korobova, F., Svitkina, T. Molecular architecture of synaptic actin cytoskeleton in hippocampal neurons reveals a mechanism of dendritic spine morphogenesis. Mol Biol Cell. 21 (1), 165-176 (2010).
- Cheadle, L., Biederer, T. The novel synaptogenic protein Farp1 links postsynaptic cytoskeletal dynamics and transsynaptic organization. J Cell Biol. 199 (6), 985-1001 (2012).
- Tarnok, K., et al. A new tool for the quantitative analysis of dendritic filopodial motility. Cytometry A. 87 (1), 89-96 (2015).
- Hendricusdottir, R., Bergmann, J. H. F-dynamics: automated quantification of dendrite filopodia dynamics in living neurons. J Neurosci Methods. 236, 148-156 (2014).
- Fanti, Z., Martinez-Perez, M. E., De-Miguel, F. F. NeuronGrowth, a software for automatic quantification of neurite and filopodial dynamics from time-lapse sequences of digital images. Dev Neurobiol. 71 (10), 870-881 (2011).
- Costantino, S., et al. Semi-automated quantification of filopodial dynamics. J Neurosci Methods. 171 (1), 165-173 (2008).
- Nilufar, S., Morrow, A. A., Lee, J. M., Perkins, T. J. FiloDetect: automatic detection of filopodia from fluorescence microscopy images. BMC Syst Biol. 7, 66 (2013).

