Et grafisk brukergrensesnitt for programvareassistert sporing av proteinkonsentrasjon i dynamiske cellulære protrusjoner
Summary
Vi presenterer en programvare løsning for semi-automatisert sporing av relativ protein konsentrasjon langs lengden av dynamiske cellulære fremspring.
Abstract
Filopodi er dynamiske, fingerlignende cellulære fremspring i forbindelse med migrasjon og cellecellekommunikasjon. For å bedre forstå de komplekse signalmekanismer som ligger til grunn for filopodial initiering, forlengelse og påfølgende stabilisering eller tilbaketrekking, er det avgjørende å bestemme den spatio-temporale proteinaktiviteten i disse dynamiske strukturer. For å analysere proteinfunksjonen i filopodi, utviklet vi nylig en halvautomatisert sporingsalgoritme som tilpasser seg filopodial forandringsendringer, slik at parallell analyse av fremspringsdynamikk og relativ proteinkonsentrasjon langs hele filopodiallengden blir mulig. Her presenterer vi en detaljert trinnvis protokoll for optimalisert cellehåndtering, bildeoppkjøp og programvareanalyse. Vi gir videre instruksjoner for bruk av valgfrie funksjoner under bildeanalyse og datarrepresentasjon, samt retningslinjer for feilsøking for alle kritiske skritt underveis. Til slutt inkluderer vi også en sammenligning av dEskorte bildeanalyseprogramvare med andre programmer tilgjengelig for kvantifisering av filopodier. Sammen gir den presenterte protokollen et rammeverk for nøyaktig analyse av proteindynamikk i filopodiale fremspring ved hjelp av bildeanalyseprogramvare.
Introduction
Spatio-temporal kontroll av actin regulatoriske proteiner er forbundet med filopodiumdynamikk 1 , 2 . Sporing av romlig oppløst proteinkonsentrasjon langs hele filopodiallengden gjennom tid er dermed avgjørende for å fremme forståelsen av mekanismene som ligger til grund for initiering, forlengelse, stabilisering eller sammenbrudd av disse dynamiske strukturer 3 , 4 . I motsetning til proteinanalyse i cytosol, hvor mange celleformendringer forekommer i større skala, er filopodier dynamiske mikrostrukturer som stadig spenner 5 og bøyer, og dermed utelukker analyse ved hjelp av en enkel tilnærming, for eksempel en linjeskanning.
Ulike programvareløsninger for sporing av filopodial form er tilgjengelig 6 , 7 , 8 , 9 . LikewIse, programvare for ratiometrisk sporing av proteindynamikk i cellekroppen er utviklet 10 , 11 . For å kombinere automatisert sporing av filopodial form og spatio-temporal proteinanalyse, har vi nylig utviklet en bildeanalyseprogramvare basert på konveks-skrogalgoritmen 12 . Denne nye analysemetoden, som drives via et grafisk brukergrensesnitt (GUI), kombinerer for første gang den relative proteinkonsentrasjonen langs filopodiallengden og veksthastigheten, slik at det muliggjør nøyaktig måling av spatiodimental proteinfordeling uavhengig av bevegelse av disse Dynamiske strukturer 12 .
Ideen bak programvaren (kildekoden er fritt tilgjengelig, se nedenfor) er at en av knutene til det konvekse skroget vil falle sammen med spissen av filopodiet ( figur 1A ). Ved å se i den etterfølgende rammen foR nærmeste toppunkt i det konvekse skroget, kan den bevegelige spissen spores gjennom hele filmen. Når spissen er oppdaget i hver ramme, brukes posisjonen til å tegne en akse ved å bli med spissen med et referansepunkt ved fotopodiumets base ( figur 1B ). Til slutt brukes ved hjelp av ekvivalente nodepunkter, hvis posisjoner bestemmes av medianpixelen med maksimal intensitet langs linjen ortogonalt til aksen, for å bestemme en ryggrad som følger filopodialformen. Ved å benytte seg av denne adaptive ryggraden, genereres en kymografi for å spore filopodial vekst og proteinkonsentrasjoner for opptil tre kanaler langs filopodiallengden ( figur 1C ).
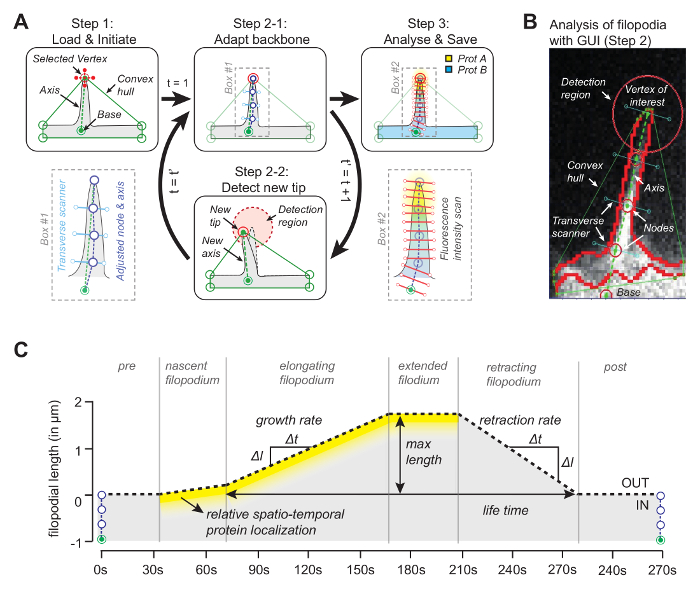
Figur 1: Arbeidsprinsipp for Image Analysis Software. ( A ) Algoritmen bakProgramvaren. I trinn 1 spesifiserer brukeren referansen (basen) og toppunktet (spissen) av filopodiet. I trinn 2-1 oppnås ryggradene i filopodium ved bruk av medianpixelen med maksimal intensitetsverdi. I trinn 3 brukes ryggraden for romlig proteinintensitetsprofil. I trinn 2-2 sporer programvaren automatisk spissen i den etterfølgende rammen. Hele prosedyren iterates. ( B ) Stillbilde av algoritmen med ekte filopodium introdusere viktige elementer som det konvekse skroget som brukes til sporing. ( C ) Oversikt over parametere som kan måles med algoritmen. Denne figuren er endret fra referanse 12 . Vennligst klikk her for å se en større versjon av denne figuren.
Bildeanalyseprogrammet betjenes i Matlab (referert til som programmeringsprogramvare) via en grafisk brukR grensesnitt. For å maksimere fleksibilitet og robusthet for den spesielle eksperimentelle innstillingen, kan brukeren justere en rekke sporingsparametere ( f.eks. Tillatt bøyevinkel og mellomrammebevegelse) og også gjøre noen korreksjoner til filmene ( f.eks. Beskjæring, rotasjon, fjerning av uønskede objekter) ( Figur 2A og tabell 1) .
| GUI | Nei. | Påbudt, bindende | Beskrivelse | Navn (i GUI) | ||||
| #1 | 1a | Y | Laster stablet .tiff-fil som representerer cellekropp (med boks innsjekket) eller opprett overliggende cellekropp fra kanaler | CellBody | ||||
| #1 | 1b | Y | <td colspan="5"> Laster stablet bildefil som svarer til protein 1Protein 1 | |||||
| #1 | 1c | Y | Laster stablet bildefil tilsvarende protein 2 | Protein 2 | ||||
| #1 | 1d | Y | Laster stablet bildefil tilsvarende protein 3 | Protein 3 | ||||
| #1 | 1e | N | Tilbakestiller alt til forhåndslastede stablede bildefiler | tilbakestille | ||||
| #1 | 2a | Y | Rullestang for å bestemme den innledende rammen for analyse i GUI-vinduet # 2 | NA | ||||
| #1 | 2b | Y | Rullestang for å bestemme den endelige rammen for analyse i GUI-vinduet # 2 | NA | ||||
| #1 | 2c | Y | Rullestang som representerer curreNt ramme | NA | ||||
| #1 | 2d | N | Den grå verdien av pikslene under hvilke alle piksler blir satt til null | NA | ||||
| #1 | 2e | N | Den grå verdien av pikslene over hvilke alle piksler blir satt til maksimale verdier | NA | ||||
| #1 | 2f | N | Angi intensitetsverdiene for pikslene angitt av <2e> & <2f> | Sett | ||||
| #1 | 2g | N | Spill den intensitetsjusterte filmen | Spille | ||||
| #1 | 2h | N | Beskjær bildet | Avling | ||||
| #1 | 2i | N | Roter bildet | Rotere | ||||
| #1 | 2j | N | Slett regioner i hele stakken | Slett regioner | ||||
| #1 | 3a | Y | Klikk for å åpne 'Analysevinduet' (GUI-vinduet # 2) | Sporingsvindu | ||||
| #1 | 3b | Y | Skriv inn størrelsen på en piksel i mikroner | Angi pixelstørrelse | ||||
| # 2 | 4 | Y | Klikk for å generere grense / kanten bildet av den overliggende celle kroppen | Grense | ||||
| # 2 | 5 | Y | Klikk for å velge base og tupp av filopodien | referanse- | ||||
| # 2 | 6a | Y | Skriv inn antall segmenter eller noder | Antall segmenter | ||||
| # 2 | 6b | Y | Angi skanne lengden (vinkelrett på aksen) | Skannebredde | ||||
| # 2 | 6c | Y | Skriv inn lengden over hvilken filopodien begynner å bøye | Nøyaktig Meas etter | ||||
| # 2 | 6d | Y | Skriv inn radiusen til tippingsdeteksjonssirkelen (dvs. område hvor toppunktet kan lokaliseres i neste ramme) | Radius av spissvarsling | ||||
| # 2 | 6e | Y | Angi den maksimale vinkelen filopodiet kan bøye fra den vertikale aksen | Vinkelgrense | ||||
| # 2 | 6f | N | Legg til referansepunkter for base og tips for den aktuelle rammen | Velg referanse | ||||
| # 2 | 6g | N | Skriv inn lengden over hvilken filopodien begynner å bøye for den aktuelle rammen | Nøyaktig Meas etter | ||||
| # 2 | <td> 6hN | Skriv inn radiusen til deteksjonssirkelen for den aktuelle rammen | Radius av spissvarsling | |||||
| # 2 | 6i | N | Etter å ha tastet alle parametrene for den bestemte rammen, klikker du for å lagre verdiene til minne og fil for videre referanse | Legg til | ||||
| # 2 | 6j | N | Klikk for å slette de angitte manuelt parametrene for den aktuelle rammen | Slett | ||||
| # 2 | 6k | N | Klikk for å slette alle parametrene som er lagret manuelt, ved hjelp av det valgfrie panelet for alle rammer | tilbakestille | ||||
| # 2 | 6L | N | Sjekk inn før sporing for å lagre alle sporingsresultater i minnet for fremtidig referanse | Historie spor | ||||
| # 2 | 7 | Y | Klikk for å starte sporing | Spor og Analyse | ||||
| # 2 | 8a | N | Klikk for å begynne å spore proteinkanalintensitet | Analyser proteinintensiteter | ||||
| # 2 | 8b | N | Sjekk inn for å spore proteinkanalintensitet langs filopodiallengden | Hele filopodi | ||||
| # 2 | 8c | N | Sjekk inn for å spore referanseproteinet eller proteinet A | ProteinA | ||||
| # 2 | 8d | N | Sjekk inn for sporing av protein B | ProteinB | ||||
| # 2 | 8e | N | Sjekk inn for å spore proteinet C | ProteinC | ||||
| # 2 | 8f | N | Sjekk inn for å spore gjennomsnittlig proteinintensitet i thE tips | Ledende tips | ||||
| # 2 | 8G | N | Skriv inn lengden på spissen | Tips Lengde | ||||
| # 2 | 8 timer | N | Angi minimumsavstanden fra basen over hvilken spissen begynner å danne | terskel | ||||
| # 2 | 8i | N | Klikk for å lagre ledende tipsanalyseresultatene til filen | Trykknapp | ||||
| # 2 | 9a | N | Klikk for å starte ratiometrisk proteinanalyse | Sammenligne | ||||
| # 2 | 9b | N | Sjekk inn for å sammenligne protein B med hensyn til A | log10 (B / A) | ||||
| # 2 | 9c | N | Sjekk inn for å sammenligne protein C med hensyn til A | log10 (C / A) | ||||
| # 2 | <td> 9dN | Sjekk inn for å sammenligne protein B med hensyn til A på spissen | log10 (B / A) | |||||
| # 2 | 9e | N | Sjekk inn for å sammenligne protein C med hensyn til A på spissen | log10 (C / A) | ||||
| # 2 | 10a | N | Velg annet fargekart (standard: Jetplot) | Fargekart | ||||
| # 2 | 10b | N | Rediger colormap | Rediger colormap | ||||
Tabell 1: Oversikt over alle funksjoner Present i GUI Windows # 1 og # 2.
Når dette er oppnådd, oppretter programmet et konvekst skrog og sporer automatisk spissen i hele filmen. Parametere hentet fra filmen, for eksempel en ratiometrisk kymografi, veksthastighet og filopodial lengde aVises og lagres også i arbeidsmappen som bilder og som datafiler. Andre parametere som filopodial levetid, veksthastighet og tilbaketrekningsrate kan deretter ekstraheres og analyseres videre fra de lagrede datafilene ( figur 2B ).
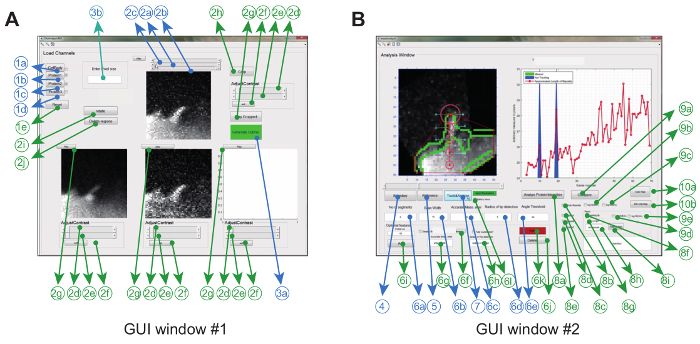
Figur 2: Grafisk brukergrensesnitt for bruk av Image Analysis Software. ( A ) GUI Window # 1 brukes til å laste og behandle bilder. Programmet kan laste opp opptil 3 proteinkanaler, hvorved to kanaler sammenlignes parvis. Vinduet leveres med obligatorisk (blå) og valgfrie funksjoner (grønn) for forhåndsbehandlingen av bildene før sporing. ( B ) GUI-vinduet # 2 brukes til å spore filopodium samt spatio-temporal og ratio-metrisk proteinanalyse. Igjen er valgfrie funksjoner merket i grønt. Denne figuren har blitt modifisertUtgitt fra referanse 12 . Vennligst klikk her for å se en større versjon av denne figuren.
Her presenterer vi en detaljert protokoll for prøvebehandling og programvarehåndtering. Vi starter med detaljerte instruksjoner om å dyrke celler og anskaffe filmer optimalisert for bildeanalyse. Denne delen om datainnsamling følges av en detaljert beskrivelse for drift av bildeanalyseprogrammet. Gjennom protokollen presenterer vi kritiske trinn og valgfrie funksjoner som bør vurderes når du samler og behandler data. Endelig analyserer vi filopodier fra forskjellige modellsystemer med bildeanalyseprogrammet, før de avsluttes med en sammenligning av den beskrevne bildanalysesoftware med andre programmer tilgjengelig for filopodial kvantifisering og en diskusjon om begrensninger og fremtidig retning.
Protocol
Representative Results
Discussion
Her presenterer vi en detaljert protokoll for sporing av filopodial vekstdynamikk og analyse av relative proteinkonsentrasjoner i disse dynamiske strukturer via den konvekse skrogalgoritmen. Ved hjelp av programvaren kan opptil 3 kanaler sammenlignes parvis i en enkelt runde, hvorved de relative konsentrasjonene av to kanaler ( dvs. proteiner) bestemmes gjennom utvidelses- / tilbaketrekningssyklusen og lagres som bilde- og datafiler i separate mapper. I tillegg til rutineoperasjoner gir programvaren også en re…
Declarações
The authors have nothing to disclose.
Acknowledgements
Forfatterne bekrefter finansiering fra DFG (EXC-1003 til MG).
Materials
| DMEM | Life Technologies | 31966-021 | |
| 10% Fetal bovine serum | Biochrom AG | L11-044 | |
| Lipofectamine 2000 | Life Technologies | 11668-027 | |
| 1% penicillin/streptomycin | Biochrom AG | 12212 | |
| Neurobasal Medium | Life Technologies | 21103-049 | |
| B27 | Life Technologies | 17504-044 | |
| HEPES (1M stock solution) | Life Technologies | 15630 | |
| Citrine-N1 | Addgene | 54593 | |
| Labtech | Thermo | 155411 | |
| Glutamax-I | Thermo | 35050-061 | |
| Hela | Leibniz Institute DSMZ | ACC-57 | |
| COS 7 | Leibniz Institute DSMZ | ACC-60 | |
| 3T3 cells | Leibniz Institute DSMZ | ACC-59 | |
| Microscope | Nicon Eclipse | ||
| Camera | Andor | DU888 Ultra | |
| Confocal Unit | Yokagawa | CSU-X1 | |
| Pyruvate | Gibco | 31966-021 |
Referências
- Dunaevsky, A., Tashiro, A., Majewska, A., Mason, C., Yuste, R. Developmental regulation of spine motility in the mammalian central nervous system. Proc Natl Acad Sci U S A. 96 (23), 13438-13443 (1999).
- Matus, A., Brinkhaus, H., Wagner, U. Actin dynamics in dendritic spines: a form of regulated plasticity at excitatory synapses. Hippocampus. 10 (5), 555-560 (2000).
- Galic, M., et al. Dynamic recruitment of the curvature-sensitive protein ArhGAP44 to nanoscale membrane deformations limits exploratory filopodia initiation in neurons. Elife. 3, e03116 (2014).
- Hotulainen, P., et al. Defining mechanisms of actin polymerization and depolymerization during dendritic spine morphogenesis. J Cell Biol. 185 (2), 323-339 (2009).
- Leijnse, N., Oddershede, L. B., Bendix, P. M. Helical buckling of actin inside filopodia generates traction. Proc Natl Acad Sci U S A. 112 (1), 136-141 (2015).
- Tsygankov, D., et al. CellGeo: a computational platform for the analysis of shape changes in cells with complex geometries. J Cell Biol. 204 (3), 443-460 (2014).
- Xiong, Y., et al. Automated characterization of cell shape changes during amoeboid motility by skeletonization. BMC Syst Biol. 4, 33 (2010).
- Styner, M., Gerig, G., Lieberman, J., Jones, D., Weinberger, D. Statistical shape analysis of neuroanatomical structures based on medial models. Med Image Anal. 7 (3), 207-220 (2003).
- Blum, H., Wathen-Dunn, W. A transformation for extracting new descriptors of shape. Models for the Perception of Speech and Visual Form: Proceedings of a Symposium. , 362-380 (1967).
- Barry, D. J., Durkin, C. H., Abella, J. V., Way, M. Open source software for quantification of cell migration, protrusions, and fluorescence intensities. J Cell Biol. 209 (1), 163-180 (2015).
- Machacek, M., et al. Coordination of Rho GTPase activities during cell protrusion. Nature. 461 (7260), 99-103 (2009).
- Saha, T., et al. Automated analysis of filopodial length and spatially resolved protein concentration via adaptive shape tracking. Mol Biol Cell. 27 (22), 3616-3626 (2016).
- Argiro, V., Bunge, M. B., Johnson, M. I. A quantitative study of growth cone filopodial extension. J Neurosci Res. 13 (1-2), 149-162 (1985).
- Mogilner, A., Rubinstein, B. The physics of filopodial protrusion. Biophys J. 89 (2), 782-795 (2005).
- Shaner, N. C., Steinbach, P. A., Tsien, R. Y. A guide to choosing fluorescent proteins. Nat Methods. 2 (12), 905-909 (2005).
- Abramoff, M. D., Magalhaes, P. J., Ram, S. J. Image Processing with ImageJ. Biophotonics International. 11, 36-42 (2004).
- Courtney, J., Woods, E., Scholz, D., Hall, W. W., Gautier, V. W. MATtrack: A MATLAB-Based Quantitative Image Analysis Platform for Investigating Real-Time Photo-Converted Fluorescent Signals in Live Cells. PLoS One. 10 (10), e0140209 (2015).
- Schell, M. J., Erneux, C., Irvine, R. F. Inositol 1,4,5-trisphosphate 3-kinase A associates with F-actin and dendritic spines via its N terminus. J Biol Chem. 276 (40), 37537-37546 (2001).
- Korobova, F., Svitkina, T. Molecular architecture of synaptic actin cytoskeleton in hippocampal neurons reveals a mechanism of dendritic spine morphogenesis. Mol Biol Cell. 21 (1), 165-176 (2010).
- Cheadle, L., Biederer, T. The novel synaptogenic protein Farp1 links postsynaptic cytoskeletal dynamics and transsynaptic organization. J Cell Biol. 199 (6), 985-1001 (2012).
- Tarnok, K., et al. A new tool for the quantitative analysis of dendritic filopodial motility. Cytometry A. 87 (1), 89-96 (2015).
- Hendricusdottir, R., Bergmann, J. H. F-dynamics: automated quantification of dendrite filopodia dynamics in living neurons. J Neurosci Methods. 236, 148-156 (2014).
- Fanti, Z., Martinez-Perez, M. E., De-Miguel, F. F. NeuronGrowth, a software for automatic quantification of neurite and filopodial dynamics from time-lapse sequences of digital images. Dev Neurobiol. 71 (10), 870-881 (2011).
- Costantino, S., et al. Semi-automated quantification of filopodial dynamics. J Neurosci Methods. 171 (1), 165-173 (2008).
- Nilufar, S., Morrow, A. A., Lee, J. M., Perkins, T. J. FiloDetect: automatic detection of filopodia from fluorescence microscopy images. BMC Syst Biol. 7, 66 (2013).

