Uma interface de usuário gráfica para rastreamento assistido por software da concentração de proteínas em protrusões celulares dinâmicas
Summary
Apresentamos uma solução de software para o rastreamento semi-automatizado da concentração relativa de proteína ao longo do comprimento de protrusões celulares dinâmicas.
Abstract
Filopodia são protrusões celulares dinâmicas, semelhantes a dedos, associadas à migração e à comunicação célula-célula. Para entender melhor os complexos mecanismos de sinalização subjacentes à iniciação filopodial, alongamento e posterior estabilização ou retração, é crucial determinar a atividade da proteína espaço-temporal nessas estruturas dinâmicas. Para analisar a função de proteína em filopodia, desenvolvemos recentemente um algoritmo de rastreamento semi-automatizado que se adapta às mudanças de forma filopodial, permitindo análises paralelas da dinâmica de protrusão e da concentração relativa de proteína ao longo de todo o comprimento filopodial. Aqui, apresentamos um protocolo passo a passo detalhado para otimizar o gerenciamento de células, a aquisição de imagens e a análise de software. Além disso, fornecemos instruções para o uso de recursos opcionais durante a análise de imagem e representação de dados, bem como diretrizes de solução de problemas para todos os passos críticos ao longo do caminho. Finalmente, também incluímos uma comparação do dEscreveu software de análise de imagem com outros programas disponíveis para quantificação de filopodia. Juntos, o protocolo apresentado fornece uma estrutura para análise precisa da dinâmica de proteínas em protrusões filopodiais usando software de análise de imagem.
Introduction
O controle espaço-temporal das proteínas reguladoras da actina está associado à dinâmica do filopodium 1 , 2 . O rastreamento da concentração de proteína resolvida espacialmente ao longo do comprimento todo filopodial através do tempo é, portanto, crucial para avançar a nossa compreensão dos mecanismos subjacentes à iniciação, alongamento, estabilização ou colapso dessas estruturas dinâmicas 3 , 4 . Ao contrário da análise de proteínas no citossolo, onde muitas mudanças de formas de células ocorrem em uma escala maior, filopodia são micro estruturas dinâmicas que constantemente se curvam 5 e se dobram, impedindo a análise usando uma abordagem simples, como uma varredura de linha.
Diferentes soluções de software para rastrear a forma filopodial estão disponíveis 6 , 7 , 8 , 9 . LikewIse, software para o rastreamento ratiométrico da dinâmica das proteínas dentro do corpo celular foi desenvolvido 10 , 11 . Para combinar o rastreamento automatizado da forma filopodial e da análise da proteína espaço-temporal, recentemente desenvolvemos um software de análise de imagem baseado no algoritmo convexo-casco 12 . Este novo método de análise, que é operado através de uma interface de usuário gráfica (GUI), combina pela primeira vez, a concentração relativa de proteína ao longo do comprimento filopodial e da velocidade de crescimento, permitindo assim a medição precisa da distribuição da proteína espaço-temporal independente do movimento desses Estruturas dinâmicas 12 .
A idéia por trás do software (o código fonte está disponível gratuitamente, veja abaixo) é que um dos vértices do casco convexo coincidirá com a ponta do filopodium ( Figura 1A ). Ao olhar para o quadro subsequente paraO vértice mais próximo do casco convexo, a dica móvel pode ser rastreada em todo o filme. Uma vez que a ponta é detectada em cada quadro, sua posição é usada para desenhar um eixo juntando a ponta com um ponto de referência na base do filopodium ( Figura 1B ). Finalmente, o uso de pontos nodais equidistantes, cujas posições são determinadas pelo pixel médio com intensidade máxima ao longo da linha ortogonal ao eixo, são usados para determinar uma espinha dorsal que segue a forma filopodial. Aproveitando esse backbone adaptativo, é gerado um kymograph para rastrear o crescimento filopodial e as concentrações de proteína para até três canais ao longo do comprimento filopodial ( Figura 1C ).
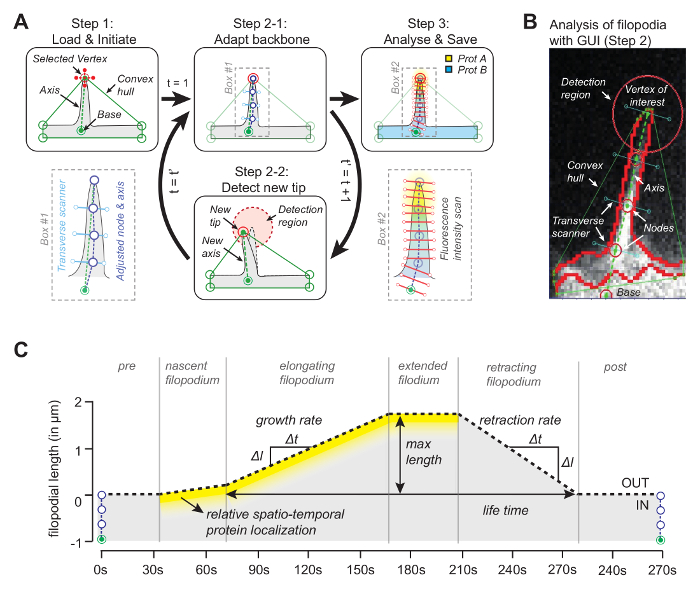
Figura 1: Princípio de Trabalho do Software de Análise de Imagem. ( A ) O algoritmo por tráso software. No Passo 1, o usuário especifica a referência (base) e o vértice (a ponta) do filopodium. Na Etapa 2-1, a espinha dorsal do filopodium é obtida usando o pixel médio com valor de intensidade máxima. Na Etapa 3, a espinha dorsal é usada para o perfil de intensidade da proteína espacial. Na Etapa 2-2, o software acompanha automaticamente a ponta no quadro subseqüente. Todo o procedimento itera. ( B ) Instantâneo do algoritmo com filopodium real que apresenta elementos importantes, como o casco convexo que está sendo usado para o rastreamento. ( C ) Visão geral dos parâmetros que podem ser medidos com o algoritmo. Esta figura foi modificada a partir da referência 12 . Clique aqui para ver uma versão maior dessa figura.
O software de análise de imagem é operado no Matlab (conhecido como software de programação) por meio de um uso gráficoR interface. Para maximizar a flexibilidade e a robustez para a configuração experimental particular, o usuário pode ajustar uma série de parâmetros de rastreamento ( por exemplo, ângulo de flexão permitido e movimento entre moldura) e também fazer algumas correções nos filmes ( por exemplo , corte, rotação, remoção de objetos indesejados) ( Figura 2A e Tabela 1) .
| GUI | Não. | Obrigatório | Descrição | Nome (em GUI) | ||||
| # 1 | 1a | Y | Carregando o arquivo .tiff empilhado que representa o corpo da célula (com caixa marcada) ou crie o corpo celular sobreposto a partir de canais | Corpo celular | ||||
| # 1 | 1b | Y | <td colspan="5"> Carregando arquivo de imagem empilhado correspondente à proteína 1Proteína 1 | |||||
| # 1 | 1c | Y | Carregando arquivo de imagem empilhado correspondente à proteína 2 | Proteína 2 | ||||
| # 1 | 1d | Y | Carregando arquivo de imagem empilhado correspondente à proteína 3 | Proteína 3 | ||||
| # 1 | 1e | N | Redefine tudo para arquivos de imagem empilhados pré-carregados | Restabelecer | ||||
| # 1 | 2a | Y | Barra de rolagem para determinar a moldura inicial para análise na janela GUI # 2 | N / D | ||||
| # 1 | 2b | Y | Barra de rolagem para determinar o quadro final para análise na janela GUI # 2 | N / D | ||||
| # 1 | 2c | Y | Barra de rolagem representando curdaQuadro nt | N / D | ||||
| # 1 | 2d | N | O valor cinza dos pixels abaixo do qual todos os pixels serão definidos como zero | N / D | ||||
| # 1 | 2e | N | O valor cinza dos pixels acima do qual todos os pixels serão definidos como valores máximos | N / D | ||||
| # 1 | 2f | N | Defina os valores de intensidade dos pixels especificados em <2e> & <2f> | Conjunto | ||||
| # 1 | 2g | N | Jogue o filme ajustado pela intensidade | Toque | ||||
| # 1 | 2h | N | Cortar imagem | Colheita | ||||
| # 1 | 2i | N | Girar imagem | Girar | ||||
| # 1 | 2j | N | Eliminar regiões na pilha inteira | Eliminar regiões | ||||
| # 1 | 3a | Y | Clique para abrir a 'Janela de análise' (janela GUI # 2) | Janela de rastreamento | ||||
| # 1 | 3b | Y | Digite o tamanho de um pixel em microns | Digite o tamanho do pixel | ||||
| # 2 | 4 | Y | Clique para gerar a imagem limite / borda do corpo celular sobreposto | fronteira | ||||
| # 2 | 5 | Y | Clique para selecionar a base e a ponta do filopodia | Referernce | ||||
| # 2 | 6a | Y | Digite o número de segmentos ou nós | Número de segmentos | ||||
| # 2 | 6b | Y | Digite o comprimento da varredura (perpendicular ao eixo) | Largura de digitalização | ||||
| # 2 | 6c | Y | Digite o comprimento acima do qual o filopodia começa a dobrar | Precise Meas after | ||||
| # 2 | 6d | Y | Digite o raio do círculo de detecção da ponta (ou seja, a área onde o vértice pode ser localizado no próximo quadro) | Radius of tip detection | ||||
| # 2 | 6e | Y | Insira o ângulo máximo que o filopodium pode dobrar do eixo vertical | Limiar de ângulo | ||||
| # 2 | 6f | N | Adicione pontos de referência para a base e a dica para esse quadro específico | Selecione referência | ||||
| # 2 | 6 g | N | Digite o comprimento acima do qual o filopodia começa a dobrar esse quadro específico | Precise Meas after | ||||
| # 2 | <Td> 6hN | Digite o raio do círculo de detecção para esse quadro específico | Radius of tip detection | |||||
| # 2 | 6i | N | Depois de inserir todos os parâmetros para o quadro específico, clique para armazenar os valores na memória e no arquivo para referência adicional | Adicionar | ||||
| # 2 | 6j | N | Clique para excluir os parâmetros manualmente configurados para essa moldura | Excluir | ||||
| # 2 | 6k | N | Clique para excluir todos os parâmetros armazenados manualmente usando o "painel de recursos opcionais" para todos os quadros | Restabelecer | ||||
| # 2 | 6l | N | Verifique antes de rastrear para armazenar todos os resultados de rastreamento na memória para futuras referências | Traço de história | ||||
| # 2 | 7 | Y | Clique para iniciar o rastreamento | Acompanhar e Analisar | ||||
| # 2 | 8a | N | Clique para iniciar o rastreamento da intensidade do canal protéico | Analisar Intensidades de Proteínas | ||||
| # 2 | 8b | N | Verifique para rastrear a intensidade do canal protéico ao longo do comprimento filopodial | Filopodias inteiras | ||||
| # 2 | 8c | N | Verifique o rastreamento da proteína de referência ou proteína A | ProteinA | ||||
| # 2 | 8d | N | Verifique o rastreamento da proteína B | ProteinB | ||||
| # 2 | 8e | N | Verifique o rastreamento da proteína C | ProteinC | ||||
| # 2 | 8f | N | Cheque para rastrear a intensidade média da proteína naE dica | Ponta principal | ||||
| # 2 | 8g | N | Digite o comprimento da dica | Comprimento da ponta | ||||
| # 2 | 8h | N | Digite a distância mínima da base acima da qual a ponta começa a formar | limite | ||||
| # 2 | 8i | N | Clique para salvar os resultados da análise de ponta líder para arquivar | Botão de apertar | ||||
| # 2 | 9a | N | Clique para iniciar a análise da proteína ratiométrica | Comparar | ||||
| # 2 | 9b | N | Verifique para comparar a proteína B com respeito a A | Log10 (B / A) | ||||
| # 2 | 9c | N | Verifique para comparar a proteína C em relação a A | Log10 (C / A) | ||||
| # 2 | <Td> 9dN | Verifique para comparar a proteína B com respeito a A na ponta | Log10 (B / A) | |||||
| # 2 | 9e | N | Verifique para comparar a proteína C com respeito a A na ponta | Log10 (C / A) | ||||
| # 2 | 10a | N | Escolha outro mapa de cores (padrão: Jetplot) | Mapa de cores | ||||
| # 2 | 10b | N | Edite o mapa de cores | Editar o mapa de cores | ||||
Tabela 1: Visão geral de todas as funções presentes na GUI Windows # 1 e # 2.
Uma vez que isso é realizado, o programa cria um casco convexo e rastreia automaticamente a dica ao longo do filme. Parâmetros extraídos do filme, como um kymograph ratiométrico, velocidade de crescimento e comprimento filopodial aÉ exibido e também armazenado na pasta de trabalho como imagens e como arquivos de dados. Outros parâmetros, tais como vida útil filopodial, taxa de crescimento e taxa de retração, podem então ser extraídos e analisados a partir dos arquivos de dados armazenados ( Figura 2B ).
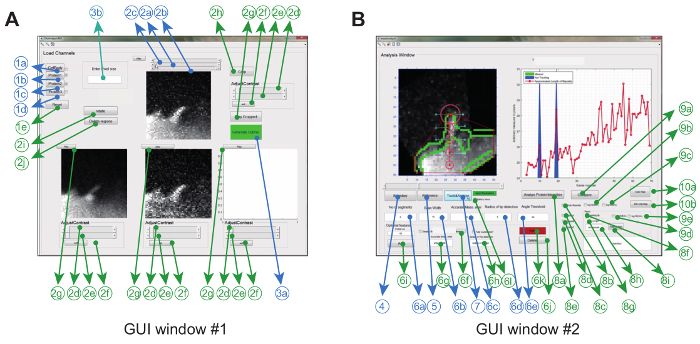
Figura 2: Interface gráfica do usuário para usar o software de análise de imagem. ( A ) GUI Janela # 1 é usada para carregar e processar imagens. O programa pode carregar até 3 canais de proteína, pelo que 2 canais são comparados em par. A janela vem com características obrigatórias (azuis) e opcionais (verde) para pré-processamento das imagens antes do rastreamento ( B ) GUI A Janela # 2 é usada para rastrear análises de proteínas filopodium e spatio-temporais e métricas. Novamente, os recursos opcionais estão marcados em verde. Esta figura foi modificadaDa referência 12 . Clique aqui para ver uma versão maior dessa figura.
Aqui, apresentamos um protocolo detalhado para preparação de amostras e manipulação de software. Começamos com instruções detalhadas sobre cultura de células e aquisição de filmes otimizados para análise de imagens. Esta seção sobre aquisição de dados é seguida por uma descrição detalhada para operar o software de análise de imagem. Ao longo do protocolo, apresentamos etapas críticas e recursos opcionais que devem ser considerados ao coletar e processar dados. Finalmente, analisamos filopodia de sistemas modelo diferentes com o software de análise de imagem, antes de fechar com uma comparação do software de análise de imagem descrito com outros programas disponíveis para quantificação filopodial e uma discussão sobre limitações e direção futura.
Protocol
Representative Results
Discussion
Aqui apresentamos um protocolo detalhado para rastrear a dinâmica do crescimento filopodial e a análise das concentrações relativas de proteína nessas estruturas dinâmicas através do algoritmo convexo-casco. Usando o software, até 3 canais podem ser comparados por pares em uma única execução, pelo que as concentrações relativas de dois canais ( ou seja, proteínas) são determinadas ao longo do ciclo de extensão / retração e armazenadas como arquivos de imagem e dados em pastas separadas. Além …
Declarações
The authors have nothing to disclose.
Acknowledgements
Os autores reconhecem o financiamento do DFG (EXC-1003 a MG).
Materials
| DMEM | Life Technologies | 31966-021 | |
| 10% Fetal bovine serum | Biochrom AG | L11-044 | |
| Lipofectamine 2000 | Life Technologies | 11668-027 | |
| 1% penicillin/streptomycin | Biochrom AG | 12212 | |
| Neurobasal Medium | Life Technologies | 21103-049 | |
| B27 | Life Technologies | 17504-044 | |
| HEPES (1M stock solution) | Life Technologies | 15630 | |
| Citrine-N1 | Addgene | 54593 | |
| Labtech | Thermo | 155411 | |
| Glutamax-I | Thermo | 35050-061 | |
| Hela | Leibniz Institute DSMZ | ACC-57 | |
| COS 7 | Leibniz Institute DSMZ | ACC-60 | |
| 3T3 cells | Leibniz Institute DSMZ | ACC-59 | |
| Microscope | Nicon Eclipse | ||
| Camera | Andor | DU888 Ultra | |
| Confocal Unit | Yokagawa | CSU-X1 | |
| Pyruvate | Gibco | 31966-021 |
Referências
- Dunaevsky, A., Tashiro, A., Majewska, A., Mason, C., Yuste, R. Developmental regulation of spine motility in the mammalian central nervous system. Proc Natl Acad Sci U S A. 96 (23), 13438-13443 (1999).
- Matus, A., Brinkhaus, H., Wagner, U. Actin dynamics in dendritic spines: a form of regulated plasticity at excitatory synapses. Hippocampus. 10 (5), 555-560 (2000).
- Galic, M., et al. Dynamic recruitment of the curvature-sensitive protein ArhGAP44 to nanoscale membrane deformations limits exploratory filopodia initiation in neurons. Elife. 3, e03116 (2014).
- Hotulainen, P., et al. Defining mechanisms of actin polymerization and depolymerization during dendritic spine morphogenesis. J Cell Biol. 185 (2), 323-339 (2009).
- Leijnse, N., Oddershede, L. B., Bendix, P. M. Helical buckling of actin inside filopodia generates traction. Proc Natl Acad Sci U S A. 112 (1), 136-141 (2015).
- Tsygankov, D., et al. CellGeo: a computational platform for the analysis of shape changes in cells with complex geometries. J Cell Biol. 204 (3), 443-460 (2014).
- Xiong, Y., et al. Automated characterization of cell shape changes during amoeboid motility by skeletonization. BMC Syst Biol. 4, 33 (2010).
- Styner, M., Gerig, G., Lieberman, J., Jones, D., Weinberger, D. Statistical shape analysis of neuroanatomical structures based on medial models. Med Image Anal. 7 (3), 207-220 (2003).
- Blum, H., Wathen-Dunn, W. A transformation for extracting new descriptors of shape. Models for the Perception of Speech and Visual Form: Proceedings of a Symposium. , 362-380 (1967).
- Barry, D. J., Durkin, C. H., Abella, J. V., Way, M. Open source software for quantification of cell migration, protrusions, and fluorescence intensities. J Cell Biol. 209 (1), 163-180 (2015).
- Machacek, M., et al. Coordination of Rho GTPase activities during cell protrusion. Nature. 461 (7260), 99-103 (2009).
- Saha, T., et al. Automated analysis of filopodial length and spatially resolved protein concentration via adaptive shape tracking. Mol Biol Cell. 27 (22), 3616-3626 (2016).
- Argiro, V., Bunge, M. B., Johnson, M. I. A quantitative study of growth cone filopodial extension. J Neurosci Res. 13 (1-2), 149-162 (1985).
- Mogilner, A., Rubinstein, B. The physics of filopodial protrusion. Biophys J. 89 (2), 782-795 (2005).
- Shaner, N. C., Steinbach, P. A., Tsien, R. Y. A guide to choosing fluorescent proteins. Nat Methods. 2 (12), 905-909 (2005).
- Abramoff, M. D., Magalhaes, P. J., Ram, S. J. Image Processing with ImageJ. Biophotonics International. 11, 36-42 (2004).
- Courtney, J., Woods, E., Scholz, D., Hall, W. W., Gautier, V. W. MATtrack: A MATLAB-Based Quantitative Image Analysis Platform for Investigating Real-Time Photo-Converted Fluorescent Signals in Live Cells. PLoS One. 10 (10), e0140209 (2015).
- Schell, M. J., Erneux, C., Irvine, R. F. Inositol 1,4,5-trisphosphate 3-kinase A associates with F-actin and dendritic spines via its N terminus. J Biol Chem. 276 (40), 37537-37546 (2001).
- Korobova, F., Svitkina, T. Molecular architecture of synaptic actin cytoskeleton in hippocampal neurons reveals a mechanism of dendritic spine morphogenesis. Mol Biol Cell. 21 (1), 165-176 (2010).
- Cheadle, L., Biederer, T. The novel synaptogenic protein Farp1 links postsynaptic cytoskeletal dynamics and transsynaptic organization. J Cell Biol. 199 (6), 985-1001 (2012).
- Tarnok, K., et al. A new tool for the quantitative analysis of dendritic filopodial motility. Cytometry A. 87 (1), 89-96 (2015).
- Hendricusdottir, R., Bergmann, J. H. F-dynamics: automated quantification of dendrite filopodia dynamics in living neurons. J Neurosci Methods. 236, 148-156 (2014).
- Fanti, Z., Martinez-Perez, M. E., De-Miguel, F. F. NeuronGrowth, a software for automatic quantification of neurite and filopodial dynamics from time-lapse sequences of digital images. Dev Neurobiol. 71 (10), 870-881 (2011).
- Costantino, S., et al. Semi-automated quantification of filopodial dynamics. J Neurosci Methods. 171 (1), 165-173 (2008).
- Nilufar, S., Morrow, A. A., Lee, J. M., Perkins, T. J. FiloDetect: automatic detection of filopodia from fluorescence microscopy images. BMC Syst Biol. 7, 66 (2013).

