Dinamik Hücresel Protrüzyonlarda Protein Konsantrasyonunun Yazılım Destekli Takibi için Grafiksel Bir Kullanıcı Arayüzü
Summary
Dinamik hücresel çıkıntıların uzunluğu boyunca göreli protein yoğunluğunun yarı otomatik olarak izlenmesi için bir yazılım çözümü sunmaktayız.
Abstract
Filopodia, göç ve hücre-hücre iletişimi ile ilişkili dinamik, parmak benzeri hücresel çıkıntılardır. Filopodial başlatma, uzama ve daha sonra stabilizasyon veya geri çekmenin altında yatan karmaşık sinyal mekanizmalarını daha iyi anlamak için, bu dinamik yapılarda uzay-zamansal protein aktivitesinin belirlenmesi çok önemlidir. Filopodiya'daki protein fonksiyonunu analiz etmek için kısa bir süre önce filopodial şekil değişikliklerine uyum sağlayan yarı otomatikleştirilmiş bir izleme algoritması geliştirdik ve böylece tüm filopodial uzunluk boyunca çıkıntı dinamiklerinin ve göreceli protein konsantrasyonunun paralel analizine izin verdik. Burada, optimize hücre işleme, görüntü toplama ve yazılım analizi için ayrıntılı bir adım adım protokol sunuyoruz. Ayrıca görüntü analizi ve veri sunumu sırasında isteğe bağlı özelliklerin kullanımı ile ilgili talimatların yanı sıra yol boyunca tüm kritik adımlar için giderme yönergeleri sağlıyoruz. Son olarak, aynı zamanda dFilopodia niceleme için hazır bulunan diğer programlarla birlikte görüntü analiz yazılımını hazırlar. Sunulan protokol, birlikte görüntü analizi yazılımı kullanarak filopodial çıkıntılar protein dinamikleri doğru analiz için bir çerçeve sağlar.
Introduction
Aktin düzenleyici proteinlerin spatio-temporal kontrolü filopodium dinamikler 1 , 2 ile ilişkilidir. Zamanla tüm filopodial uzunluk boyunca uzaysal olarak çözülen protein konsantrasyonunun izlenmesi, bu dinamik yapıların başlatılması, uzatılması, stabilizasyonu veya çökmesinin altında yatan mekanizmaları anlamak için çok önemlidir 3 , 4 . Birçok hücre şekli değişikliğinin daha büyük bir ölçekte gerçekleştiği sitoplazmada protein analizinin aksine, filopodiya, sürekli olarak 5 virajını kavrayan ve virüs oluşturan dinamik mikro yapılar olup, bu nedenle çizgi tarama gibi basit bir yaklaşımla analiz yapılmamaktadır.
Filopodial şekli izlemek için farklı yazılım özümleri mevcuttur 6 , 7 , 8 , 9 . LikewHücre gövdesi içerisindeki protein dinamiklerinin oraniyometrik izlenmesi için yazılım geliştirilmiştir. 10 , 11 . Filopodial şekil ve uzay-zamanlı protein analizinin otomatik izlenmesini birleştirmek için son zamanlarda konveks-gövde algoritmasına 12 dayalı bir görüntü analiz yazılımı geliştirdik. Bir grafiksel kullanıcı arabirimi (GUI) aracılığıyla çalıştırılan bu yeni analiz yöntemi, filopodial uzunluk ve büyüme hızı boyunca göreli protein konsantrasyonunu ilk kez birleştirir ve böylece uzay-temporal protein dağılımının bu ölçümlerin hareketlerinden bağımsız olarak doğru ölçümünü sağlar Dinamik yapılar 12 .
Yazılımın arkasındaki fikir (kaynak kodu serbestçe bulunur, aşağıya bakınız), konveks gövdesinin köşelerinden biri filopodyumun ucuyla örtüşecektir ( Şekil 1A ). Sonraki çerçeveye bakarak,Dışbükey gövdenin en yakın tepe noktası, hareketli uç tüm film boyunca izlenebilir. Ucu her karede algılandıktan sonra, konumu, filopodyum tabanında bir referans noktası ile ucu birleştirerek bir eksen çizmek için kullanılır ( Şekil 1B ). Son olarak, konumları eksene dik olan çizgide maksimum yoğunluk ile medyan piksel ile belirlenen eşit uzaklıktaki düğüm noktaları kullanılarak, filopodial şekli takip eden omurga belirlenir. Bu adaptif omurgadan yararlanarak, filopodial uzunluk boyunca üç kanala kadar filopodial büyüme ve protein konsantrasyonlarını izlemek için bir kymograph oluşturulur ( Şekil 1C ).
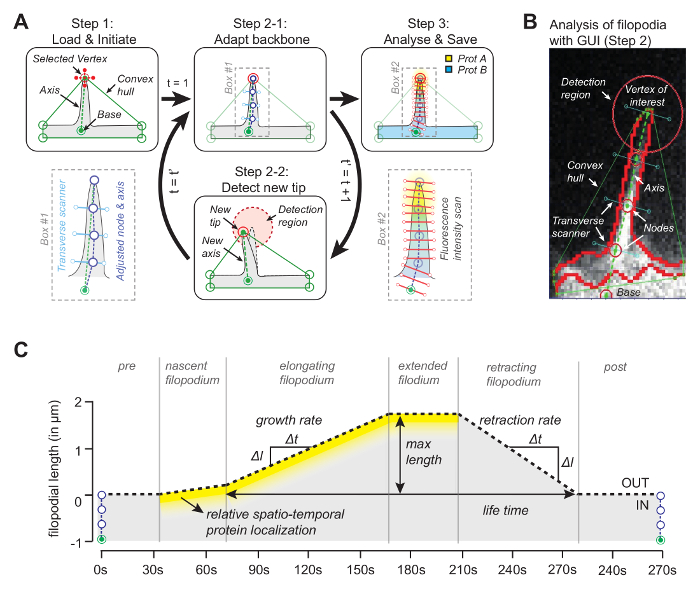
Şekil 1: Görüntü Analiz Yazılımının Çalışma Prensibi. ( A ) Arkasındaki algoritmayazılım. Adım1'de kullanıcı, filopodyumun referansını (tabanı) ve tepeyi (ucu) belirtir. Adım 2-1'de, filopodyumun omurgası, maksimum yoğunluk değerine sahip medyan piksel kullanılarak elde edilir. Adım 3'te omurga mekansal protein yoğunluğu profili için kullanılır. Adım 2-2'de, yazılım otomatik olarak sonraki çerçevedeki ucu izler. Bütün prosedür tekrarlanır. ( B ) İzlem için kullanılan konveks gövde gibi önemli elementleri sunan gerçek filopodyumlu algoritmanın anlık görüntüsü. ( C ) Algoritma ile ölçülebilen parametrelere genel bakış. Bu rakam referans 12'den değiştirildi. Bu rakamın daha büyük bir versiyonunu görmek için lütfen tıklayınız.
Görüntü analiz yazılımı, grafiksel bir kullanım yoluyla Matlab'da (programlama yazılımı olarak anılır) çalıştırılırR arabirimi. Belli deneysel ayarlar için esneklik ve dayanıklılığı en üst düzeye çıkarmak için kullanıcı bir dizi izleme parametresini ayarlayabilir ( örneğin, izin verilen eğme açısı ve çerçeve içi hareketi) ve filmlerde bazı düzeltmeler yapabilir ( örneğin, kırpma, döndürme, istenmeyen nesnelerin giderilmesi) ( Şekil 2A ve Tablo 1) .
| GUI | Yok hayır. | Zorunlu | Açıklama | İsim (GUI'de) | ||||
| 1. | 1 A | Y | Hücre gövdesini (kutu işaretli olarak) gösteren yığınlanmış .tiff dosyasını yüklemek veya kanallardan üst üste yerleştirilen hücre gövdesi oluşturmak | Vücut hücresi | ||||
| 1. | 1b | Y | <td colspan="5"> Proteine 1 karşılık gelen istiflenmiş resim dosyası yükleniyorProtein 1 | |||||
| 1. | 1c | Y | Protein 2'ye karşılık gelen yığılmış görüntü dosyasını yükleme | Protein 2 | ||||
| 1. | 1d | Y | Protein 3'e karşılık gelen yığınlanmış görüntü dosyasını yükleme | Protein 3 | ||||
| 1. | 1e | N- | Önceden yüklenmiş yığılmış görüntü dosyalarına her şeyi sıfırlar | Reset | ||||
| 1. | 2a | Y | GUI penceresinde # 2 analiz için ilk kareyi belirlemek için kaydırma çubuğu | NA | ||||
| 1. | 2b | Y | GUI penceresinde # 2 analiz için son kareyi belirlemek için kaydırma çubuğu | NA | ||||
| 1. | 2c | Y | Curre'yi temsil eden kaydırma çubuğuNt çerçevesi | NA | ||||
| 1. | 2d | N- | Tüm piksellerin altında sıfıra ayarlanacağı piksellerin gri değeri | NA | ||||
| 1. | 2e | N- | Yukarıdaki tüm piksellerin maksimum değerlere ayarlanacağı piksellerin gri değeri | NA | ||||
| 1. | 2f | N- | <2e> & <2f> ile belirtilen piksellerin yoğunluk değerlerini ayarlayın. | Set | ||||
| 1. | 2 g | N- | Yoğunluğu ayarlanmış filmi oynat | Oyun | ||||
| 1. | 2s | N- | Görüntüyü kırp | ekin | ||||
| 1. | 2i | N- | Resmi Döndür | Döndürme | ||||
| 1. | 2j | N- | Tüm yığında bölgeleri sil | Bölgeleri sil | ||||
| 1. | 3 A | Y | 'Analiz Penceresi'ni açmak için tıklayın (GUI penceresi # 2) | İzleme Penceresi | ||||
| 1. | 3b | Y | Mikron cinsinden bir piksel boyutunu girin | Piksel boyutunu girin | ||||
| 2. | 4 | Y | Üst üste yerleştirilen hücre gövdesinin sınır / kenar görüntüsünü oluşturmak için tıklayın | Sınır | ||||
| 2. | 5 | Y | Filopodiyanın taban ve ucunu seçmek için tıklayın | Referernce | ||||
| 2. | 6a | Y | Segmentlerin veya düğümlerin sayısını girin | Segment sayısı | ||||
| 2. | 6b | Y | Tarama uzunluğunu girin (eksene dik olarak) | Tarama Genişliği | ||||
| 2. | 6c | Y | Filopodinin yukarıya doğru bükülmeye başlama uzunluğunu girin | Sonra Doğru Ölçümler | ||||
| 2. | 6d | Y | İpucu algılama çemberinin yarıçapını girin (yani, vertex'in bir sonraki çerçevede lokalize edilebileceği alan) | İpucu algılama yarıçapı | ||||
| 2. | 6e | Y | Filopodium'un dikey eksenden kıvrılabileceği maksimum açıyı girin | Açı Eşiği | ||||
| 2. | 6f | N- | Belirli çerçeve için taban ve uç için referans noktaları ekleyin | Referans seç | ||||
| 2. | 6g | N- | Belli bir çerçeve için filopodinin bükülmeye başlandığı uzunluğu girin | Sonra Doğru Ölçümler | ||||
| 2. | <td> 6hN- | Belirli çerçeve için algılama çemberinin yarıçapını girin | İpucu algılama yarıçapı | |||||
| 2. | 6i | N- | Belirli bir çerçeve için tüm parametreleri girdikten sonra, daha sonraki referanslar için bellek ve dosyalara değerleri depolamak için tıklayın. | Eklemek | ||||
| 2. | 6j'nin | N- | Bu karenin elle ayar parametrelerini silmek için tıklayın | silmek | ||||
| 2. | 6k | N- | Tüm kareler için 'isteğe bağlı özellikler paneli' kullanılarak manuel olarak saklanan tüm parametreleri silmek için tıklayın | Reset | ||||
| 2. | 6l | N- | Gelecekteki referans için tüm izleme sonuçlarını belleğe kaydetmek için izleme işleminden önce kontrol edin | Geçmiş izi | ||||
| 2. | 7 | Y | İzlemeyi başlatmak için tıklayın | Takip ve Analiz | ||||
| 2. | 8a | N- | Protein kanalı yoğunluğunu izlemeye başlamak için tıklayın | Protein yoğunluklarını analiz edin | ||||
| 2. | 8b | N- | Filopodial uzunluk boyunca protein kanal yoğunluğunu izlemek için kontrol edin | Bütün filopodi | ||||
| 2. | 8c | N- | Referans proteinini veya protein A'yı izlemek için kontrol edin | Protein A | ||||
| 2. | 8d | N- | B proteinini izlemek için kontrol edin | ProteinB | ||||
| 2. | 8e | N- | Proteini C izlemek için kontrol edin | ProteinC | ||||
| 2. | 8f | N- | Ortalama protein yoğunluğunu izlemek için kontrol edin.E uç | Öne çıkan ipucu | ||||
| 2. | 8g | N- | İpucunun uzunluğunu girin | İpucu Uzunluğu | ||||
| 2. | 8h | N- | Ucun üstünde şekillendirme başladığı minimum mesafeyi girin | eşik | ||||
| 2. | 8i | N- | Öncü ipucu analiz sonuçlarını dosyaya kaydetmek için tıklayın. | Butona basınız | ||||
| 2. | 9a | N- | Ratiyometrik protein analizi başlatmak için tıklayın | Karşılaştırmak | ||||
| 2. | 9b | N- | A proteinine B proteinini karşılaştırmak için giriş yapın. | log 10 (B / A) | ||||
| 2. | 9c | N- | Protein C'yi A'ya göre karşılaştırmak için kontrol edin | log 10 (C / A) | ||||
| 2. | <td> 9dN- | Protein B'yi uçtaki A'ya göre karşılaştırmak için kontrol edin | log 10 (B / A) | |||||
| 2. | 9e | N- | Protein C'yi uçtaki A'ya göre karşılaştırmak için kontrol edin | log 10 (C / A) | ||||
| 2. | 10a | N- | Diğer renk haritasını seçin (varsayılan: Jetplot) | Renk Haritası | ||||
| 2. | 10b | N- | Renk haritasını düzenle | Renk haritasını düzenle | ||||
Tablo 1: GUI'de Bulunan Tüm İşlevlere Genel Bakış Windows # 1 ve # 2.
Bu gerçekleştirildiğinde, program bir konveks gövde oluşturur ve film boyunca ucu otomatik olarak izler. Filmden çıkarılan parametreler, örneğin bir sıçanometrik kymograph, büyüme hızı ve filopodial uzunluk aYeniden görüntülenir ve iş klasörüne görüntüler ve veri dosyaları olarak saklanır. Filopodial yaşam süresi, büyüme hızı ve geri çekme oranı gibi diğer parametreler daha sonra çıkarılabilir ve depolanmış veri dosyalarından daha fazla analiz edilebilir ( Şekil 2B ).
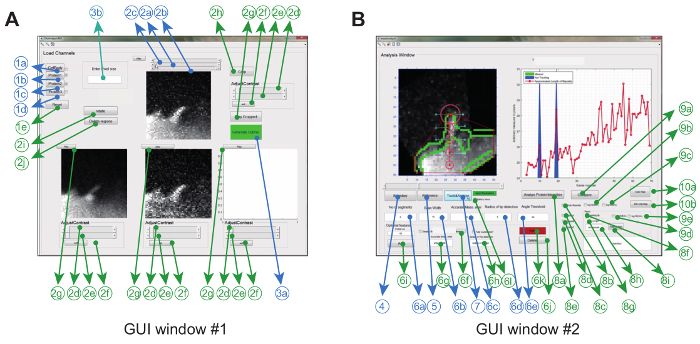
Şekil 2: Görüntü Analiz Yazılımının Kullanımı için Grafik Kullanıcı Arayüzü. ( A ) GUI Penceresi # 1, resim yüklemek ve işlemek için kullanılır. Program 3 kanala kadar yükleyebilir, böylece 2 kanal karşılaştırılır. Pencere, izlemeden önce görüntülerin ön işleme tabi tutulması için zorunlu (mavi) ve isteğe bağlı özellikler (yeşil) ile birlikte gelir ( B ) GUI Pencere # 2, filopodyumun yanı sıra zamansal ve oran-metrik protein analizi izlemek için kullanılır. Yine, isteğe bağlı özellikler yeşil olarak işaretlenmiştir. Bu rakam modifi olduKaynakça 12 . Bu rakamın daha büyük bir versiyonunu görmek için lütfen tıklayınız.
Burada, örnek hazırlama ve yazılım kullanımı için ayrıntılı bir protokol sunmaktayız. Hücrelerin kültürlenmesi ve görüntü analizi için optimize edilmiş filmlerin edinilmesi ile ilgili ayrıntılı talimatlarla başlayacağız. Veri edinimi ile ilgili bu bölüm, görüntü analiz yazılımını çalıştırmak için ayrıntılı bir açıklama izliyor. Protokol boyunca, verileri toplamak ve işlemek için dikkate alınması gereken kritik adımları ve isteğe bağlı özellikleri tanıtmaktayız. Son olarak, açıklanan görüntü analiz yazılımının filopodial niceleme için mevcut olan diğer programlarla karşılaştırılması ve sınırlamalar ve geleceğe yönelik bir tartışma ile kapanmadan önce, filopodiyi görüntü analiz yazılımı ile farklı model sistemlerinden analiz ediyoruz.
Protocol
Representative Results
Discussion
Burada, filopodial büyüme dinamikleri ve konveks gövde algoritması yoluyla bu dinamik yapılardaki bağıl protein konsantrasyonlarının izlenmesi için ayrıntılı bir protokol sunuyoruz. Yazılımı kullanarak, iki kanalın ( yani proteinler) göreli konsantrasyonlarının uzatma / geri çekme çevrimi boyunca belirlendiği ve ayrı klasörler halinde görüntü ve veri dosyaları olarak depolandığı tek bir programda üç kanala tek tek karşılaştırılabilir. Yazılım, rutin işlemlere ek olarak,…
Declarações
The authors have nothing to disclose.
Acknowledgements
Yazarlar, DFG'den (MG için EXC-1003) finansman sağlamışlardır.
Materials
| DMEM | Life Technologies | 31966-021 | |
| 10% Fetal bovine serum | Biochrom AG | L11-044 | |
| Lipofectamine 2000 | Life Technologies | 11668-027 | |
| 1% penicillin/streptomycin | Biochrom AG | 12212 | |
| Neurobasal Medium | Life Technologies | 21103-049 | |
| B27 | Life Technologies | 17504-044 | |
| HEPES (1M stock solution) | Life Technologies | 15630 | |
| Citrine-N1 | Addgene | 54593 | |
| Labtech | Thermo | 155411 | |
| Glutamax-I | Thermo | 35050-061 | |
| Hela | Leibniz Institute DSMZ | ACC-57 | |
| COS 7 | Leibniz Institute DSMZ | ACC-60 | |
| 3T3 cells | Leibniz Institute DSMZ | ACC-59 | |
| Microscope | Nicon Eclipse | ||
| Camera | Andor | DU888 Ultra | |
| Confocal Unit | Yokagawa | CSU-X1 | |
| Pyruvate | Gibco | 31966-021 |
Referências
- Dunaevsky, A., Tashiro, A., Majewska, A., Mason, C., Yuste, R. Developmental regulation of spine motility in the mammalian central nervous system. Proc Natl Acad Sci U S A. 96 (23), 13438-13443 (1999).
- Matus, A., Brinkhaus, H., Wagner, U. Actin dynamics in dendritic spines: a form of regulated plasticity at excitatory synapses. Hippocampus. 10 (5), 555-560 (2000).
- Galic, M., et al. Dynamic recruitment of the curvature-sensitive protein ArhGAP44 to nanoscale membrane deformations limits exploratory filopodia initiation in neurons. Elife. 3, e03116 (2014).
- Hotulainen, P., et al. Defining mechanisms of actin polymerization and depolymerization during dendritic spine morphogenesis. J Cell Biol. 185 (2), 323-339 (2009).
- Leijnse, N., Oddershede, L. B., Bendix, P. M. Helical buckling of actin inside filopodia generates traction. Proc Natl Acad Sci U S A. 112 (1), 136-141 (2015).
- Tsygankov, D., et al. CellGeo: a computational platform for the analysis of shape changes in cells with complex geometries. J Cell Biol. 204 (3), 443-460 (2014).
- Xiong, Y., et al. Automated characterization of cell shape changes during amoeboid motility by skeletonization. BMC Syst Biol. 4, 33 (2010).
- Styner, M., Gerig, G., Lieberman, J., Jones, D., Weinberger, D. Statistical shape analysis of neuroanatomical structures based on medial models. Med Image Anal. 7 (3), 207-220 (2003).
- Blum, H., Wathen-Dunn, W. A transformation for extracting new descriptors of shape. Models for the Perception of Speech and Visual Form: Proceedings of a Symposium. , 362-380 (1967).
- Barry, D. J., Durkin, C. H., Abella, J. V., Way, M. Open source software for quantification of cell migration, protrusions, and fluorescence intensities. J Cell Biol. 209 (1), 163-180 (2015).
- Machacek, M., et al. Coordination of Rho GTPase activities during cell protrusion. Nature. 461 (7260), 99-103 (2009).
- Saha, T., et al. Automated analysis of filopodial length and spatially resolved protein concentration via adaptive shape tracking. Mol Biol Cell. 27 (22), 3616-3626 (2016).
- Argiro, V., Bunge, M. B., Johnson, M. I. A quantitative study of growth cone filopodial extension. J Neurosci Res. 13 (1-2), 149-162 (1985).
- Mogilner, A., Rubinstein, B. The physics of filopodial protrusion. Biophys J. 89 (2), 782-795 (2005).
- Shaner, N. C., Steinbach, P. A., Tsien, R. Y. A guide to choosing fluorescent proteins. Nat Methods. 2 (12), 905-909 (2005).
- Abramoff, M. D., Magalhaes, P. J., Ram, S. J. Image Processing with ImageJ. Biophotonics International. 11, 36-42 (2004).
- Courtney, J., Woods, E., Scholz, D., Hall, W. W., Gautier, V. W. MATtrack: A MATLAB-Based Quantitative Image Analysis Platform for Investigating Real-Time Photo-Converted Fluorescent Signals in Live Cells. PLoS One. 10 (10), e0140209 (2015).
- Schell, M. J., Erneux, C., Irvine, R. F. Inositol 1,4,5-trisphosphate 3-kinase A associates with F-actin and dendritic spines via its N terminus. J Biol Chem. 276 (40), 37537-37546 (2001).
- Korobova, F., Svitkina, T. Molecular architecture of synaptic actin cytoskeleton in hippocampal neurons reveals a mechanism of dendritic spine morphogenesis. Mol Biol Cell. 21 (1), 165-176 (2010).
- Cheadle, L., Biederer, T. The novel synaptogenic protein Farp1 links postsynaptic cytoskeletal dynamics and transsynaptic organization. J Cell Biol. 199 (6), 985-1001 (2012).
- Tarnok, K., et al. A new tool for the quantitative analysis of dendritic filopodial motility. Cytometry A. 87 (1), 89-96 (2015).
- Hendricusdottir, R., Bergmann, J. H. F-dynamics: automated quantification of dendrite filopodia dynamics in living neurons. J Neurosci Methods. 236, 148-156 (2014).
- Fanti, Z., Martinez-Perez, M. E., De-Miguel, F. F. NeuronGrowth, a software for automatic quantification of neurite and filopodial dynamics from time-lapse sequences of digital images. Dev Neurobiol. 71 (10), 870-881 (2011).
- Costantino, S., et al. Semi-automated quantification of filopodial dynamics. J Neurosci Methods. 171 (1), 165-173 (2008).
- Nilufar, S., Morrow, A. A., Lee, J. M., Perkins, T. J. FiloDetect: automatic detection of filopodia from fluorescence microscopy images. BMC Syst Biol. 7, 66 (2013).

