植物/微生物の分子相互作用およびシグナル伝達の同時および系統的分析のための水耕栽培共生システム
Summary
記載された水耕共培養システムは、金属メッシュスクリーンを有する完全な植物を支持し、それらを細菌と共培養する。植物組織、細菌、および分泌された分子は、下流の分析のために別々に採取することができ、同時に植物宿主および相互作用する微生物または微生物の両方の分子応答を調べることができる。
Abstract
自然な植物 – 微生物相互作用を模倣する実験デザインは、複雑な植物 – 微生物シグナル伝達プロセスを描写するために非常に重要である。 シロイヌナズナ – アグロバクテリウム・ツメファシエンス 細菌の病原性および植物相互作用を研究するための優れたモデルシステムを提供します。植物 – アグロバクテリウム相互作用の以前の研究は、植物細胞浮遊培養、植物の人工的創傷、または合成化学物質による微生物毒性因子または植物防御の人為的誘発に大きく依存していた。しかし、これらの方法は、植物および微生物が空間的および時間的な仕方で認識し応答する、植物の天然シグナル伝達とは異なる。この研究は、インタクトな植物が金属メッシュスクリーンによって支持され、 アグロバクテリウムと共培養された水耕共培養系を提示する。この共存系において、micrを誘導する合成植物ホルモンまたは化学物質は存在しない陰茎毒性または植物防御が補われる。水耕性共培養系は、植物の微生物との自然な相互作用および植物のシグナル伝達ホメオスタシスに非常に類似している。植物根は、 アグロバクテリウムを含む培地から分離することができ、植物宿主および相互作用する微生物の両方のシグナル伝達および応答を同時にかつ系統的に調べることができる。任意の時点/間隔で、植物組織または細菌を様々な「オミックス」分析のために別々に収穫することができ、このシステムの効力および効力を実証する。水耕共培養系は、1)多様な植物微生物系の相反するシグナル伝達、2)植物宿主と複数の微生物種( すなわち微生物のコンソーシアムまたは微生物)との間のシグナル伝達、3)栄養素および化学物質の関与方法植物微生物シグナル伝達における微生物、および4)微生物が植物宿主とどのように相互作用し、生物的Oに対する植物耐性に寄与するかr非生物的ストレス。
Introduction
植物関連微生物は、生物地球化学サイクル、バイオレメディエーション、気候変動の緩和、植物の成長と健康、生物的および非生物的ストレスに対する植物耐性において重要な役割を果たす。微生物は直接植物細胞壁の接触を介して間接的に化学分泌を介して1、2、3シグナリングの両方の植物と相互作用します。固着生物として、植物は病原体による感染に抵抗する直接的および間接的メカニズムを開発してきた。間接的な防御は、二次植物代謝物の生産と病原体4,5の侵入に対する拮抗生物の魅力を含むのに対し、直接防御は、構造的な防御および防御タンパク質の発現を含みます。植物由来の滲出液、分泌物、粘液、ムシゲル、および溶解物は、根圏の物理化学的特性を変化させて誘引または反発する宿主に向かって微生物6 。根分泌物の化学組成は種特異的であり、それによって、そのような化合物を認識することができる特定の微生物が根圏6で繁殖することを可能にする選択的フィルターとして役立つ。したがって、適合性の微生物種は、植物宿主1の有益性または有害性のいずれかに、それらの関連性を活性化および強化するように刺激され得る。
微生物および化学的暴露の大部分は、ルート構造と土壌エアインタフェース2、6、7、8で起こるからである。根圏における理解植物微生物相互作用は、植物生産性と生態系の機能を強化するための鍵でありますしかしながら、地下の植物 – 微生物相互作用および相反応答の試験は、その興味深いことに複雑かつ動的な性質、および厳密に制御可能な増殖条件下での天然根構造および植物形態を有する適切な実験モデルの欠如を含む。最も頻繁に研究植物病原の一つとして、 アグロバクテリウムは桜、リンゴ、ナシ、ブドウなどの農園芸重要性を持つ植物の広い範囲に感染し、9ローズ 。 アグロバクテリウムは理解植物-病原体相互作用のための重要なモデル生物であり、植物の形質転換および植物工学10、11、12、13、14における強力なツールです。
分子は、 アグロバクテリウムの相互作用が十分に数十年にわたって研究されている、およびアグロバクテリウム病原性の現在の理解は9広範であるプラント-F "> 11、15、16。 アグロバクテリウムの病原性は、主に17を感知する、植物由来の信号を感知その毒性プログラムおよび細胞間通信の細かい調節をもたらす、いわゆる定足数のその進化能力に起因する。ザアグロバクテリウムの病原性プログラムは根圏で利用可能ないくつかの信号によって調節され、2成分系、は、chvg / Iシステムとビラ/ Gシステムの2つのセットを含むている。根圏における酸性条件は、 は、chvg / I、 ビラ/ Gの転写を活性化します、およびvirE0 、 virE1 、 virH1 、 virH2 、およびVI型分泌系(T6SS) 18の遺伝子を含むアグロバクテリウムの病原性に関与するいくつかの他の遺伝子が含まれる。アセトシリンゴン(4'-ヒドロキシ-3 '、5 ' – ジメトキシアセトフェノン)、Vリン酸化シグナル伝達機構によるirA / G二成分系19 。その後、VirA / Gはvirレギュロン全体を活性化し、その結果、腫瘍誘導性(Ti)プラスミドからトランスファーDNA(T-DNA)と呼ばれる約20kbのバクテリアDNA断片を植物核16に移入して組み込む。 T-DNAは、植物ホルモンインドール-3-酢酸(IAA)( iaaMおよびiaaH )およびサイトカイニン( ipt )の合成に関与する遺伝子を保有し、一旦植物細胞で発現すると、これらの植物ホルモンが大量に産生される。これは、植物9、11、20のために慢性および復活問題であるクラウンゴール病として知られる異常な組織増殖および植物腫瘍発達、もたらします。 IAAは、 アグロバクテリウムビルレンスを抑制するために、またはアグロバクテリウムを減少させるために、サリチル酸およびγ-アミノ酪酸と一緒に作用する m個のクオラムセンシング(QS)17、21、22。この抑制に対抗するために、T-DNAはまた、 アグロバクテリウムの病原性を促進するために、 アグロバクテリウムクオラムセンシングを活性化し、病原体22,23のための栄養源となるオピンの生合成のための遺伝子を運びます。
アグロバクテリウム –プラント相互作用の全体的な深い理解と、植物宿主への得られたT-DNA転移にもかかわらず、相互作用の初期段階での複雑なシグナル伝達事象はあまり理解されていない。これは、部分的に、 アグロバクテリウム –植物シグナル伝達を調べるための従来のアプローチの限界に起因する。植物細胞懸濁液培養および人工部位特異的創傷は、分子植物 – 微生物相互作用を研究するために一般的に使用されている24 、。EF "> 26、27はしかし、細胞懸濁液は、一般的な植物の形態に欠け、特に、植物の懸濁細胞は、微生物の走化性および病原性28、29を活性化するために非常に重要なルート構造と根の滲出液を、持っていない植物の形態の維持。ルート構造が直接感染した植物組織30、31に誘導される植物防御関連遺伝子の検出結果として、サイト固有の感染を容易に人為的に負傷植物によって対処されてきた。しかし、人工的な創傷は、自然の中で病原体感染と大きく異なっています特に、創傷がジャスモン酸(JA)の蓄積をもたらし、天然植物のシグナル伝達と防御を全身的に妨害する26。また、合成化学物質は、人工的に植物宿主応答を誘導するために使用される26 。または病原体の病原性である。 植物体中の濃度を反映するような化学物質の補充が可能であるが、そのような補充は徐々に微生物28、32によって検知された走化性勾配を生成取り囲む根圏へ根滲出液の拡散を考慮していません。植物 – 微生物相互作用を研究する従来のアプローチの限界を考えると、得られたデータの精度と深さは妨げられ、制限され、従来のアプローチから生成された知識は植物に直接翻訳されない可能性がある。植物 – アグロバクテリウムシグナリングの多くの局面は、特に、疾患の症状がまだ発症していない初期の相互作用の段階では、完全には理解されていない。
従来のアプローチの限界を修正するために、この研究は、安価で、しっかりと制御可能で、柔軟な水耕性のc研究者が分子植物 – 微生物相互作用の初期段階で複雑なシグナル伝達経路と応答経路に深い洞察を得ることを可能にする。水耕栽培は広く植物栄養素、根の滲出液、成長条件、および植物33、34上の金属毒性の影響を研究するために使用されてきました。水耕栽培モデルには、小さな空間的要件、様々な植物組織の接近可能性、栄養素/環境条件の厳密な制御、害虫/病害の制御などのいくつかの利点があります。水耕栽培システムはまた、典型的には2〜3週間後に成長を制限する寒天/フィトアガルめっき技術と比較して、植物の生育を制限するものではない。重要なのは、全体のプラント構造物の維持管理は、微生物の走化性および病原性の誘導8、29のために必要な自然なルート分泌を促進します。システムの説明ここでのベッドは選択肢33、34よりも簡単かつ少ない労働集約的です。これは、より少ない部品を使用し、標準はさみ以外の工具を必要としません。それは、植物生長の強力な支持体としての金属メッシュ(ナイロン33とは対照的に)と、微生物増殖を助けるための振盪による無菌条件下での単純な通気方法を使用する。さらに、このシステムは、根の幅を制限することなく多様な植物種に適応する、植物成長をサポートするために様々なサイズの金属メッシュを使用することができる。
ここに提示されている水耕共培養系では、接種された細菌の増殖を支える有機化合物を植物の根が分泌する無菌水耕システムで植物を栽培する。この共培養システムでは、植物ホルモン、防御エリシター、または病原性誘導化学物質などの人工化学物質は補充されず、自然細胞を反映する植物 – 微生物相互作用中のホメオスタシスシグナル。この水耕共培養システムでは、同時に遺伝子、アグロバクテリウムによる感染時シロイヌナズナ COL-0根組織における発現、ならびにシロイヌナズナとの共培養時にアグロバクテリウム遺伝子の活性化を決定することが可能でした。このシステムは、 アグロバクテリウム ( Agrobacterium )との同時培養(感染)の際に、植物根へのアグロバクテリウムの付着および植物根の分泌のプロファイルを研究するのに適していることがさらに実証された( 図1 )。
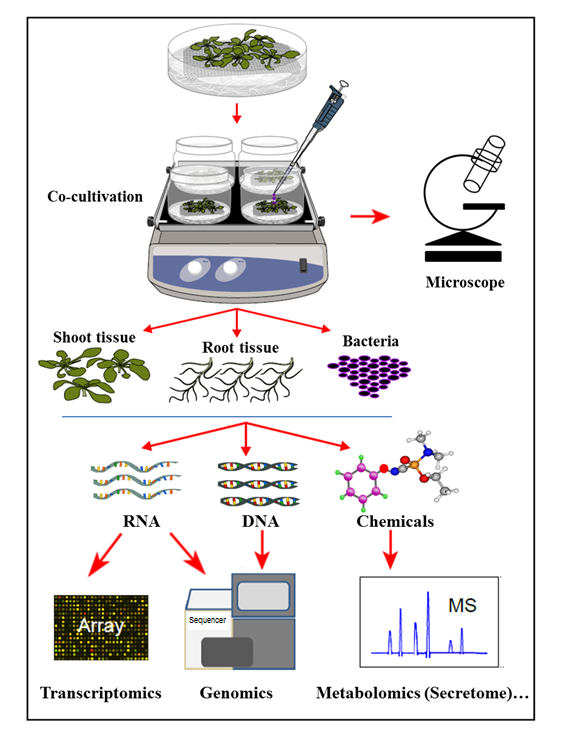
図1:サンプル分析を用いた水耕共培養系の概要。植物をメッシュの上に成長させる(メッシュの上に芽を入れる)が、水耕培地に浸漬され、その後、バクテリアfまたは共培養する。次いで、植物組織および細菌を分離して、同時に抽出および分析する。この数字は参考文献35から変更されています。
Protocol
Representative Results
Discussion
根分泌の漸進的な性質を考慮すると、 プラナで生産されたビルレンス誘発化学物質の濃度および動的植物 – 微生物相互作用に対するそれらの効果は、空間的および時間的勾配において生じる。この水耕栽培共生システムでは、微生物毒性または植物防御を誘導する合成植物ホルモンまたは化学物質は補充されない。対照的に、従来のアプローチを用いて、アセトシリンゴン…
Declarações
The authors have nothing to disclose.
Acknowledgements
Brian WeselowskiとAlexander W. Eastmanに感謝し、彼らの助けと役に立つ議論をしていただきたいと思います。私たちはDrsにも感謝したいと思います。 Eugene W. Nester、Lingrui Zhang、Haitao Shen、Yuhai Cui、Greg Thornの協力を得て、有用な議論と原稿の批評を読んだ。この研究は、農業および農業食品カナダ、Growing Forward-AgriFlex(RBPI番号2555)およびGrowing Forward IIプロジェクト番号1670(その職務の一環として著者が実施したもの)によって資金提供されました。この研究はまた、ZC Yuanに授与されたカナダの自然科学・工学研究評議会(NSERC)Discovery Grant RGPIN-2015-06052によって部分的に資金提供された。
Materials
| plant seeds (Arabidopsis thaliana Col-0) | Arabidopsis Biological Resource Centre | CS7000 | https://abrc.osu.edu/order-stocks |
| bacteria (Agrobacterium tumefaciens C58) | University of Washington | N/A | |
| labeled bacteria | in-house | optional, depends on downstream analyses | |
| vortex | (various) | ||
| microcentrifuge tubes | (various) | ||
| microcentrifuge | (various) | ||
| 5% sodium hypochlorite | (various) | ||
| double distilled water | (various) | ||
| autoclave | (various) | ||
| micropipette | (various) | ||
| 70 % ethanol | (various) | ||
| Murashige and Skoog (MS) basal salts | Sigma-Aldrich | M5524 | |
| sucrose | (various) | ||
| MES | (various) | ||
| B5 vitamin mix | Sigma-Aldrich | G1019 | |
| phytoagar | (various) | ||
| deep Petri dishes | (various) | ||
| stainless steel mesh | Ferrier Wire Goods Company Ltd | N/A | grade: 304; mesh count: 40 × 40; wire DIA: 0.01 |
| micropore tape, 1 inch | 3M | 1530-1 | |
| diurnal growth chamber | (various) | ||
| cylindrical glass tanks, 100 × 80 mm | Pyrex | 3250 | other sizes can be used, in which case liquid content may need adjustment |
| flow hood | (various) | ||
| forcepts | (various) | ||
| yeast extract | (various) | ||
| tryptone | (various) | ||
| MgSO4 | (various) | ||
| shaking incubator | (various) | ||
| spectrophotometer | (various) | ||
| NaCl | (various) | ||
| shaker | (various) | ||
| scissors | (various) | optional, depends on downstream analyses | |
| fluorescence microscope | (various) | optional, depends on downstream analyses | |
| microscope slides and cover slips | (various) | optional, depends on downstream analyses | |
| nail polish | (various) | optional, depends on downstream analyses | |
| Bacterial RNA extraction kit | (various) | optional, depends on downstream analyses | |
| plant RNA extraction kit (RNeasy Plant Mini Kit) | Qiagen | 74903 or 74904 | optional, depends on downstream analyses |
| material and equipment for RT-qPCR | (various) | optional, depends on downstream analyses | |
| material and equipment for microarray analysis | (various) | optional, depends on downstream analyses | |
| liquid nitrogen | (various) | optional, depends on downstream analyses | |
| mortar and pestle | (various) | optional, depends on downstream analyses | |
| 0.2 µm pore filter | (various) | optional, depends on downstream analyses | |
| 50 mL conical tubes | (various) | optional, depends on downstream analyses | |
| freeze dryer | (various) | optional, depends on downstream analyses | |
| sealable test tubes | (various) | optional, depends on downstream analyses | |
| ethyl acetate | (various) | optional, depends on downstream analyses | |
| nitrogen gas | (various) | optional, depends on downstream analyses | |
| material and equipment for HPLC | (various) | optional, depends on downstream analyses | |
| material and equipment for ESI-TOF-MS | (various) | optional, depends on downstream analyses |
Referências
- Lambers, H., Mougel, C., Jaillard, B., Hinsinger, P. Plant-microbe-soil interactions in the rhizosphere: An evolutionary perspective. Plant Soil. 321 (1), 83-115 (2009).
- Philippot, L., Raaijmakers, J. M., Lemanceau, P., vander Putten, W. H. Going back to the roots: The microbial ecology of the rhizosphere. Nat. Rev. Microbiol. 11 (11), 789-799 (2013).
- Somers, E., Vanderleyden, J., Srinivasan, M. Rhizosphere bacterial signalling: A Love Parade beneath our feet. Crit. Rev. Microbiol. 30 (4), 205-240 (2004).
- Barah, P., Winge, P., Kusnierczyk, A., Tran, D. H., Bones, A. M. Molecular signatures in Arabidopsis thaliana in response to insect attack and bacterial infection. PLoS ONE. 8 (3), (2013).
- Zhang, J., Zhou, J. -. M. Plant immunity triggered by microbial molecular signatures. Mol. Plant. 3 (5), 783-793 (2010).
- Paterson, E., Gebbing, T., Abel, C., Sim, A., Telfer, G. Rhizodeposition shapes rhizosphere microbial community structure in organic soil. New Phytol. 173 (3), 600-610 (2006).
- Hartmann, A., Schmid, M., van Tuinen, D., Berg, G. Plant-driven selection of microbes. Plant Soil. 321 (1-2), 235-257 (2008).
- Micallef, S. A., Shiaris, M. P., Colon-Carmona, A. Influence of Arabidopsis thaliana accessions on rhizobacterial communities and natural variation in root exudates. J. Exp. Bot. 60 (6), 1729-1742 (2009).
- Burr, T., Otten, L. Crown gall of grape: Biology and disease management. Annu. Rev. Phytopathol. 37, 53-80 (2001).
- Clough, S. J., Bent, A. F. Floral dip: A simplified method for Agrobacterium-mediated transformation of Arabidopsis thaliana. Plant J. 16 (6), 735-743 (1998).
- Binns, A. N., Costantino, P., Spaink, H. P., Kondorosi, A., Hooykaas, P. J. J. The Agrobacterium oncogenes. The Rhizobiaceae. , (1998).
- Gheysen, G., Angenon, G., Van Montagu, M., Lindsey, K. Agrobacterium-mediated plant transformation: a scientifically intriguing story with significant applications. Transgenic Plant Research. , (1998).
- Valvekens, D., Von Montagu, M. V., Van Lijsebettens, M. Agrobacterium tumefaciens-mediated transformation of Arabidopsis thaliana root explants by using kanamycin selection. Proc. Natl. Acad. Sci. USA. 85 (15), 5536-5540 (1988).
- Krysan, P. J., Young, J. C., Sussman, M. R. T-DNA as an insertional mutagen in Arabidopsis. Plant Cell. 11 (12), 2283 (1999).
- Chilton, M. D., et al. Stable incorporation of plasmid DNA into higher plant cells: The molecular basis of crown gall tumorigenesis. Cell. 11 (2), 263-271 (1977).
- Gelvin, S. B. Agrobacterium in the genomics age. Plant Physiol. 150 (4), 1665-1676 (2009).
- Subramoni, S., Nathoo, N., Klimov, E., Yuan, Z. -. C. Agrobacterium tumefaciens responses to plant-derived signaling molecules. Front. Plant Sci. 5, 322 (2014).
- Yuan, Z., Liu, P., Saenkham, P., Kerr, K., Nester, E. W. Transcriptome profiling and functional analysis of Agrobacterium tumefaciens reveals a general conserved response to acidic conditions (pH 5.5) and a complex acid-mediated signaling involved in Agrobacterium-plant interactions. J. Bacteriol. 190 (2), 494-507 (2008).
- Stachel, S. E., Messens, E., Van Montagu, M., Zambryski, P. Identification of the signal molecules produced by wounded plant cells that activate T-DNA transfer in Agrobacterium tumefaciens. Nature. 318 (6047), 624-629 (1985).
- Memelink, J., de Pater, B. S., Hoge, J. H. C., Schilperoort, R. A. T-DNA hormone biosynthetic genes: Phytohormones and gene expression in plants. Dev. Genet. 8 (5-6), 321-337 (1987).
- Yuan, Z. -. C., et al. The plant signal salicylic acid shuts down expression of the vir regulon and activates quormone-quenching genes in Agrobacterium. Proc. Natl. Acad. Sci. USA. 104 (28), 11790-11795 (2007).
- Yuan, Z. -. C., Haudecoeur, E., Faure, D., Kerr, K. F., Nester, E. W. Comparative transcriptome analysis of Agrobacterium tumefaciens in response to plant signal salicylic acid, indole-3-acetic acid and γ-amino butyric acid reveals signalling cross-talk and Agrobacterium-plant co-evolution. Cell. Microbiol. 10 (11), 2339-2354 (2008).
- Li, P. L., Farrand, S. K. The replicator of the nopaline-type Ti plasmid pTiC58 is a member of the repABC family and is influenced by the TraR-dependent quorum-sensing regulatory system. J. Bacteriol. 182 (1), 179-188 (2000).
- Atkinson, M. M., Huang, J., Knopp, J. A. Hypersensitivity of suspension-cultured tobacco cells to pathogenic bacteria. Phytopathology. 75 (11), 1270-1274 (1985).
- Veena, ., Jiang, H., Doerge, R. W., Gelvin, S. B. Transfer of T-DNA and Vir proteins to plant cells by Agrobacterium tumefaciens induces expression of host genes involved in mediating transformation and suppresses host defense gene expression. Plant J. 35 (2), 219-236 (2003).
- León, J., Rojo, E., Sanchez-Serrano, J. J. Wound signalling in plants. J. Exp. Bot. 52 (354), 1-9 (2001).
- Ditt, R. F., Kerr, K. F., de Figueiredo, P., Delrow, J., Comai, L., Nester, E. W. The Arabidopsis thaliana transcriptome in response to Agrobacterium tumefaciens. Mol. Plant Microbe In. 19 (6), 665-681 (2006).
- Mandimba, G., Heulin, T., Bally, R., Guckert, A., Balandreau, J. Chemotaxis of free-living nitrogen-fixing bacteria towards maize mucilage. Plant Soil. 90 (1-3), 129-139 (1986).
- Rudrappa, T., Czymmek, K. J., Pare, P. W., Bais, H. P. Root-secreted malic acid recruits beneficial soil bacteria. Plant Physiol. 148 (3), 1547-1556 (2008).
- Lee, C. W., et al. Agrobacterium tumefaciens promotes tumor induction by modulating pathogen defense in Arabidopsis thaliana. Plant Cell. 21 (9), 2948-2962 (2009).
- Reymond, P., Weber, H., Damond, M., Farmer, E. E. Differential gene expression in response to mechanical wounding and insect feeding in Arabidopsis. Plant Cell. 12 (5), 707-720 (2000).
- Matthysse, A. G. Attachment of Agrobacterium to plant surfaces. Front. Plant Sci. 5, 252 (2014).
- Alatorre-Cobos, F., et al. An improved, low-cost, hydroponic system for growing Arabidopsis and other plant species under aseptic conditions. BMC Plant Biol. 14 (1), 69 (2014).
- Conn, S. J., et al. Protocol: Optimising hydroponic growth systems for nutritional and physiological analysis of Arabidopsis thaliana and other plants. Plant Methods. 9 (1), 4 (2013).
- Nathoo, N. . Identification of putative plant defense genes using a novel hydroponic co-cultivation technique for studying plant-pathogen interaction. , (2015).
- Shaner, N. C., Campbell, R. E., Steinbach, P. A., Giepmans, B. N., Palmer, A. E., Tsien, R. Y. Improved monomeric red, orange and yellow fluorescent proteins derived from Discosoma sp. red fluorescent protein. Nat. Biotechnol. 22 (12), 1567-1572 (2004).
- Wise, A. A., Liu, H., Binns, A. N. Nucleic acid extraction from Agrobacterium strains. Methods Mol. Bio. 343, 67-76 (2006).
- Nolan, T., Hands, R. E., Bustin, S. A. Quantification of mRNA using real-time RT-PCR. Nat. Protoc. 1 (3), 1559-1582 (2006).
- Salter, M. G., Conlon, H. E. Extraction of plant RNA. Methods Mol. Bio. 362, 309-314 (2007).
- Bao, Y., Wang, S., Yang, X., Li, T., Xia, Y., Meng, X. Metabolomic study of the intervention effects of Shuihonghuazi Formula, a Traditional Chinese Medicinal formulae, on hepatocellular carcinoma (HCC) rats using performance HPLC/ESI-TOF-MS. J. Ethnopharmacol. 198, 468-478 (2017).
- Korves, T. M., Bergelson, J. A developmental response to pathogen infection in Arabidopsis. Plant Physiol. 133 (1), 339-347 (2003).
- Lyons, R., Rusu, A., Stiller, J., Powell, J., Manners, J. M., Kazan, K. Investigating the association between flowering time and defense in the Arabidopsis thaliana-Fusarium oxysporum interaction. PLoS ONE. 10 (6), e0127699 (2015).
- Badri, D. V., Weir, T. L., vander Lelie, D., Vivanco, J. M. Rhizosphere chemical dialogues: Plant-microbe interactions. Curr. Opin. Biotechnol. 20 (6), 642-650 (2009).
- Baerson, S. R., et al. Detoxification and transcriptome response in Arabidopsis seedlings exposed to the allelochemical benzoxazolin-2(3H)-one. J. Biol. Chem. 280 (23), 21867-21881 (2005).

