Um sistema de co-cultivo hidropônico para análise simultânea e sistemática de interações e sinalização moleculares de plantas/microorganismos
Summary
O sistema de cocultivação hidropônico descrito suporta plantas intactas com telas de malha metálica e as coculata com bactérias. O tecido vegetal, as bactérias e as moléculas segregadas podem então ser colhidas separadamente para análises a jusante, permitindo simultaneamente que as respostas moleculares de ambos os hospedeiros de plantas e micróbios ou microbiomas interativos sejam investigadas.
Abstract
Um projeto experimental que imita as interações planta-microbio natural é muito importante para delinear os complexos processos de sinalização de plantas e micróbios. Arabidopsis thaliana – Agrobacterium tumefaciens Fornece um excelente sistema modelo para estudar a patogênese bacteriana e as interações das plantas. Estudos prévios das interações de plantas e antibacterianos dependeram em grande parte das culturas de suspensão de células vegetais, do ferimento artificial de plantas ou da indução artificial de fatores de virulência microbiana ou defesas de plantas por produtos químicos sintéticos. No entanto, esses métodos são distintos da sinalização natural in planta , onde plantas e micróbios reconhecem e respondem de maneiras espaciais e temporais. Este trabalho apresenta um sistema de cocultivação hidropônica onde as plantas intactas são suportadas por telas de malha metálica e coculadas com Agrobacterium . Neste sistema de cocultivação, nenhuma fitohormona sintética ou química que induz micr.A virulência obial ou a defesa da planta são complementadas. O sistema de cocultivação hidropônica parece muito parecido com as interações planta-micróbio natural e sinalização da homeostase na planta . As raízes das plantas podem ser separadas do meio contendo Agrobacterium , e a sinalização e as respostas dos hospedeiros da planta e dos micróbios que interagem podem ser investigadas simultaneamente e sistematicamente. Em qualquer ponto / intervalo de tempo dado, os tecidos ou bactérias da planta podem ser colhidos separadamente para várias análises "ôticas", demonstrando o poder e a eficácia deste sistema. O sistema de co-cultura hidropónica pode ser facilmente adaptado para estudar: 1) a sinalização recíproca dos diversos sistemas de planta e microorganismos, 2) a sinalização entre uma planta hospedeira e várias espécies microbianas (ou seja consórcio microbiano ou microbiomas), 3) como nutrientes e produtos químicos estão implicados Na sinalização de microbios vegetais e 4) como os micróbios interagem com os hospedeiros da planta e contribuem para a tolerância das plantas ao biotico oEstresses abióticos.
Introduction
Os micróbios associados à planta desempenham papéis importantes no ciclismo biogeoquimico, na biorremediação, na mitigação das alterações climáticas, no crescimento e na saúde das plantas e na tolerância das plantas aos estresses bióticos e abióticos. Os microorganismos interagem com as plantas diretamente através do contato da parede celular da planta e indiretamente através de secreção química e sinalização 1 , 2 , 3 . Como organismos sésseis, as plantas desenvolveram mecanismos diretos e indiretos para resistir à infecção por agentes patogênicos. As defesas diretas incluem defesas estruturais e a expressão de proteínas de defesa, enquanto que as defesas indiretas incluem a produção secundária de metabólitos das plantas e a atração de organismos antagônicos aos patógenos invasores 4 , 5 . Os exsudados de raiz derivados de plantas, secreções, mucilagens, mucigel e lisados alteram as propriedades físico-químicas da rizosfera para atrair ou repelirMicróbios em relação aos seus anfitriões 6 . A composição química da secreção de raízes é específica da espécie, servindo assim como um filtro seletivo que permite que certos microrganismos capazes de reconhecer esses compostos florescerem na rizósfera 6 . Assim, espécies microbianas compatíveis podem ser estimuladas para ativar e melhorar suas associações, quer para benefício ou prejuízo do hospedeiro da planta 1 .
Compreender as interações planta-microbio na rizósfera é fundamental para melhorar a produtividade da planta e o funcionamento do ecossistema, uma vez que a maioria da exposição microbiana e química ocorre na estrutura radicular e na interface solo-ar 2 , 6 , 7 , 8 . No entanto, o exame das interações subterrâneas de plantas e micróbios e respostas recíprocas tem sido um desafio devido à sua intrigante Natureza complexa e dinâmica e falta de modelos experimentais adequados com estrutura radicular natural e morfologia da planta sob condições de crescimento bem controláveis. Como um dos agentes farmacêuticos mais bem estudados, Agrobacterium infecta uma grande variedade de plantas com importância agrícola e horticultural, incluindo cereja, maçã, pera, uva e rosa 9 . Agrobacterium é um organismo modelo importante para a compreensão das interações planta-patógeno e é uma ferramenta poderosa na transformação de plantas e engenharia de plantas 10 , 11 , 12 , 13 , 14 .
As interacções Molecular Plant- Agrobacterium foram bem estudadas há várias décadas e a compreensão atual da patogenicidade de Agrobacterium é extensa 9 ,F. "> 11 , 15 , 16. A patogenicidade de Agrobacterium é em grande parte atribuída às suas capacidades evoluídas de percepção de sinais derivados de plantas, resultando na modulação fina de seu programa de virulência e comunicação célula a célula, o chamado sensor de quorum 17 . O programa Agrobacterium virulence é regulado por vários sinais disponíveis na rizosfera e envolve dois conjuntos de sistemas de 2 componentes, o sistema ChvG / I eo sistema VirA / G. As condições ácidas na rizósfera ativam a transcrição de chvG / I , virA / G , E vários outros genes envolvidos na patogenicidade de Agrobacterium , incluindo virE0 , virE1 , virH1 , virH2 e genes do sistema de secreção de tipo VI (T6SS) 18. Compostos fenólicos derivados de plantas, incluindo a aceossiringona (4'-hidroxi-3 ', 5 '-dimetoxiacetofenona), ative o VSistema IrA / G de 2 componentes através de mecanismos de sinalização de fosforilação 19 . VirA / G ativa então todo o regulador vir , resultando na transferência e integração de um fragmento de DNA bacteriano de ~ 20 kb chamado DNA de transferência (DNA-T) do plasmídeo indutor de tumor (Ti) para o núcleo da planta 16 . T-DNA carrega genes responsáveis pela síntese dos hormônios vegetais ácido indol-3-acético (IAA) ( iaaM e iaaH ) e citoquinina ( ipt ), e uma vez expressa em células vegetais, produzem-se grandes quantidades dessas fitohormonas. Isso resulta em proliferação anormal de tecidos e desenvolvimento de tumores vegetais, conhecida como doença coronária, que é um problema crônico e ressurgente para as plantas 9 , 11 , 20 . IAA também atua coletivamente com ácido salicílico e ácido gamma-amino butírico para reprimir a virulência de Agrobacterium ou para reduzir Agrobacteriu M quorum sensing (QS) 17 , 21 , 22 . Para contrariar essa repressão, o T-DNA também contém genes para a biossíntese de opina, que ativa o Agrobacterium quorum detectando para promover a patogenicidade de Agrobacterium e também serve como fonte de nutrientes para o patógeno 22 , 23 .
Apesar de uma compreensão geral e profunda das interações de plantas de Agrobacterium e da transferência de T-DNA resultante para o hospedeiro da planta, os complexos eventos de sinalização na fase inicial de interação são menos bem compreendidos. Isto é parcialmente devido às limitações das abordagens convencionais para investigar a sinalização das plantas de Agrobacterium . As culturas em suspensão de células vegetais e os ferimentos artificiais específicos do local são comumente usados para estudar interações moleculares de plantas e micróbios 24 ,. ef "> 26, 27 No entanto, as suspensões de células não possuem morfologia da planta normal, em particular, as células em suspensão planta não tem estruturas de raiz e exsudatos de raízes, que são muito importantes para a activação de quimiotaxia microbiana e virulência 28, 29 A manutenção da morfologia da planta. E a estrutura da raiz foi abordada por feridas artificiais, o que facilita a infecção específica do local, resultando na detecção de genes induzidos por defesa da planta em tecido de plantas infectadas diretamente 30 , 31. Contudo, o ferimento artificial é significativamente diferente da infecção por patógenos na natureza , Particularmente como ferimento leva ao acúmulo de ácido jasmônico (JA), que interfere sistematicamente com a sinalização e defesa de plantas naturais 26. Além disso, os produtos químicos sintéticos são tipicamente usados para induzir artificialmente as respostas do hospedeiro da plantaOu virulência de patógenos. Embora seja possível a suplementação de compostos químicos que refletem as concentrações em planta , essa suplementação não explica gradualmente a difusão de exsudatos radiculares na rizósfera circundante, o que gera um gradiente quimiotático detectado pelos micróbios 28 , 32 . Dadas as limitações das abordagens convencionais para estudar as interações planta-microbio, a precisão e a profundidade dos dados obtidos podem ser impedidas e restritivas e o conhecimento gerado a partir das abordagens convencionais pode não se traduz diretamente na planta . Muitos aspectos da sinalização das plantas – Agrobacterium ainda não são totalmente compreendidos, particularmente no estágio inicial das interações, quando os sintomas da doença ainda não se desenvolveram.
Para alterar as limitações das abordagens convencionais, este trabalho apresenta um c hidroponico barato, bem controlável e flexívelSistema de ocultivação que permite aos pesquisadores obter informações mais aprofundadas sobre as vias complexas de sinalização e resposta no estágio inicial das interações de plantas-micróbios moleculares. O hidroponia tem sido amplamente utilizado para estudar nutrientes de plantas, exsudatos radiculares, condições de crescimento e os efeitos da toxicidade metálica nas plantas 33 , 34 . Existem várias vantagens dos modelos hidropônicos, incluindo os pequenos requisitos espaciais, a acessibilidade de vários tecidos vegetais, o controle rigoroso das condições nutrientes / ambientais e o controle de pragas / doenças. Os sistemas hidropônicos também são menos limitantes para o crescimento das plantas em comparação com as técnicas de revestimento em ágar / fitoagulação, que geralmente restringem o crescimento após 2-3 semanas. Importante, a manutenção de estruturas de plantas inteiras facilita a secreção de raízes naturais necessárias para a quimiotaxia microbiana e indução de virulência 8 , 29 . O sistema descriA cama aqui é mais simples e menos intensiva em mão-de-obra do que as alternativas 33 , 34 . Ele usa menos peças e não requer ferramentas além das tesouras padrão. Ele usa malha de metal (em oposição ao nylon 33 ) como um forte suporte para o crescimento da planta e um método simples de aeração sob condições estéreis através de agitação para suportar o crescimento microbiano. Além disso, o sistema pode usar malhas metálicas de vários tamanhos para suportar o crescimento da planta, que acomoda diversas espécies de plantas sem restringir a largura de suas raízes.
No sistema de cocultivação hidropônica apresentado aqui, as plantas são cultivadas em um sistema hidropônico estéril onde as raízes das plantas secretam compostos orgânicos que suportam o crescimento de bactérias inoculadas. Neste sistema de cocultivação, não são suplementados produtos químicos artificiais, como hormonas de plantas, elicitores de defesa ou produtos químicos indutores de virulência, que refletem a célula natural- assinalando a homeostase durante as interações planta-micróbio. Com este sistema de cocultivação hidropônica, foi possível determinar simultaneamente a expressão gênica no tecido da raiz de Col-0 de Arabidopsis thaliana após a infecção por Agrobacterium , bem como a ativação de genes de Agrobacterium após a cocultivação com Arabidopsis . Demonstrou-se ainda que este sistema é adequado para estudar a ligação de Agrobacterium às raízes das plantas, bem como o perfil secreto da raiz da planta, após a cocultivação (infecção) com Agrobacterium ( Figura 1 ).
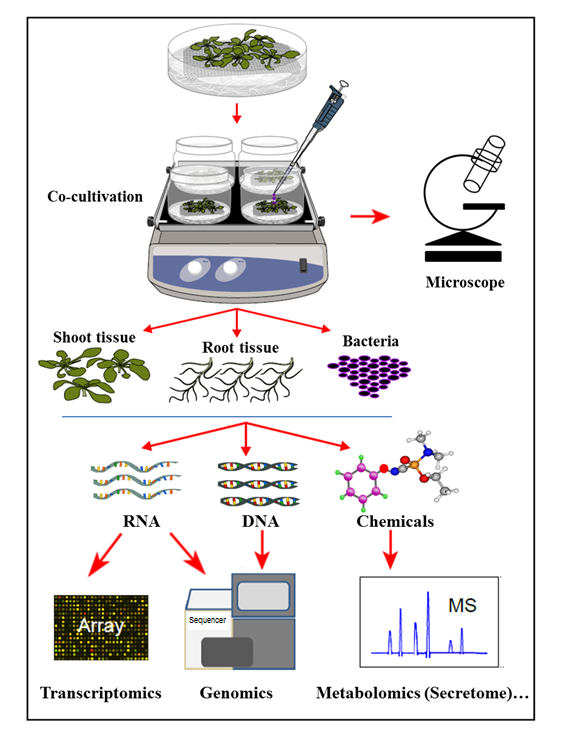
Figura 1: Visão geral do sistema de cocultivação hidropônica, com análises de amostras. As plantas são cultivadas em cima da malha (brotos acima da malha), com as raízes imersas em meio hidropônico que é então inoculado com bactérias fOu coculture. Os tecidos vegetais e as bactérias são então separados para extrações e análises simultâneas. Esta figura foi modificada a partir da referência 35 .
Protocol
Representative Results
Discussion
Dada a natureza gradual da secreção de raízes, a concentração de produtos químicos indutores de virulência produzidos em planta e seus efeitos sobre interações dinâmicas de plantas e micróbios ocorrem em gradientes espaciais e temporais. Neste sistema de co-cultivo hidropônico, não é suplementada nenhuma fitohormona sintética ou química que induz virulência microbiana ou defesas de plantas. Em contraste, usando abordagens convencionais, a adição de produtos químicos sintéticos, como a aceto…
Declarações
The authors have nothing to disclose.
Acknowledgements
Gostaríamos de agradecer Brian Weselowski e Alexander W. Eastman por sua ajuda e discussão útil. Também gostaríamos de agradecer aos Drs. Eugene W. Nester, Lingrui Zhang, Haitao Shen, Yuhai Cui e Greg Thorn por sua ajuda, discussões úteis e leitura crítica do manuscrito. Esta pesquisa foi financiada pela Agricultura e Agroalimentar Canadá, Growing Forward-AgriFlex (RBPI número 2555) e Growing Forward II projeto número 1670, conduzido pelos autores como parte de suas funções. Este estudo também foi parcialmente financiado pelo Conselho de Pesquisa em Ciências Naturais e Engenharia do Canadá (CRSNG) Discovery Grant RGPIN-2015-06052 concedido a ZC Yuan.
Materials
| plant seeds (Arabidopsis thaliana Col-0) | Arabidopsis Biological Resource Centre | CS7000 | https://abrc.osu.edu/order-stocks |
| bacteria (Agrobacterium tumefaciens C58) | University of Washington | N/A | |
| labeled bacteria | in-house | optional, depends on downstream analyses | |
| vortex | (various) | ||
| microcentrifuge tubes | (various) | ||
| microcentrifuge | (various) | ||
| 5% sodium hypochlorite | (various) | ||
| double distilled water | (various) | ||
| autoclave | (various) | ||
| micropipette | (various) | ||
| 70 % ethanol | (various) | ||
| Murashige and Skoog (MS) basal salts | Sigma-Aldrich | M5524 | |
| sucrose | (various) | ||
| MES | (various) | ||
| B5 vitamin mix | Sigma-Aldrich | G1019 | |
| phytoagar | (various) | ||
| deep Petri dishes | (various) | ||
| stainless steel mesh | Ferrier Wire Goods Company Ltd | N/A | grade: 304; mesh count: 40 × 40; wire DIA: 0.01 |
| micropore tape, 1 inch | 3M | 1530-1 | |
| diurnal growth chamber | (various) | ||
| cylindrical glass tanks, 100 × 80 mm | Pyrex | 3250 | other sizes can be used, in which case liquid content may need adjustment |
| flow hood | (various) | ||
| forcepts | (various) | ||
| yeast extract | (various) | ||
| tryptone | (various) | ||
| MgSO4 | (various) | ||
| shaking incubator | (various) | ||
| spectrophotometer | (various) | ||
| NaCl | (various) | ||
| shaker | (various) | ||
| scissors | (various) | optional, depends on downstream analyses | |
| fluorescence microscope | (various) | optional, depends on downstream analyses | |
| microscope slides and cover slips | (various) | optional, depends on downstream analyses | |
| nail polish | (various) | optional, depends on downstream analyses | |
| Bacterial RNA extraction kit | (various) | optional, depends on downstream analyses | |
| plant RNA extraction kit (RNeasy Plant Mini Kit) | Qiagen | 74903 or 74904 | optional, depends on downstream analyses |
| material and equipment for RT-qPCR | (various) | optional, depends on downstream analyses | |
| material and equipment for microarray analysis | (various) | optional, depends on downstream analyses | |
| liquid nitrogen | (various) | optional, depends on downstream analyses | |
| mortar and pestle | (various) | optional, depends on downstream analyses | |
| 0.2 µm pore filter | (various) | optional, depends on downstream analyses | |
| 50 mL conical tubes | (various) | optional, depends on downstream analyses | |
| freeze dryer | (various) | optional, depends on downstream analyses | |
| sealable test tubes | (various) | optional, depends on downstream analyses | |
| ethyl acetate | (various) | optional, depends on downstream analyses | |
| nitrogen gas | (various) | optional, depends on downstream analyses | |
| material and equipment for HPLC | (various) | optional, depends on downstream analyses | |
| material and equipment for ESI-TOF-MS | (various) | optional, depends on downstream analyses |
Referências
- Lambers, H., Mougel, C., Jaillard, B., Hinsinger, P. Plant-microbe-soil interactions in the rhizosphere: An evolutionary perspective. Plant Soil. 321 (1), 83-115 (2009).
- Philippot, L., Raaijmakers, J. M., Lemanceau, P., vander Putten, W. H. Going back to the roots: The microbial ecology of the rhizosphere. Nat. Rev. Microbiol. 11 (11), 789-799 (2013).
- Somers, E., Vanderleyden, J., Srinivasan, M. Rhizosphere bacterial signalling: A Love Parade beneath our feet. Crit. Rev. Microbiol. 30 (4), 205-240 (2004).
- Barah, P., Winge, P., Kusnierczyk, A., Tran, D. H., Bones, A. M. Molecular signatures in Arabidopsis thaliana in response to insect attack and bacterial infection. PLoS ONE. 8 (3), (2013).
- Zhang, J., Zhou, J. -. M. Plant immunity triggered by microbial molecular signatures. Mol. Plant. 3 (5), 783-793 (2010).
- Paterson, E., Gebbing, T., Abel, C., Sim, A., Telfer, G. Rhizodeposition shapes rhizosphere microbial community structure in organic soil. New Phytol. 173 (3), 600-610 (2006).
- Hartmann, A., Schmid, M., van Tuinen, D., Berg, G. Plant-driven selection of microbes. Plant Soil. 321 (1-2), 235-257 (2008).
- Micallef, S. A., Shiaris, M. P., Colon-Carmona, A. Influence of Arabidopsis thaliana accessions on rhizobacterial communities and natural variation in root exudates. J. Exp. Bot. 60 (6), 1729-1742 (2009).
- Burr, T., Otten, L. Crown gall of grape: Biology and disease management. Annu. Rev. Phytopathol. 37, 53-80 (2001).
- Clough, S. J., Bent, A. F. Floral dip: A simplified method for Agrobacterium-mediated transformation of Arabidopsis thaliana. Plant J. 16 (6), 735-743 (1998).
- Binns, A. N., Costantino, P., Spaink, H. P., Kondorosi, A., Hooykaas, P. J. J. The Agrobacterium oncogenes. The Rhizobiaceae. , (1998).
- Gheysen, G., Angenon, G., Van Montagu, M., Lindsey, K. Agrobacterium-mediated plant transformation: a scientifically intriguing story with significant applications. Transgenic Plant Research. , (1998).
- Valvekens, D., Von Montagu, M. V., Van Lijsebettens, M. Agrobacterium tumefaciens-mediated transformation of Arabidopsis thaliana root explants by using kanamycin selection. Proc. Natl. Acad. Sci. USA. 85 (15), 5536-5540 (1988).
- Krysan, P. J., Young, J. C., Sussman, M. R. T-DNA as an insertional mutagen in Arabidopsis. Plant Cell. 11 (12), 2283 (1999).
- Chilton, M. D., et al. Stable incorporation of plasmid DNA into higher plant cells: The molecular basis of crown gall tumorigenesis. Cell. 11 (2), 263-271 (1977).
- Gelvin, S. B. Agrobacterium in the genomics age. Plant Physiol. 150 (4), 1665-1676 (2009).
- Subramoni, S., Nathoo, N., Klimov, E., Yuan, Z. -. C. Agrobacterium tumefaciens responses to plant-derived signaling molecules. Front. Plant Sci. 5, 322 (2014).
- Yuan, Z., Liu, P., Saenkham, P., Kerr, K., Nester, E. W. Transcriptome profiling and functional analysis of Agrobacterium tumefaciens reveals a general conserved response to acidic conditions (pH 5.5) and a complex acid-mediated signaling involved in Agrobacterium-plant interactions. J. Bacteriol. 190 (2), 494-507 (2008).
- Stachel, S. E., Messens, E., Van Montagu, M., Zambryski, P. Identification of the signal molecules produced by wounded plant cells that activate T-DNA transfer in Agrobacterium tumefaciens. Nature. 318 (6047), 624-629 (1985).
- Memelink, J., de Pater, B. S., Hoge, J. H. C., Schilperoort, R. A. T-DNA hormone biosynthetic genes: Phytohormones and gene expression in plants. Dev. Genet. 8 (5-6), 321-337 (1987).
- Yuan, Z. -. C., et al. The plant signal salicylic acid shuts down expression of the vir regulon and activates quormone-quenching genes in Agrobacterium. Proc. Natl. Acad. Sci. USA. 104 (28), 11790-11795 (2007).
- Yuan, Z. -. C., Haudecoeur, E., Faure, D., Kerr, K. F., Nester, E. W. Comparative transcriptome analysis of Agrobacterium tumefaciens in response to plant signal salicylic acid, indole-3-acetic acid and γ-amino butyric acid reveals signalling cross-talk and Agrobacterium-plant co-evolution. Cell. Microbiol. 10 (11), 2339-2354 (2008).
- Li, P. L., Farrand, S. K. The replicator of the nopaline-type Ti plasmid pTiC58 is a member of the repABC family and is influenced by the TraR-dependent quorum-sensing regulatory system. J. Bacteriol. 182 (1), 179-188 (2000).
- Atkinson, M. M., Huang, J., Knopp, J. A. Hypersensitivity of suspension-cultured tobacco cells to pathogenic bacteria. Phytopathology. 75 (11), 1270-1274 (1985).
- Veena, ., Jiang, H., Doerge, R. W., Gelvin, S. B. Transfer of T-DNA and Vir proteins to plant cells by Agrobacterium tumefaciens induces expression of host genes involved in mediating transformation and suppresses host defense gene expression. Plant J. 35 (2), 219-236 (2003).
- León, J., Rojo, E., Sanchez-Serrano, J. J. Wound signalling in plants. J. Exp. Bot. 52 (354), 1-9 (2001).
- Ditt, R. F., Kerr, K. F., de Figueiredo, P., Delrow, J., Comai, L., Nester, E. W. The Arabidopsis thaliana transcriptome in response to Agrobacterium tumefaciens. Mol. Plant Microbe In. 19 (6), 665-681 (2006).
- Mandimba, G., Heulin, T., Bally, R., Guckert, A., Balandreau, J. Chemotaxis of free-living nitrogen-fixing bacteria towards maize mucilage. Plant Soil. 90 (1-3), 129-139 (1986).
- Rudrappa, T., Czymmek, K. J., Pare, P. W., Bais, H. P. Root-secreted malic acid recruits beneficial soil bacteria. Plant Physiol. 148 (3), 1547-1556 (2008).
- Lee, C. W., et al. Agrobacterium tumefaciens promotes tumor induction by modulating pathogen defense in Arabidopsis thaliana. Plant Cell. 21 (9), 2948-2962 (2009).
- Reymond, P., Weber, H., Damond, M., Farmer, E. E. Differential gene expression in response to mechanical wounding and insect feeding in Arabidopsis. Plant Cell. 12 (5), 707-720 (2000).
- Matthysse, A. G. Attachment of Agrobacterium to plant surfaces. Front. Plant Sci. 5, 252 (2014).
- Alatorre-Cobos, F., et al. An improved, low-cost, hydroponic system for growing Arabidopsis and other plant species under aseptic conditions. BMC Plant Biol. 14 (1), 69 (2014).
- Conn, S. J., et al. Protocol: Optimising hydroponic growth systems for nutritional and physiological analysis of Arabidopsis thaliana and other plants. Plant Methods. 9 (1), 4 (2013).
- Nathoo, N. . Identification of putative plant defense genes using a novel hydroponic co-cultivation technique for studying plant-pathogen interaction. , (2015).
- Shaner, N. C., Campbell, R. E., Steinbach, P. A., Giepmans, B. N., Palmer, A. E., Tsien, R. Y. Improved monomeric red, orange and yellow fluorescent proteins derived from Discosoma sp. red fluorescent protein. Nat. Biotechnol. 22 (12), 1567-1572 (2004).
- Wise, A. A., Liu, H., Binns, A. N. Nucleic acid extraction from Agrobacterium strains. Methods Mol. Bio. 343, 67-76 (2006).
- Nolan, T., Hands, R. E., Bustin, S. A. Quantification of mRNA using real-time RT-PCR. Nat. Protoc. 1 (3), 1559-1582 (2006).
- Salter, M. G., Conlon, H. E. Extraction of plant RNA. Methods Mol. Bio. 362, 309-314 (2007).
- Bao, Y., Wang, S., Yang, X., Li, T., Xia, Y., Meng, X. Metabolomic study of the intervention effects of Shuihonghuazi Formula, a Traditional Chinese Medicinal formulae, on hepatocellular carcinoma (HCC) rats using performance HPLC/ESI-TOF-MS. J. Ethnopharmacol. 198, 468-478 (2017).
- Korves, T. M., Bergelson, J. A developmental response to pathogen infection in Arabidopsis. Plant Physiol. 133 (1), 339-347 (2003).
- Lyons, R., Rusu, A., Stiller, J., Powell, J., Manners, J. M., Kazan, K. Investigating the association between flowering time and defense in the Arabidopsis thaliana-Fusarium oxysporum interaction. PLoS ONE. 10 (6), e0127699 (2015).
- Badri, D. V., Weir, T. L., vander Lelie, D., Vivanco, J. M. Rhizosphere chemical dialogues: Plant-microbe interactions. Curr. Opin. Biotechnol. 20 (6), 642-650 (2009).
- Baerson, S. R., et al. Detoxification and transcriptome response in Arabidopsis seedlings exposed to the allelochemical benzoxazolin-2(3H)-one. J. Biol. Chem. 280 (23), 21867-21881 (2005).

