Un système de co-culture hydroponique pour l'analyse simultanée et systématique des interactions et signalisation moléculaire des plantes / microbes
Summary
Le système de cocultivation hydroponique décrit supporte des plantes intactes avec des écrans en mèches métalliques et les cocultive avec des bactéries. Les tissus végétaux, les bactéries et les molécules sécrétées peuvent ensuite être récoltées séparément pour des analyses en aval, permettant simultanément d'étudier les réponses moléculaires des hôtes de la plante et des microbes ou microbiomes en interaction.
Abstract
Une conception expérimentale imitant les interactions naturelles plantes-microbes est très importante pour délimiter les processus complexes de signalisation des plantes-microbes. Arabidopsis thaliana – Agrobacterium tumefaciens Fournit un excellent système modèle pour étudier la pathogenèse bactérienne et les interactions avec les plantes. Les études antérieures sur les interactions plantes- acides gras ont largement dépendu des cultures en suspension de cellules végétales, des blessures artificielles de plantes ou de l'induction artificielle de facteurs de virulence microbienne ou des défenses végétales par des produits chimiques de synthèse. Cependant, ces méthodes sont distinctes de la signalisation naturelle in planta , où les plantes et les microbes reconnaissent et répondent selon les manières spatiales et temporelles. Ce travail présente un système de cocultivation hydroponique où les plantes intactes sont soutenues par des écrans à mailles métalliques et cocultivées avec Agrobacterium . Dans ce système de cocultivation, aucune phytohormone synthétique ou chimique qui induit une micr.La virulence obial ou la défense végétale est complétée. Le système de cocultivation hydroponique ressemble étroitement aux interactions naturelles plantes-microbes et à la signalisation de l'homéostasie in planta . Les racines des plantes peuvent être séparées du milieu contenant Agrobacterium , et la signalisation et les réponses des hôtes de la plante et des microbes en interaction peuvent être étudiées simultanément et systématiquement. À n'importe quel moment / intervalle donné, les tissus végétaux ou les bactéries peuvent être récoltés séparément pour diverses analyses "omiques", démontrant la puissance et l'efficacité de ce système. Le système de cocultivation hydroponique peut être facilement adapté pour étudier: 1) la signalisation réciproque de divers systèmes de microbes végétaux, 2) la signalisation entre un hôte végétal et plusieurs espèces microbiennes ( c.-à-d. Les consortiums microbiens ou les microbiomes), 3) comment les éléments nutritifs et les produits chimiques sont impliqués Dans la signalisation des plantes-microbes, et 4) comment les microbes interagissent avec les hôtes de la plante et contribuent à la tolérance des plantes au biotique oR stress abiotique.
Introduction
Les microbes associés aux plantes jouent un rôle important dans le cyclisme biogéochimique, la bioremédiation, l'atténuation des changements climatiques, la croissance et la santé des plantes et la tolérance des plantes aux contraintes biotiques et abiotiques. Les microorganismes interagissent avec les plantes directement à travers le contact de la paroi cellulaire de la plante et indirectement par la sécrétion chimique et la signalisation 1 , 2 , 3 . En tant qu'organes sessiles, les plantes ont développé des mécanismes directs et indirects pour résister à l'infection par des agents pathogènes. Les défenses directes comprennent les défenses structurelles et l'expression des protéines de défense, tandis que les défenses indirectes incluent la production secondaire de métabolites de plantes et l'attraction d'organismes antagonistes aux pathogènes envahissants 4 , 5 . Les exsudats de racines dérivés des plantes, les sécrétions, les mucilages, les mucigel et les lysats modifient les propriétés physico-chimiques de la rhizosphère pour attirer ou repousserDes microbes vers leurs hôtes 6 . La composition chimique de la sécrétion des racines est spécifique à l'espèce, servant ainsi de filtre sélectif qui permet à certains microorganismes capables de reconnaître de tels composés se développer dans la rhizosphère 6 . Ainsi, des espèces microbiennes compatibles peuvent être stimulées pour activer et améliorer leurs associations, soit au bénéfice ou au détriment de l'hôte 1 de la plante.
La compréhension des interactions plantes-microbes dans la rhizosphère est essentielle pour améliorer la productivité des plantes et le fonctionnement de l'écosystème, car une majorité de l'exposition microbienne et chimique se produit à la structure racine et à l'interface sol-air 2 , 6 , 7 , 8 . Cependant, l'examen des interactions souterraines des plantes-microbes et des réponses réciproques a été un défi en raison de son intrigué La nature complexe et dynamique et le manque de modèles expérimentaux appropriés avec structure racine naturelle et morphologie végétale dans des conditions de croissance très contrôlables. Comme l'un des phytopathogènes les plus fortement étudiés, Agrobacterium infecte un large éventail de plantes d'importance agricole et horticole, y compris la cerise, la pomme, la poire, le raisin et la rose 9 . Agrobacterium est un organisme modèle important pour la compréhension des interactions plantes-pathogènes et est un outil puissant dans la transformation des plantes et l'ingénierie des usines 10 , 11 , 12 , 13 , 14 .
Les interactions Molecular Plant- Agrobacterium ont été bien étudiées depuis plusieurs décennies et la compréhension actuelle de la pathogenèse d' Agrobacterium est importante 9 ,F "> 11 , 15 , 16. La pathogénicité de l' agrobacterium est largement attribuée à ses capacités évoluées de perception des signaux dérivés des plantes, ce qui entraîne la modulation fine de son programme de virulence et la communication cellulaire à cellulaire, appelée détection de quorum 17 . Le programme Agrobacterium virulence est réglementé par plusieurs signaux disponibles dans la rhizosphère et implique deux ensembles de systèmes à 2 composants, le système ChvG / I et le système VirA / G. Les conditions acides de la rhizosphère activent la transcription de chvG / I , virA / G , Et plusieurs autres gènes impliqués dans la pathogénicité d' Agrobacterium , y compris virE0 , virE1 , virH1 , virH2 et les gènes du système de sécrétion de type VI (T6SS) 18. Composés phénoliques dérivés des plantes, y compris l'acétosyringone (4'-hydroxy-3 ', 5 '-diméthoxyacétophénone), activez le VSystème à 2 composants irA / G par des mécanismes de signalisation de phosphorylation 19 . VirA / G active alors tout le régulateur vir , ce qui entraîne le transfert et l'intégration d'un fragment d'ADN bactérien de ~ 20 kb appelé ADN de transfert (ADN-T) à partir de son plasmide inducteur de tumeur (Ti) dans le noyau de la plante 16 . T-DNA porte des gènes responsables de la synthèse des hormones végétales de l'acide indole-3-acétique (IAA) ( iaaM et iaaH ) et de la cytokinine ( ipt ), et une fois exprimée dans les cellules végétales, de grandes quantités de ces phytohormones sont produites. Il en résulte une prolifération anormale des tissus et un développement de la tumeur végétale, connue sous le nom de maladie de la couronne, qui est un problème chronique et résurgent pour les plantes 9 , 11 , 20 . IAA agit également collectivement avec de l'acide salicylique et de l'acide gamma-amino butyrique pour réprimer Agrobacterium virulence ou pour réduire Agrobacteriu M quorum sensing (QS) 17 , 21 , 22 . Pour contrer cette répression, l'ADN-T porte également des gènes pour la biosynthèse de l'opine, qui active Agrobacterium quorum qui détecte la pathogénicité d' Agrobacterium et sert également de source nutritive pour le pathogène 22 , 23 .
En dépit d'une compréhension générale globale des interactions avec les plantes d' Agrobacterium et du transfert d'ADN-T résultant dans l'hôte de la plante, les événements de signalisation complexes au stade initial de l'interaction sont moins bien compris. Ceci est en partie dû aux limites des approches classiques pour enquêter sur la signalisation des plantes Agrobacterium . Les cultures en suspension de cellules végétales et les blessures artificielles spécifiques au site sont couramment utilisées pour étudier les interactions moléculaires-microbes 24 ,. ef "> 26, 27 Cependant, les suspensions cellulaires manquent la morphologie des plantes typiques, en particulier, les cellules de suspension des plantes ne sont pas des structures profondes et exsudats racinaires, qui sont très importants pour l' activation chimiotactisme microbienne et la virulence 28, 29 Le maintien de la morphologie des plantes. Et la structure racine a été traitée par des plantes blessant artificiellement, ce qui facilite l'infection spécifique au site, ce qui entraîne la détection de gènes induits par la défense des plantes dans les tissus végétaux infectés directement 30 , 31. Cependant, les blessures artificielles sont significativement différentes de l'infection pathogène dans la nature , D'autant plus que les blessures entraînent une accumulation d'acide jasmonique (JA), qui interfère systémiquement avec la signalisation et la défense des plantes naturelles 26. En outre, les produits chimiques de synthèse sont généralement utilisés pour induire artificiellement les réponses de l'hôte de la planteOu la virulence des agents pathogènes. Bien que la supplémentation de tels composés chimiques reflétant les concentrations en planta soit possible, une telle supplémentation ne tient pas compte de la diffusion des exsudats radiculaires progressivement dans la rhizosphère environnante, ce qui génère un gradient chimiotactique détecté par les microbes 28 , 32 . Compte tenu des limites des approches conventionnelles pour étudier les interactions entre les plantes et les microbes, la précision et la profondeur des données obtenues pourraient être entravées et restrictives, et les connaissances générées par les approches conventionnelles ne peuvent pas se traduire directement en plan . De nombreux aspects de la signalisation des plantes- Agrobacterium ne sont pas encore entièrement compris, en particulier au stade précoce des interactions, lorsque les symptômes de la maladie ne sont pas encore développés.
Pour modifier les limites des approches conventionnelles, ce travail présente un hydroponique c caisse peu coûteux, étroitement contrôlable et flexibleSystème d'ocultivation qui permet aux chercheurs d'acquérir des connaissances plus approfondies sur les voies complexes de signalisation et de réponse au stade initial des interactions moléculaire-microbe moléculaire. La culture hydroponique a été largement utilisée pour étudier les nutriments des plantes, les exsudats radiculaires, les conditions de croissance et les effets de la toxicité métallique sur les plantes 33 , 34 . Il existe plusieurs avantages des modèles hydroponiques, y compris les petites exigences spatiales, l'accessibilité de divers tissus végétaux, le contrôle strict des conditions nutritives / environnementales et le contrôle des ravageurs / maladies. Les systèmes hydroponiques sont également moins limitatifs pour la croissance des plantes par rapport aux techniques de gélose / phytoagar, ce qui limite généralement la croissance après 2-3 semaines. Fait important, le maintien de structures entières facilite la sécrétion naturelle des racines nécessaire à la chimiotaxie microbienne et à l'induction de la virulence 8 , 29 . Le système décriLe lit ici est plus simple et moins exigeant en main-d'œuvre que les alternatives 33 , 34 . Il utilise moins de pièces et ne nécessite aucun outil autre que les ciseaux standard. Il utilise des mailles métalliques (par opposition au nylon 33 ) comme un solide support pour la croissance des plantes et une méthode simple d'aération dans des conditions stériles en secouant pour soutenir la croissance microbienne. En outre, le système peut utiliser des grilles métalliques de différentes tailles pour soutenir la croissance des plantes, ce qui permet d'inclure diverses espèces de plantes sans limiter la largeur de leurs racines.
Dans le système de cocultivation hydroponique présentée ici, les plantes sont cultivées dans un système hydroponique stérile où les racines des plantes sécrètent des composés organiques soutenant la croissance des bactéries inoculées. Dans ce système de cocultivation, aucun produit chimique artificiel, tel que des hormones végétales, un élicateur de défense ou des produits chimiques inductifs de la virulence, est complété, ce qui reflète la cellule naturelle- signification de l'homéostasie lors des interactions plantes-microbes. Avec ce système de cocultivation hydroponique, il a été possible de déterminer simultanément l'expression des gènes dans le tissu racidique d'Arabidopsis thaliana Col-0 lors d'une infection par Agrobacterium , ainsi que l'activation des gènes Agrobacterium lors de la cocultivation avec Arabidopsis . Il a également été démontré que ce système est approprié pour étudier l'attachement d' Agrobacterium aux racines des plantes, ainsi que le profil de la racine de la plante, lors de la cocultivation (infection) avec Agrobacterium ( Figure 1 ).
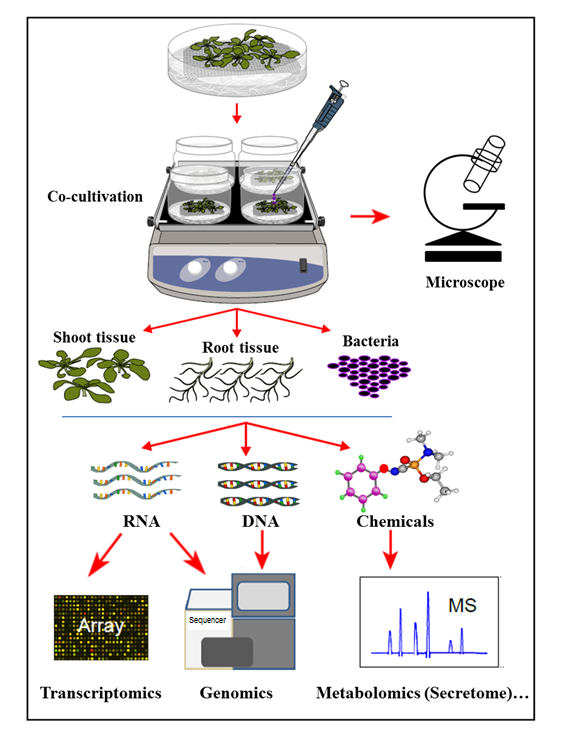
Figure 1: Vue d'ensemble du système de cocultivation hydroponique, avec des analyses d'échantillons. Les plantes sont cultivées au-dessus du maillage (pousses au-dessus du maillage), les racines étant immergées dans un milieu hydroponique qui est ensuite inoculé avec des bactéries fOu coculture. Les tissus végétaux et les bactéries sont ensuite séparés pour des extractions et des analyses simultanées. Ce chiffre a été modifié à partir de la référence 35 .
Protocol
Representative Results
Discussion
Compte tenu de la nature graduelle de la sécrétion racinaire, la concentration des produits chimiques induisant la virulence produite en planta et leurs effets sur les interactions dynamiques plantes-microbes se produisent dans les gradients spatiaux et temporels. Dans ce système de co-culture hydroponique, aucune phytohormone synthétique ou chimique qui induit une virulence microbienne ou des défenses végétales n'est complétée. En revanche, en utilisant des approches conventionnelles, l'ajout …
Declarações
The authors have nothing to disclose.
Acknowledgements
Nous tenons à remercier Brian Weselowski et Alexander W. Eastman pour leur aide et leur discussion utile. Nous tenons également à remercier les Drs. Eugene W. Nester, Lingrui Zhang, Haitao Shen, Yuhai Cui et Greg Thorn pour leur aide, des discussions utiles et une lecture critique du manuscrit. Cette recherche a été financée par Agriculture et Agroalimentaire Canada, Growing Forward-AgriFlex (RBPI numéro 2555) et Growing Forward II numéro de projet 1670, menée par les auteurs dans le cadre de leurs tâches. Cette étude a également été financée en partie par la subvention de découverte RGPIN-2015-06052 de recherche en sciences naturelles et en génie du Canada (CRSNG) décerné à ZC Yuan.
Materials
| plant seeds (Arabidopsis thaliana Col-0) | Arabidopsis Biological Resource Centre | CS7000 | https://abrc.osu.edu/order-stocks |
| bacteria (Agrobacterium tumefaciens C58) | University of Washington | N/A | |
| labeled bacteria | in-house | optional, depends on downstream analyses | |
| vortex | (various) | ||
| microcentrifuge tubes | (various) | ||
| microcentrifuge | (various) | ||
| 5% sodium hypochlorite | (various) | ||
| double distilled water | (various) | ||
| autoclave | (various) | ||
| micropipette | (various) | ||
| 70 % ethanol | (various) | ||
| Murashige and Skoog (MS) basal salts | Sigma-Aldrich | M5524 | |
| sucrose | (various) | ||
| MES | (various) | ||
| B5 vitamin mix | Sigma-Aldrich | G1019 | |
| phytoagar | (various) | ||
| deep Petri dishes | (various) | ||
| stainless steel mesh | Ferrier Wire Goods Company Ltd | N/A | grade: 304; mesh count: 40 × 40; wire DIA: 0.01 |
| micropore tape, 1 inch | 3M | 1530-1 | |
| diurnal growth chamber | (various) | ||
| cylindrical glass tanks, 100 × 80 mm | Pyrex | 3250 | other sizes can be used, in which case liquid content may need adjustment |
| flow hood | (various) | ||
| forcepts | (various) | ||
| yeast extract | (various) | ||
| tryptone | (various) | ||
| MgSO4 | (various) | ||
| shaking incubator | (various) | ||
| spectrophotometer | (various) | ||
| NaCl | (various) | ||
| shaker | (various) | ||
| scissors | (various) | optional, depends on downstream analyses | |
| fluorescence microscope | (various) | optional, depends on downstream analyses | |
| microscope slides and cover slips | (various) | optional, depends on downstream analyses | |
| nail polish | (various) | optional, depends on downstream analyses | |
| Bacterial RNA extraction kit | (various) | optional, depends on downstream analyses | |
| plant RNA extraction kit (RNeasy Plant Mini Kit) | Qiagen | 74903 or 74904 | optional, depends on downstream analyses |
| material and equipment for RT-qPCR | (various) | optional, depends on downstream analyses | |
| material and equipment for microarray analysis | (various) | optional, depends on downstream analyses | |
| liquid nitrogen | (various) | optional, depends on downstream analyses | |
| mortar and pestle | (various) | optional, depends on downstream analyses | |
| 0.2 µm pore filter | (various) | optional, depends on downstream analyses | |
| 50 mL conical tubes | (various) | optional, depends on downstream analyses | |
| freeze dryer | (various) | optional, depends on downstream analyses | |
| sealable test tubes | (various) | optional, depends on downstream analyses | |
| ethyl acetate | (various) | optional, depends on downstream analyses | |
| nitrogen gas | (various) | optional, depends on downstream analyses | |
| material and equipment for HPLC | (various) | optional, depends on downstream analyses | |
| material and equipment for ESI-TOF-MS | (various) | optional, depends on downstream analyses |
Referências
- Lambers, H., Mougel, C., Jaillard, B., Hinsinger, P. Plant-microbe-soil interactions in the rhizosphere: An evolutionary perspective. Plant Soil. 321 (1), 83-115 (2009).
- Philippot, L., Raaijmakers, J. M., Lemanceau, P., vander Putten, W. H. Going back to the roots: The microbial ecology of the rhizosphere. Nat. Rev. Microbiol. 11 (11), 789-799 (2013).
- Somers, E., Vanderleyden, J., Srinivasan, M. Rhizosphere bacterial signalling: A Love Parade beneath our feet. Crit. Rev. Microbiol. 30 (4), 205-240 (2004).
- Barah, P., Winge, P., Kusnierczyk, A., Tran, D. H., Bones, A. M. Molecular signatures in Arabidopsis thaliana in response to insect attack and bacterial infection. PLoS ONE. 8 (3), (2013).
- Zhang, J., Zhou, J. -. M. Plant immunity triggered by microbial molecular signatures. Mol. Plant. 3 (5), 783-793 (2010).
- Paterson, E., Gebbing, T., Abel, C., Sim, A., Telfer, G. Rhizodeposition shapes rhizosphere microbial community structure in organic soil. New Phytol. 173 (3), 600-610 (2006).
- Hartmann, A., Schmid, M., van Tuinen, D., Berg, G. Plant-driven selection of microbes. Plant Soil. 321 (1-2), 235-257 (2008).
- Micallef, S. A., Shiaris, M. P., Colon-Carmona, A. Influence of Arabidopsis thaliana accessions on rhizobacterial communities and natural variation in root exudates. J. Exp. Bot. 60 (6), 1729-1742 (2009).
- Burr, T., Otten, L. Crown gall of grape: Biology and disease management. Annu. Rev. Phytopathol. 37, 53-80 (2001).
- Clough, S. J., Bent, A. F. Floral dip: A simplified method for Agrobacterium-mediated transformation of Arabidopsis thaliana. Plant J. 16 (6), 735-743 (1998).
- Binns, A. N., Costantino, P., Spaink, H. P., Kondorosi, A., Hooykaas, P. J. J. The Agrobacterium oncogenes. The Rhizobiaceae. , (1998).
- Gheysen, G., Angenon, G., Van Montagu, M., Lindsey, K. Agrobacterium-mediated plant transformation: a scientifically intriguing story with significant applications. Transgenic Plant Research. , (1998).
- Valvekens, D., Von Montagu, M. V., Van Lijsebettens, M. Agrobacterium tumefaciens-mediated transformation of Arabidopsis thaliana root explants by using kanamycin selection. Proc. Natl. Acad. Sci. USA. 85 (15), 5536-5540 (1988).
- Krysan, P. J., Young, J. C., Sussman, M. R. T-DNA as an insertional mutagen in Arabidopsis. Plant Cell. 11 (12), 2283 (1999).
- Chilton, M. D., et al. Stable incorporation of plasmid DNA into higher plant cells: The molecular basis of crown gall tumorigenesis. Cell. 11 (2), 263-271 (1977).
- Gelvin, S. B. Agrobacterium in the genomics age. Plant Physiol. 150 (4), 1665-1676 (2009).
- Subramoni, S., Nathoo, N., Klimov, E., Yuan, Z. -. C. Agrobacterium tumefaciens responses to plant-derived signaling molecules. Front. Plant Sci. 5, 322 (2014).
- Yuan, Z., Liu, P., Saenkham, P., Kerr, K., Nester, E. W. Transcriptome profiling and functional analysis of Agrobacterium tumefaciens reveals a general conserved response to acidic conditions (pH 5.5) and a complex acid-mediated signaling involved in Agrobacterium-plant interactions. J. Bacteriol. 190 (2), 494-507 (2008).
- Stachel, S. E., Messens, E., Van Montagu, M., Zambryski, P. Identification of the signal molecules produced by wounded plant cells that activate T-DNA transfer in Agrobacterium tumefaciens. Nature. 318 (6047), 624-629 (1985).
- Memelink, J., de Pater, B. S., Hoge, J. H. C., Schilperoort, R. A. T-DNA hormone biosynthetic genes: Phytohormones and gene expression in plants. Dev. Genet. 8 (5-6), 321-337 (1987).
- Yuan, Z. -. C., et al. The plant signal salicylic acid shuts down expression of the vir regulon and activates quormone-quenching genes in Agrobacterium. Proc. Natl. Acad. Sci. USA. 104 (28), 11790-11795 (2007).
- Yuan, Z. -. C., Haudecoeur, E., Faure, D., Kerr, K. F., Nester, E. W. Comparative transcriptome analysis of Agrobacterium tumefaciens in response to plant signal salicylic acid, indole-3-acetic acid and γ-amino butyric acid reveals signalling cross-talk and Agrobacterium-plant co-evolution. Cell. Microbiol. 10 (11), 2339-2354 (2008).
- Li, P. L., Farrand, S. K. The replicator of the nopaline-type Ti plasmid pTiC58 is a member of the repABC family and is influenced by the TraR-dependent quorum-sensing regulatory system. J. Bacteriol. 182 (1), 179-188 (2000).
- Atkinson, M. M., Huang, J., Knopp, J. A. Hypersensitivity of suspension-cultured tobacco cells to pathogenic bacteria. Phytopathology. 75 (11), 1270-1274 (1985).
- Veena, ., Jiang, H., Doerge, R. W., Gelvin, S. B. Transfer of T-DNA and Vir proteins to plant cells by Agrobacterium tumefaciens induces expression of host genes involved in mediating transformation and suppresses host defense gene expression. Plant J. 35 (2), 219-236 (2003).
- León, J., Rojo, E., Sanchez-Serrano, J. J. Wound signalling in plants. J. Exp. Bot. 52 (354), 1-9 (2001).
- Ditt, R. F., Kerr, K. F., de Figueiredo, P., Delrow, J., Comai, L., Nester, E. W. The Arabidopsis thaliana transcriptome in response to Agrobacterium tumefaciens. Mol. Plant Microbe In. 19 (6), 665-681 (2006).
- Mandimba, G., Heulin, T., Bally, R., Guckert, A., Balandreau, J. Chemotaxis of free-living nitrogen-fixing bacteria towards maize mucilage. Plant Soil. 90 (1-3), 129-139 (1986).
- Rudrappa, T., Czymmek, K. J., Pare, P. W., Bais, H. P. Root-secreted malic acid recruits beneficial soil bacteria. Plant Physiol. 148 (3), 1547-1556 (2008).
- Lee, C. W., et al. Agrobacterium tumefaciens promotes tumor induction by modulating pathogen defense in Arabidopsis thaliana. Plant Cell. 21 (9), 2948-2962 (2009).
- Reymond, P., Weber, H., Damond, M., Farmer, E. E. Differential gene expression in response to mechanical wounding and insect feeding in Arabidopsis. Plant Cell. 12 (5), 707-720 (2000).
- Matthysse, A. G. Attachment of Agrobacterium to plant surfaces. Front. Plant Sci. 5, 252 (2014).
- Alatorre-Cobos, F., et al. An improved, low-cost, hydroponic system for growing Arabidopsis and other plant species under aseptic conditions. BMC Plant Biol. 14 (1), 69 (2014).
- Conn, S. J., et al. Protocol: Optimising hydroponic growth systems for nutritional and physiological analysis of Arabidopsis thaliana and other plants. Plant Methods. 9 (1), 4 (2013).
- Nathoo, N. . Identification of putative plant defense genes using a novel hydroponic co-cultivation technique for studying plant-pathogen interaction. , (2015).
- Shaner, N. C., Campbell, R. E., Steinbach, P. A., Giepmans, B. N., Palmer, A. E., Tsien, R. Y. Improved monomeric red, orange and yellow fluorescent proteins derived from Discosoma sp. red fluorescent protein. Nat. Biotechnol. 22 (12), 1567-1572 (2004).
- Wise, A. A., Liu, H., Binns, A. N. Nucleic acid extraction from Agrobacterium strains. Methods Mol. Bio. 343, 67-76 (2006).
- Nolan, T., Hands, R. E., Bustin, S. A. Quantification of mRNA using real-time RT-PCR. Nat. Protoc. 1 (3), 1559-1582 (2006).
- Salter, M. G., Conlon, H. E. Extraction of plant RNA. Methods Mol. Bio. 362, 309-314 (2007).
- Bao, Y., Wang, S., Yang, X., Li, T., Xia, Y., Meng, X. Metabolomic study of the intervention effects of Shuihonghuazi Formula, a Traditional Chinese Medicinal formulae, on hepatocellular carcinoma (HCC) rats using performance HPLC/ESI-TOF-MS. J. Ethnopharmacol. 198, 468-478 (2017).
- Korves, T. M., Bergelson, J. A developmental response to pathogen infection in Arabidopsis. Plant Physiol. 133 (1), 339-347 (2003).
- Lyons, R., Rusu, A., Stiller, J., Powell, J., Manners, J. M., Kazan, K. Investigating the association between flowering time and defense in the Arabidopsis thaliana-Fusarium oxysporum interaction. PLoS ONE. 10 (6), e0127699 (2015).
- Badri, D. V., Weir, T. L., vander Lelie, D., Vivanco, J. M. Rhizosphere chemical dialogues: Plant-microbe interactions. Curr. Opin. Biotechnol. 20 (6), 642-650 (2009).
- Baerson, S. R., et al. Detoxification and transcriptome response in Arabidopsis seedlings exposed to the allelochemical benzoxazolin-2(3H)-one. J. Biol. Chem. 280 (23), 21867-21881 (2005).

