Quantificação de microrganismos em baixas concentrações, utilizando microscopia holográfica Digital (DHM)
Summary
Microscopia holográfica digital (DHM) é uma técnica volumétrica que permite amostras de imagens realizadas 50-100x mais espesso que brightfield microscopia em resolução comparável, com o foco de pós-processamento. Aqui DHM é usado para identificar, contando e acompanhamento de microorganismos em muito baixas densidades e em comparação com medições de densidade óptica, contagem de placa e contagem direta.
Abstract
Com a detecção e contagem de amostras bacterianas esparsas tem muitas aplicações na comida, bebidas e indústrias de processamento farmacêutico, em diagnósticos médicos e para a detecção de vida por missões robóticas para outros planetas e luas do sistema solar. Atualmente, esparsas amostras bacterianas são contadas por microscopia de chapeamento ou epifluorescência de cultura. Placas de cultura exigem longo tempo de incubação (dias ou semanas) e microscopia de epifluorescência requer mancha extensa e concentração da amostra. Aqui, demonstramos como usar fora do eixo microscopia holográfica digital (DHM) para enumerar as bactérias em culturas muito diluídas (100-104 células/mL). Em primeiro lugar, a construção da DHM personalizado é discutida, juntamente com instruções detalhadas sobre a criação de um instrumento de baixo custo. São discutidos os princípios da holografia, e um modelo estatístico é usado para estimar quanto tempo vídeos devem ser para detectar células, com base nas características desempenho óptico do instrumento e a concentração da solução bacteriana (tabela 2) . A detecção de células em 105, 104, 103e 100 células/mL é demonstrada em tempo real usando hologramas un-reconstruídos. Reconstrução de imagens de amplitude e fase é demonstrada usando um pacote de software open-source.
Introduction
Determinação da precisa contagens bacterianas em amostras muito diluídas é crucial em muitas aplicações: alguns exemplos são a água e a comida qualidade análise1,2,3; deteção de patógenos no sangue, líquido cefalorraquidiano ou escarro4,5; produção de produtos farmacêuticos, incluindo água estéril6; e análise ambiental da Comunidade em ambientes oligotróficos como o aberto oceano e sedimentos7,8,9. Existe também uma crescente interesse para detecção de possível vida microbiana existente nas luas geladas de Júpiter e Saturno, particularmente Europa10,11 e Enceladus12,13, 14, que são conhecidos por terem subsuperficiais oceanos líquidos. Porque nenhuma missão desde Viking em 1978 tem tentado encontrar existe vida em outro planeta, tem havido desenvolvimento limitado de tecnologias e instrumentos de identificação bacteriana e contando durante espaço missões15.
Os métodos tradicionais de contagem de placa encontrar apenas células viáveis, que podem representar uma minoria das espécies em cepas ambientais, às vezes < 1%16. Placas requerem dias ou semanas de incubação para o máximo de sucesso, dependendo da estirpe. Microscopia de epifluorescência substituiu, em larga medida, contagens das placas como o padrão ouro para enumeração microbiana rápida e exata. Corantes de fluorescentes nucleic-ácido-rotulagem como laranja de acridina, dicloridrato 6-diamidino-2-phenylindole (DAPI), SYBR Green ou 4 ‘ que se ligam aos ácidos nucleicos são os corantes típicos usados17,18,19 , apesar de muitos estudos utilizam indicadores fluorescentes de grama assinar20,21,22,23,24. Usando esses métodos sem etapas de pré-concentração leva à limites de detecção (LoDs) de ~ 105 células por mL. Melhorias no LoD são possíveis usando filtragem. Uma amostra de líquido é filtrada vácuo em uma membrana, geralmente em policarbonato e idealmente preto para reduzir a fundo. Baixo-fundo corantes tais como as manchas de DNA mencionadas acima podem ser aplicadas directamente para o filtro de25. Para contagem exata pelo olho, as células de5 ~ 10 são necessárias por filtro, o que significa que, para amostras mais diluídas do que 10 ~5 células por mL, volumes significativos de amostra devem ser recolhidas e filtradas. Dispositivos de varredura a laser foram desenvolvidos a fim de explorar sistematicamente todas as regiões do filtro e, assim, reduzir o número de células necessárias para a contagem, empurrando os limites de detecção até ~ 102 células por mL26. No entanto, estes não estão disponíveis na maioria dos laboratórios e exigem hardware sofisticado, assim como software que permitem a confirmação de perita que observou partículas é as bactérias e não os restos.
Para referência, adultos com sepse geralmente começam a mostrar sintomas de < 100 células/mL de sangue e bebês em < 10 células/mL. Uma recolha de sangue de um adulto leva 10 mL e de uma criança, 1ml. Métodos baseados em PCR são inibidos pela presença de humano e não-patogênicas flora DNA e pelos componentes do PCR-inibindo o sangue27,28. Apesar de uma variedade de técnicas emergentes, culturas permanecem o padrão ouro para o diagnóstico de infecções da corrente sanguínea, especialmente em zonas mais rurais ou nações em desenvolvimento. Detecção de vida em outros planetas, cálculos termodinâmicos podem estimar o orçamento de energia para a vida e, portanto, a biomassa possível esperada. 1 – 100 células/mL são esperados ser termodinamicamente razoável na Europa29. Pode ser facilmente visto destes números que a detecção de um pequeno número de células em grandes quantidades de solução aquosa é um importante problema sem solução.
Neste artigo, demonstramos a detecção de Serratia marcescens e Shewanella oneidensis (tipo selvagem e non-motile mutante) em concentrações de 105, 104, 10 e 100 células/mL usando um fora do eixo3 microscópio digital holográfico (DHM). A principal vantagem da DHM por microscopia de luz tradicional é a imagem simultânea de um volume de amostra espesso em alta resolução — nessa implementação, a câmara de amostra foi de 0,8 mm de espessura. Estas câmaras de amostra foram construídas pelo macio-litografia de polidimetilsiloxano (PDMS) de um molde de alumínio de precisão usinadas com uma tolerância de ± 50 µm. Isto representa uma melhoria de aproximadamente 100 vezes em profundidade de campo através de microscopia de luz de alta potência. DHM também fornece informações de fase quantitativa, permitindo a medição do comprimento do percurso óptico (produto do índice de refração e espessura). DHM e técnicas semelhantes têm sido utilizadas para monitoramento de bacteriano e ciclo celular de levedura e cálculo de massa seca bacteriana30,31,32; diferenças de dispersão mesmo podem ser usadas para diferenciar estirpes bacterianas33.
O instrumento que usamos é costume-construído especificamente para uso com microorganismos, como publicado anteriormente34,,35, e seu design e construção são demonstrados e discutidos. Soluções aquosas são continuamente fornecidas a uma câmara de amostra de volume µ l 0,25 através de bomba de seringa; a taxa de fluxo é determinada pela taxa de quadros da câmera para assegurar imagens volume da amostra inteira. Um cálculo estatístico prevê que o número de volumes de amostra que deve ser fotografada a fim de detectar um número significativo de células em uma dada concentração.
Para aplicações de célula-deteção, reconstrução de hologramas em amplitude e fase de imagens não era exigida; análise foi realizada no holograma cru. Isso economiza espaço em disco e recursos computacionais significativos: um holograma de 500 Mb vídeo será 1 a 2 Tb quando reconstruído. No entanto, podemos discutir reconstrução através da profundidade da amostra para confirmar que os hologramas representam a espécie desejada. Uma característica importante da DHM é sua capacidade de monitorar tanto a intensidade e a fase das imagens. Organismos que são quase transparentes em intensidade (como a maioria das células biológicas) aparecem claramente em fase. Como é uma técnica livre de rótulo, não são utilizados corantes. Este é um umdvantage para aplicações de voo de espaço possível, desde que corantes podem não sobreviver as condições de uma missão e — mais importante — não pode ser presumida a trabalhar com organismos extraterrestres, que não podem usar o DNA ou RNA para codificação. Também é uma vantagem para o trabalho em ambientes extremos, tais como o Ártico e Antártico, onde corantes podem ser difíceis de trazer para o local remoto e podem degradar após armazenamento. Reconstrução de imagens em fase e amplitude é executada usando um pacote de software open-source que disponibilizamos no GitHub (SHAMPOO) ou usando o ImageJ.
Protocol
Representative Results
Discussion
Reconstrução numérica de hologramas: para a reconstrução numérica de hologramas, é usado o método do espectro angular (ASM). Isso envolve a convolução do holograma com a função Green para a DHM. O wavefront complexo da imagem em um determinado plano focal pode ser calculada utilizando o teorema da convolução Fourier como segue:
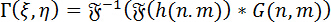 (1)
(1)
Onde <img alt="Equation 3" src="/files/f…
Declarações
The authors have nothing to disclose.
Acknowledgements
Os autores reconhecem o Gordon e Betty Moore Foundation Grants 4037 e 4038 ao Instituto de tecnologia da Califórnia, para o financiamento deste trabalho.
Materials
| Bacto Yeast | BD Biosciences | 212750 | |
| Bacto Tryptone | BD Biosciences | 211705 | |
| Sodium chloride | Sigma-Aldrich | 7710 | Many options for purchase |
| Bacto Agar | BD Biosciences | 214010 | |
| 10 cm Petri dishes | VWR | 10053-704 | |
| 15 mL culture tubes | Falcon (Corning Life Sciences) | 352002 | Loose-capped |
| Petroff-Hauser chamber | Electron Microscopy Sciences | 3920 | |
| 10 mL syringes | BD Biosciences | 309604 | Luer-Lok tip not necessary |
| Male Luer to 1/16” barbed fitting | McMaster-Carr | 51525K291 | |
| Autoclavable 1/16” ID PVC tubing for flow | Nalgene | 8000-0004 | Sold by length, purchase accordingly |
| Syringe pump | Harvard Apparatus | PHD 2000 | |
| Sample Chamber | Custom | n/a | See Materials Section |
| Holographic Microscope | Custom | n/a | See Wallace et al. |
| Open-source software | Custom | https://github.com/bmorris3/shampoo |
Referências
- Akineden, O., Weirich, S., Abdulmawjood, A., Failing, K., Buelte, M. Application of a Fluorescence Microscopy Technique for Detecting Viable Mycobacterium avium ssp. paratuberculosis Cells in Milk. Food Anal. Methods. 8, 499-506 (2015).
- Deibl, J., Paulsen, P., Bauer, F. Rapid enumeration of total aerobic microbial counts on meat and in meat products by means of the direct epifluorescence filter technique. Wien Tierarztl Monat. 85, 327-333 (1998).
- Huang, J., Li, Y., Slavik, M. F., Tao, Y., Huff, G. R. Identification and enumeration of Salmonella on sample slides of poultry carcass wash water using image analysis with fluorescent microscopy. Transactions of the Asae. 42, 267-273 (1999).
- Durtschi, J. D., et al. Increased sensitivity of bacterial detection in cerebrospinal fluid by fluorescent staining on low-fluorescence membrane filters. J Med Microbiol. 54, 843-850 (2005).
- Panicker, R. O., Soman, B., Saini, G., Rajan, J. A Review of Automatic Methods Based on Image Processing Techniques for Tuberculosis Detection from Microscopic Sputum Smear Images. J Med Syst. 40, (2016).
- Riepl, M., et al. Applicability of solid-phase cytometry and epifluorescence microscopy for rapid assessment of the microbiological quality of dialysis water. Nephrol Dial Transplant. 26, 3640-3645 (2011).
- Hwang, C. Y., Cho, B. C. Virus-infected bacteria in oligotrophic open waters of the East Sea, Korea. Aquat Microb Ecol. 30, 1-9 (2002).
- Kepner, R. L., Pratt, J. R. Use of Fluorochromes for Direct Enumeration of Total Bacteria in Environmental-Samples – Past and Present. Microbiol Rev. 58, 603-615 (1994).
- Queric, N. V., Soltwedel, T., Arntz, W. E. Application of a rapid direct viable count method to deep-sea sediment bacteria. J Microbiol Meth. 57, 351-367 (2004).
- Prieto-Ballesteros, O., Vorobyova, E., Parro, V., Rodriguez Manfredi, J. A., Gomez, F. Strategies for detection of putative life on Europa. Adv Space Res. 48, 678-688 (2011).
- Gleeson, D. F., et al. Biosignature Detection at an Arctic Analog to Europa. Astrobiology. 12, 135-150 (2012).
- Carr, C. E., Zuber, M. T., Ruvkun, G., Ieee, . 2013 Ieee Aerospace Conference IEEE Aerospace Conference Proceedings. , (2013).
- Konstantinidis, K., et al. A lander mission to probe subglacial water on Saturn’s moon Enceladus for life. Acta Astronautica. 106, 63-89 (2015).
- McKay, C. P., Anbar, A. D., Porco, C., Tsou, P. Follow the Plume: The Habitability of Enceladus. Astrobiology. 14, 352-355 (2014).
- Hoover, R. B., Hoover, R. B., Levin, G. V., Rozanov, A. Y., Wickramasinghe, N. C. . Instruments, Methods, and Missions for Astrobiology Xvii. , (2015).
- Handelsman, J. Metagenomics: application of genomics to uncultured microorganisms. Microbiol Mol Biol Rev: MMBR. 68, 669-685 (2004).
- Lebaron, P., Parthuisot, N., Catala, P. Comparison of blue nucleic acid dyes for flow cytometric enumeration of bacteria in aquatic systems. Appl Environ Microbiol. 64, 1725-1730 (1998).
- Marie, D., Partensky, F., Jacquet, S., Vaulot, D. Enumeration and cell cycle analysis of natural populations of marine picoplankton by flow cytometry using the nucleic acid stain SYBR Green I. Appl Environ Microbiol. 63, 186-193 (1997).
- Noble, R. T., Fuhrman, J. A. Use of SYBR Green I for rapid epifluorescence counts of marine viruses and bacteria. Aquat Microb Ecol. 14, 113-118 (1998).
- Forster, S., Snape, J. R., Lappin-Scott, H. M., Porter, J. Simultaneous fluorescent gram staining and activity assessment of activated sludge bacteria. Appl Environ Microbiol. 68, 4772-4779 (2002).
- Lauer, B. A., Reller, L. B., Mirrett, S. Comparison Of Acridine-Orange And Gram Stains For Detection Of Microorganisms In Cerebrospinal-Fluid And Other Clinical Specimens. J Clin Microbiol. 14, 201-205 (1981).
- Mason, D. J., Shanmuganathan, S., Mortimer, F. C., Gant, V. A. A fluorescent gram stain for flow cytometry and epifluorescence microscopy. Appl Environ Microbiol. 64, 2681-2685 (1998).
- Saida, H., Ytow, N., Seki, H. Photometric application of the Gram stain method to characterize natural bacterial populations in aquatic environments. Appl Environ Microbiol. 64, 742-747 (1998).
- Sizemore, R. K., Caldwell, J. J., Kendrick, A. S. Alternate Gram Staining Technique Using A Fluorescent Lectin. Appl Environ Microbiol. 56, 2245-2247 (1990).
- Bitton, G., Dutton, R. J., Foran, J. A. New Rapid Technique For Counting Microorganisms Directly On Membrane Filters. Stain Technology. 58, 343-346 (1983).
- Broadaway, S. C., Barton, S. A., Pyle, B. H. Rapid staining and enumeration of small numbers of total bacteria in water by solid-phase laser cytometry. Appl Environ Microbiol. 69, 4272-4273 (2003).
- Yagupsky, P., Nolte, F. S. Quantitative aspects of septicemia. Clin Microbiol Rev. 3, 269-279 (1990).
- Kang, D. K., et al. Rapid detection of single bacteria in unprocessed blood using Integrated Comprehensive Droplet Digital Detection. Nat Commun. 5, 5427 (2014).
- Hand, K. P. . Report of the Europa Lander Science Definition Team. , (2017).
- Popescu, G., et al. Optical imaging of cell mass and growth dynamics. Am J Physiol-Cell Physiol. 295, C538-C544 (2008).
- Mir, M., et al. Optical measurement of cycle-dependent cell growth. Proc Natl Acad Sci U S A. 108, 13124-13129 (2011).
- Rappaz, B., et al. Noninvasive characterization of the fission yeast cell cycle by monitoring dry mass with digital holographic microscopy. J.Biomed Opt. 14, 034049 (2009).
- Jo, Y., et al. Label-free identification of individual bacteria using Fourier transform light scattering. Opt. Express. 23, 15792-15805 (2015).
- Wallace, J. K., et al. Robust, compact implementation of an off-axis digital holographic microscope. Opt. Express. 23, 17367-17378 (2015).
- Lindensmith, C. A., et al. A Submersible, Off-Axis Holographic Microscope for Detection of Microbial Motility and Morphology in Aqueous and Icy Environments. Plos One. 11, e0147700 (2016).
- Dumas, E. M., Ozenne, V., Mielke, R. E., Nadeau, J. L. Toxicity of CdTe Quantum Dots in Bacterial Strains. IEEE Trans. NanoBiosci. 8, 58-64 (2009).
- Frisk, A., Jyot, J., Arora, S. K., Ramphal, R. Identification and functional characterization of flgM, a gene encoding the anti-sigma 28 factor in Pseudomonas aeruginosa. J Bacteriol. 184, 1514-1521 (2002).
- Adler, J., Templeton, B. The effect of environmental conditions on the motility of Escherichia coli. Journal Gen Microbiol. 46, 175-184 (1967).
- Piedrahita-Quintero, P., Castaneda, R., Garcia-Sucerquia, J. Numerical wave propagation in Image J. Appl Opt. 54, 6410-6415 (2015).
- Schnars, U., Jüptner, W. P. Digital recording and numerical reconstruction of holograms. Meas. Sci. Technol. 13, R85 (2002).

