Agrégation de récepteur de Surface cellulaire microscopie confocale révèle par spectroscopie de corrélation d’Image
Summary
Anticorps qui se fixent à des récepteurs sur la surface de la cellule cible peuvent conférer conformation et altérations clusters. Ces changements dynamiques ont des implications pour caractériser le développement des médicaments dans les cellules cibles. Ce protocole utilise la microscopie confocale et spectroscopie de corrélation d’image par le biais de ImageJ/Fidji pour quantifier l’ampleur du récepteur clustering sur la surface de la cellule.
Abstract
Microscopie confocale fournit une méthodologie accessible pour capturer les interactions subcellulaires critiques pour la caractérisation et le développement d’agents précliniques marquées avec des sondes fluorescentes. Avec des avancées récentes en anticorps cytotoxique vecteurs, comprendre les altérations induites par ces agents dans le domaine de l’agrégation des récepteurs et l’internalisation est d’une importance cruciale. Ce protocole s’appuie sur la méthodologie bien établie de l’immunocytochimie fluorescent et la distribution de Fidji open source d’ImageJ, avec son autocorrélation intégrée et des fonctions mathématiques d’image, pour effectuer la corrélation d’images spatiales spectroscopie (ICS). Ce protocole quantitates l’intensité de fluorescence des récepteurs étiquetées en fonction de la zone de la poutre du microscope confocal. Ceci fournit une mesure quantitative de l’état d’agrégation de molécule cible sur la surface de la cellule. Cette méthodologie est axée sur la caractérisation des cellules statiques avec un potentiel d’enquêtes temporelle de l’agrégation des récepteurs. Ce protocole présente une méthodologie accessible pour fournir la quantification du regroupement des événements qui se produisent à la surface cellulaire, utilisant bien mis en place des techniques et des appareils d’imagerie non spécialisés.
Introduction
Le développement d’anticorps thérapeutiques a montré un succès remarquable dans le traitement des tumeurs multiples types1. Les progrès récents des conjugués anticorps-médicament (ADC) comme les mécanismes de prestation pour les composés cytotoxiques a élargi les conditions requises pour comprendre la dynamique des interactions entre les anticorps : récepteurs à la cellule de surface2. Après le succès de ciblage d’un anticorps dirigé contre le récepteur de surface cellulaire, ces complexes peuvent induire des patrons d’agrégation semblables à celles observées dans d’interactions ligand : anticorps3. Altérations dans l’agrégation des récepteurs peuvent induire des modifications de la membrane et le résultat dans l’internalisation du récepteur et son retrait de la surface de la cellule. Dans le contexte d’un conjugué d’anticorps-médicament, ce processus libère ensuite la charge cytotoxique dans les endosomes intériorisée et par la suite le cytoplasme, entraînant dans la mort cellulaire efficace.
Microscopie confocale a fourni un moyen efficace de visualiser ces interactions importantes des anticorps et leur cible les récepteurs4. Pour explorer les changements de l’agrégation de la molécule cible à la surface cellulaire ce protocole utilise le post-traitement des images de microscopie confocale par une image spatiale corrélation spectroscopie (ICS) technique 5,6,7 .
La Fondation de la spectroscopie de corrélation d’image est l’observation que les fluctuations d’intensité de fluorescence spatiale partagent une relation à l’état de densité et de regroupement des structures étiquetées. Cette relation est établie suivant le calcul d’une fonction d’autocorrélation spatiale d’une image capturée5.
Toutes les variantes de la spectroscopie de corrélation d’image exigent le calcul d’une autocorrélation d’image. Il est suivi en ajustant cette fonction à une courbe gaussienne bidimensionnelle pour extraire des paramètres d’État agrégation quantitative contenue dans l’image. En termes simples le calcul d’une autocorrélation d’image consiste à comparer toutes les paires possibles pixel contenu dans une image et en calculant la probabilité que les deux tout aussi aussi brillant que l’autre. C’est visualisé en fonction de la distance et directions de pixel séparation8.
Le cadre théorique pour la spectroscopie de corrélation d’image a été établi et défini par Petersen et Wiseman et coll.. 5 , 6. dans le présent protocole, les calculs de l’autocorrélation sont effectuées en Fidji/ImageJ comme une application de tableur, la base de la fonction d’autocorrélation spatiale intensité fluctuation peut être décrit comme (Eq 1):
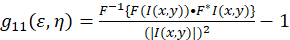
où F représente la transformation de Fourier ; F−1 l’inverse de Fourier transform ; F * son conjugué complexe ; et le décalage spatial variables ε et η. Dans ICS spatiale, comme décrit dans le présent protocole, la fonction d’autocorrélation peut être calculée à l’aide d’un 2D fast Fourier transform algorithm7,9. L’autocorrélation à zéro séparation spatiale, autrement connue comme zéro lag, g11(0,0), fournit l’inverse nombre moyen de particules présentes par surface de faisceau du microscope. Il peut être obtenu en installant la fonction d’autocorrélation spatiale d’une fonction gaussienne bidimensionnelle (Eq 2):
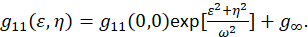
Comme les pixels capturés dans une image sont contenus dans une zone définie et ces mesures ne s’étendent pas à l’infini, le terme g∞ est utilisé comme une compensation pour tenir compte des corrélations spatiales à longue distance, contenues dans l’image. Pour les agrégats moléculaires taille, ω est la fonction de point-propagation du microscope et décrite par la pleine largeur à moitié-maximum de la fonction d’autocorrélation spatiale. La zone comprise dans la fonction de point de répandre de l’instrument peut être étalonnée à l’aide de perles fluorescentes sous résolution.
Pour l’image corrélation spectroscopie protocole décrit ci-après, l’autocorrélation et fonctions mathématiques doit remplir l’ICS sont effectuées à l’aide de la plate-forme d’imagerie-traitement de l’open source, Fidji10, une distribution de la ImageJ programme11,12. Fidji/ImageJ utilise le Fourier rapide préinstallée dans la fonction FFT Math. Cette fonction permet de réduire le temps de calcul requis de ce calcul en réduisant l’éventail des données par un facteur de deux dans chaque dimension13. Comme la fonction d’autocorrélation 2D est approximativement symétrique en x, axe y, une parcelle de profil de ligne unique à travers l’image de l’autocorrélation peut être utilisée pour mesurer l’autocorrélation brute en fonction du GAL spatiale. N’importe quel bruit de zéro lag est supprimé avant le calcul plus loin, à l’amplitude résultante de l’autocorrélation (valeur de crête, g(0)T) corrigé pour le fond avec l’expression (Eq 3):
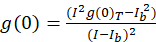
où j’aib est l’intensité moyenne d’une région de fond, à l’exclusion de la cellule. La densité de cluster, ou la densité des objets fluorescents, est définie par (Eq 4):
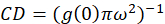
Dans le protocole décrit ci-après, nous avons plus simplement le calcul de la densité de cluster (CD), avec l’hypothèse fondée sur l’observation selon laquelle une fonction d’autocorrélation normalisée décroîtra à une valeur approchante 1.0 avec l’augmentation de décalage spatial. Avec un décalage spatial maximal, il y a non plus aucune corrélation de fluorescence valeurs d’intensité et donc sans une corrélation les calculs dans cette région sont calcul de la valeur d’une intensité multipliée par l’intensité qui est ensuite divisée par le la place de cette intensité, qui, par définition, est égale à 1.0. Ainsi, la densité d’une fonction d’autocorrélation normalisée cluster peut être calculée en soustrayant 1.0 de la fonction d’autocorrélation normalisée avant de prendre sa réciproque (Eq 5):
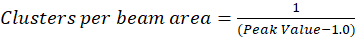
Étalonnage supplémentaire de la région de faisceau peut être effectuée afin de quantifier le nombre de clusters contenus dans le domaine de la fonction point de répandre de l’instrument. Cet étalonnage doit être effectué en utilisant les mêmes conditions optiques utilisées lors de l’analyse de la spectroscopie de corrélation d’image.
Protocol
Representative Results
Discussion
La technique de spectroscopie de corrélation d’image (ICS) que nous décrivons dans ce protocole utilise des microscopes confocaux standards sans besoin de détecteurs spécialisés. La technique ICS décrite applique des méthodes immunocytochimie bien établi à fournir pour l’échantillonnage rapide de multiples conditions de traitement pour l’analyse statistique accrue. Cette méthodologie fait avec une légère réduction de précision absolue par rapport aux techniques alternatives molécule unique basé sur…
Declarações
The authors have nothing to disclose.
Acknowledgements
Les auteurs reconnaissent le soutien financier du NHMRC (1084178 de bourses et subventions 1087850, 1030469, 1075898 (AMS)), Cancer Australie, Ludwig Cancer, John T Reid Trusts, Cure Brain Cancer Foundation, La Trobe University et l’Agence du Cancer de Victoria. Financement du programme de soutien opérationnel Infrastructure fournie par le gouvernement de Victoria, l’Australie est également reconnu.
Materials
| Nunc Lab-Tek II Chambered Coverglass – 8 well | ThermoFisher Scientific | 155409 | |
| DPBS, no calcium, no magnesium | ThermoFisher Scientific | 14190144 | |
| Fetal Bovine Serum | ThermoFisher Scientific | 10099141 | |
| TrypLE Express Enzyme (1x), no phenol red | ThermoFisher Scientific | 12604021 | |
| Penicillin-Streptomycin (10,000 U/mL) | ThermoFisher Scientific | 15140122 | |
| GlutaMAX Supplement | ThermoFisher Scientific | 35050061 | |
| Goat anti-Human IgG (H+L) Cross-Adsorbed Secondary Antibody, Alexa Fluor 488 | ThermoFisher Scientific | A11013 | |
| TetraSpeck Fluorescent Microsphere Standards 0.1µm | ThermoFisher Scientific | T7279 | |
| Cetuximab | Merck Serono | 3023715501 | |
| Parafilm M 38mx100mm | Merck Millipore | BRND701605 | |
| 16% Paraformaldehyde (formaldehyde) aqueous solution | ProSciTech | C004 | |
| Recombinant Human EGF | R&D System | 236-EG |
Referências
- Scott, A. M., Wolchok, J. D., Old, L. J. Antibody therapy of cancer. Nature reviews. Cancer. 12 (4), 278-287 (2012).
- Parslow, A. C., Parakh, S., Lee, F. -. T., Gan, H., Scott, A. Antibody-Drug Conjugates for Cancer Therapy. Biomedicines. 4 (3), 14 (2016).
- Sorkin, A., Waters, C. M. Endocytosis of growth factor receptors. BioEssays. 15 (6), 375-382 (1993).
- Pawley, J. . Handbook of Biological Confocal Microscopy. , (2006).
- Petersen, N. O., Höddelius, P. L., Wiseman, P. W., Seger, O., Magnusson, K. E. Quantitation of membrane receptor distributions by image correlation spectroscopy: concept and application. Biophysical Journal. 65 (3), 1135-1146 (1993).
- Wiseman, P. W., Petersen, N. O. Image Correlation Spectroscopy. II. Optimization for Ultrasensitive Detection of Preexisting Platelet-Derived Growth Factor-β Receptor Oligomers on Intact Cells. Biophysical Journal. 76 (2), 963-977 (1999).
- Costantino, S., Comeau, J. W. D., Kolin, D. L., Wiseman, P. W. Accuracy and Dynamic Range of Spatial Image Correlation and Cross-Correlation Spectroscopy. Biophysical Journal. 89 (2), 1251-1260 (2005).
- Claire Robertson, S. C. G. Theory and practical recommendations for autocorrelation-based image correlation spectroscopy. Journal of Biomedical Optics. 17 (8), 080801 (2012).
- Ciccotosto, G. D., Kozer, N., Chow, T. T. Y., Chon, J. W. M., Clayton, A. H. A. Aggregation Distributions on Cells Determined by Photobleaching Image Correlation Spectroscopy. Biophysical Journal. 104 (5), 1056-1064 (2013).
- Schindelin, J., Arganda-Carreras, I., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Abràmoff, M. D., Magalhães, P. J., Ram, S. J. Image processing with ImageJ. Biophotonics International. 11 (7), 36-42 (2004).
- Collins, T. J. ImageJ for microscopy. BioTechniques. 43 (1 Suppl), 25-30 (2007).
- Rappaz, B., Wiseman, P. W. Image correlation spectroscopy for measurements of particle densities and colocalization. Current protocols in cell biology. , (2013).
- tferr/Scripts: BAR 1.5.1. Zenodo Available from: https://zenodo.org/record/495245 (2017)
- Elson, E. L. Fluorescence Correlation Spectroscopy: Past, Present, Future. Biophysical Journal. 101 (12), 2855-2870 (2011).
- Jares-Erijman, E. A., Jovin, T. M. Imaging molecular interactions in living cells by FRET microscopy. Current opinion in chemical biology. 10 (5), 409-416 (2006).
- Huang, B., Bates, M., Zhuang, X. Super-Resolution Fluorescence Microscopy. dx.doi.org.ez.library.latrobe.edu.au. 78 (1), 993-1016 (2009).
- Schermelleh, L., Heintzmann, R., Leonhardt, H. A guide to super-resolution fluorescence microscopy. The Journal of Cell Biology. 190 (2), 165-175 (2010).
- Nohe, A., Petersen, N. O. Image Correlation Spectroscopy. Sci. Signal. (417), pl7 (2007).

