Гель seq: Метод для подготовки библиотека одновременных секвенирования ДНК и РНК с помощью матрицы Гидрогель
Summary
Гель seq позволяет исследователям одновременно готовить библиотек для обоих ДНК – и РНК seq в незначительной добавленной стоимости, начиная от 100-1000 ячеек с помощью простой гидрогеля устройства. Этот документ представляет детальный подход для изготовления устройства, а также биологические протокол для создания парных библиотек.
Abstract
В за последние пять лет был достигнут только возможность усилить и последовательности ДНК или РНК от небольшой начальный образцов. К сожалению стандартные протоколы для генерации геномной или транскриптомики библиотеки являются несовместимыми и исследователи должны выбрать, следует ли последовательность ДНК или РНК для конкретной выборки. Гель seq решает эту проблему, позволяя исследователей подготовить одновременно библиотек для ДНК и РНК, начиная с 100-1000 ячеек с помощью простой гидрогеля устройства. Этот документ представляет детальный подход для изготовления устройства, а также биологические протокол для создания парных библиотек. Мы разработали гель seq, так что он легко может осуществляться другими исследователями; Многие лаборатории генетики уже есть необходимое оборудование, чтобы воспроизвести изготовление гель seq устройства. Наш протокол использует часто используемые наборы для обоих целом Стенограмма амплификации (WTA) и подготовка библиотеки, которые также могут быть знакомы исследователей уже разбираются в генерации геномных и транскриптомики библиотеки. Наш подход позволяет исследователям привнести власть ДНК и РНК последовательности на одном образце без разделения и с незначительной добавленной стоимости.
Introduction
Следующее поколение виртуализации (НГС) оказывает глубокое влияние на способ исследования генетики. Там, где исследователи раз сосредоточено на Секвенирование генома всего видов, это теперь возможно последовательности генома единый опухоли или даже одну ячейку в одном эксперименте. 1 NGS также сделал экономически эффективным для последовательности РНК стенограммы, нашли в ячейке, сбор данных, известный как транскриптом. В за последние пять лет был достигнут только возможность усилить и последовательности ДНК или РНК от небольшой начальный образцов. 2 , 3 , К сожалению, 4 стандартные протоколы несовместимы и исследователи должны выбрать, следует ли последовательность ДНК или РНК для данного образца. Когда образец начала достаточно велик, он может разделить пополам. В меньших масштабах однако потери материала за счет расщепления образцов может повлиять на качество библиотеки, и объединения образцов может среднем, интересные вариации между ячейками. 5 Кроме того, исследователи все больше заинтересованы в изучении образцов, которые нельзя разделить, например отдельные ячейки или биопсии малых гетерогенных опухоли. 6
Для решения этой проблемы, три были недавно разработаны протоколы для последовательностей ДНК и РНК из того же образца начала: гель seq7,8G и T-seq и DR-seq9. Эта статья представляет подробный протокол для гель seq, который может использоваться для одновременно создавать библиотеки ДНК и РНК из всего лишь 100 клеток в незначительной добавленной стоимости. Роман аспект гель seq является возможность отделить ДНК и РНК, исключительно на основе размера с использованием недорогих гидрогеля матрицы. Основные инновации гель-Seq протокола является физическое разделение ДНК из РНК. Это разделение достигается электрофорезно используя комбинацию полиакриламида мембран, которые используют преимущества размер различия между этими молекулами. Положить эти различия размер в контексте, рассмотрим как отражаются ДНК и РНК: в то время как ДНК существует на шкале микрон и могут быть просмотрены с помощью традиционных Микроскопы, РНК существует в нанометровом масштабе и должны отражаться с использованием сложных методов например крио электрон микроскопия. 10
Подход к отделять ДНК и РНК в этот протокол показан на рисунке 1. Левая панель показывает, что ДНК и РНК Свободный плавающий в растворе вблизи мембраны. Когда электрическое поле применяется, как показано в правой панели, ДНК и РНК испытывают электрофоретические сила, которая побуждает миграции через мембрану. Путем настройки свойства мембраны, мы создали полупроницаемую мембрану, разделяющую ДНК от РНК. Молекулы ДНК толкнул мембраны, но запутаться на границе из-за их больших размеров. Малые молекулы РНК, с другой стороны, можно перенастроить и ткут свой путь через мембрану. Этот процесс, известный как reptation, подобно тому, как змея движется через траву. В конечном итоге эти молекулы РНК остановлены мембраной второй, высокой плотности, который является слишком сложным для даже меньше полимеров (> 200 пар оснований) чтобы пробираться через. После того, как физически отделены, ДНК и РНК можно восстановить и обработаны для получения информации о геноме и транскриптом. Хотя мы можем отделить ДНК и РНК, мы нашли, что лучшие результаты получаются, если РНК обратной транскрипции cDNA перед разлукой. Гибридов cDNA/РНК более стабильны, чем РНК только и может еще пройти через low-density мембраны.
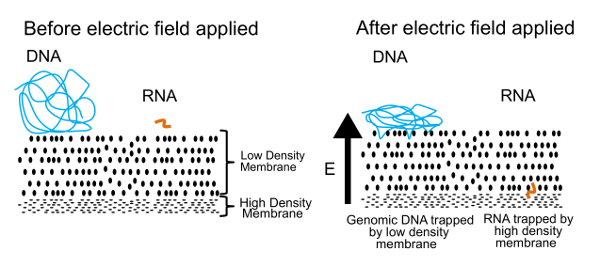
Рисунок 1 . Гель seq принцип. Основной принцип, используемый для физически отделить ДНК и РНК. В прикладной электрического поля малых молекул РНК мигрируют через low-density мембраны, но большие молекулы ДНК оказываются на поверхности. Этот показатель был воспроизведен из ссылка 7 с разрешения Королевского общества химии. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
Этот документ подробно описывает, как изготовление гель seq устройства и биологических протокол для создания паре библиотеки ДНК и РНК. Обзор как показано на рисунке 2. Устройство изготавливается путем наслаивать три разные плотности полиакриламидных гелей друг на друга в процесса, аналогичного процессу создания стандартных гели для укладки. 11 биологического протокола начинается с 100-1000 ячеек приостановлено в PBS. Клетки лизированы и РНК преобразуется в cDNA прежде чем устройство используется для разделения геномной ДНК от гибридов cDNA/РНК. После разделения и восстановления, геномных и транскриптомики библиотеки готовятся с использованием процесса, который внимательно следит за протокол комплект подготовки стандартной библиотеки всего генома. Более подробно о разработке и проверке гель seq можно прочитать в лаборатории на публикацию чип «гель seq: всего генома и транскриптом последовательности путем одновременной низкозатратных, ДНК и РНК подготовки библиотеки с использованием полупроницаемой гидрогеля барьеры .” 7
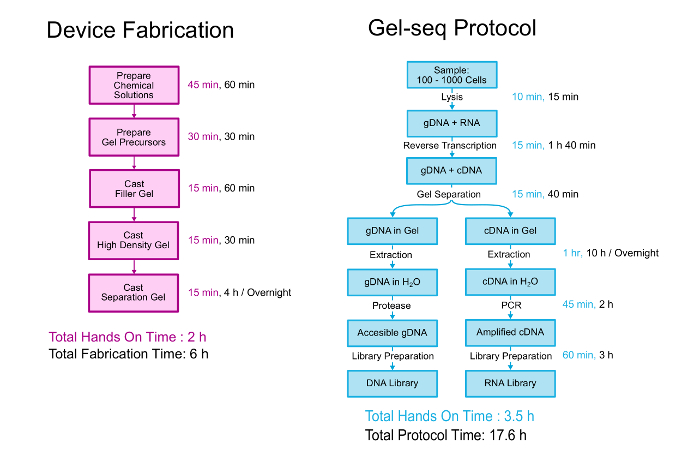
Рисунок 2 . Гель seq протокол. Обзор шагов для изготовления гель seq устройства и протокол для генерации парных библиотеки ДНК и РНК. Части этой фигуры были воспроизведены из ссылка 7 с разрешения Королевского общества химии. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
Для создания библиотеки ДНК и РНК из одной клетки, исследователи следует рассмотреть возможность использования G & T-seq или DR-след G и T-Seq, как гель seq, опирается на физическое разделение РНК от геномной ДНК. Этот подход основывается на матричная РНК (мРНК) 3′ polyadenylated хвост как выпадающее цель. МРНК захватывается на магнитный шарик, используя праймер биотинилированным oligo-dT. После того, как был захвачен мРНК бисер удерживаются на месте с магнитом и супернатанта, содержащий геномной ДНК могут быть удалены и переведен в другой трубки. После завершения этого физического разделения отдельных библиотек могут создаваться мРНК и ДНК. 8 этот подход работает хорошо, если РНК интерес polyadenylated, однако он не может использоваться для изучения стенограммы non-polyadenylated, таких как рибосомная РНК, tRNA, или РНК от прокариот.
DR-seq зависит от предварительного усиления шаг, где cDNA полученных от РНК и ДНК размножаются в том же трубе. Этот образец затем надвое и обрабатываются параллельно подготовить ДНК и РНК seq библиотек. Чтобы различать геномной ДНК и cDNA, производный от РНК, DR-seq занимает вычислительной подход. Последовательности, где присутствуют только экзонов вычислительно подавляются в геномной ДНК-данных, как те могли происходить от ДНК или РНК. 9 преимуществом этого подхода является, что ДНК и РНК/cDNA нужно не быть разделены физически как это делается в гель seq и G и T-seq. Недостатком, однако, является, что DR-seq требует априорных знаний о геноме и транскриптом (то есть, экзонов против интронов) и не может быть идеально подходит для приложений, таких как последовательности ядер, в которых многие стенограммы еще не полностью сращивания и по-прежнему содержать интроны. 12
Роман аспект гель seq является возможность отделить ДНК и РНК в сотни клеток, исключительно на основе размера. Этот метод требует априори знания геном или транскриптом, устойчива против неполной сплайсинга и не ограничивается стенограммы поли adenylated. Для приложений, где исследователь может начать с по крайней мере 100 клеток гель seq обеспечивает простой подход с использованием материалов, дешево и широко доступны.
Protocol
Representative Results
Discussion
Есть несколько критических шагов, связанных с гель seq устройства изготовления, а также сам протокол. Во время изготовления мы рекомендуем, начиная с толщины предписанные слоя для различных регионов геля. Мы потратили значительное время тестирования изготовление различных вариантов и ?…
Declarações
The authors have nothing to disclose.
Acknowledgements
Финансирование для этой работы, предусмотренных в университете Сан-Диего, национальной науки выпускник исследовательских стипендий программы Фонда, низ предоставляют R01-HG007836 и корейского министерства науки, ИКТ и будущего планирования.
Более ранние версии несколько фигур были впервые опубликованы в «Hoople, г. и др. Гель seq: всего генома и транскриптом последовательности путем одновременной низкозатратных, ДНК и РНК подготовки библиотеки с использованием полупроницаемой гидрогеля барьеров. Лаборатории на чип 17, 2619-2630, doi:10.1039 / c7lc00430c (2017).» Лаборатории на чипе санкционировало повторное использование фигур в этой публикации.
Materials
| Reagents | |||
| Acrylamide Monomer | Sigma Aldrich | A8887-100G | |
| Ammonium Persulfate | Sigma Aldrich | A3678-25G | |
| Ampure XP Beads | Beckman Coulter | A63880 | Referred to in the text as solid phase reversible immobilization (SPRI) beads |
| DNA Gel Loading Dye (6x) | ThermoFisher Scientific | R0611 | Referred to in the text as 6X loading dye |
| Ethyl alcohol | Sigma Aldrich | E7023-500ML | |
| KAPA SYBR FAST One-Step qRT-PCR Kits | Kapa BioSystems | 7959613001 | Referred to in the text as 2X qPCR mix |
| N,N′-Methylenebis(acrylamide) | Sigma Aldrich | 146072-100G | Also known as bis-acrylamide |
| NexteraXT DNA Library Preparation Kit (referred to in the text as library preparation kit) | Illumina | FC-131-1024 | Includes: TD (referred to in the text as transposase buffer), ATM (referred to in the text as transposase), NT (referred to in the text as transposase stop buffer), and NPM (Referred to in the text as library prep PCR mix) |
| Nuclease Free Water | Millipore | 3098 | |
| Protease | Qiagen | 19155 | |
| SMART-Seq v4 Kit (referred to in the text as whole-transcript amplification (WTA) kit) | Takara/Clontech | 634888 | Includes: Lysis buffer, RNase inhibitor, 3’ SMART-Seq CDS Primer II A (referred to in the text as RT primer), 5X Ultra Low First Strand Buffer (referred to in the text as first strand buffer), SMART-Seq v4 Oligonucleotide (referred to in the text as template switch oligonucleotide (TSO)), SMART-Scribe Reverse Transcriptase (referred to in the text as reverse transcriptase), and PCR Primer II A (referred to in the text as cDNA PCR primer) |
| Random hexamer with WTA adapter | IDT | n/a | 5′-AAGCAGTGGTATCAACGCAGAGTAC-NNNNNN-3′ |
| Sucrose | Sigma Aldrich | S0389-500G | |
| TEMED | Sigma Aldrich | T9281-25ML | |
| DNA AWAY Surface Decontaminant | ThermoFisher Scientific | 7010PK | Referred to in the text as DNA removal product |
| Tris-Borate-EDTA buffer (10X concentration) | Sigma Aldrich | T4415-1L | |
| SYBR Gold Nucleic Acid Gel Stain (10,000X Concentrate in DMSO) | ThermoFisher Scientific | S11494 | Referred to in the text as gel stain |
| Equipment | |||
| BD Precisionglide syringe needles, gauge 18 | Sigma Aldrich | Z192554 | Any equivalent hardware is acceptable |
| Branson CPX series ultrasonic bath | Sigma Aldrich | Z769363 | Any equivalent hardware is acceptable |
| Empty Gel Cassettes, mini, 1.0 mm | ThermoFisher Scientific | NC2010 | Any equivalent hardware is acceptable |
| Mesh Filter Plate – Corning HTS Transwell 96 well permeable supports – 8.0 µm pore size | Sigma Aldrich | CLS3374 | Referred to in the text as 8 um mesh filter plate |
| PowerPac HC Power Supply | Bio-Rad | 1645052 | Any equivalent hardware is acceptable |
| Qubit Fluorometer | ThermoFisher Scientific | Q33216 | Any equivalent hardware is acceptable |
| Vacufuge Concentrator | Eppendorf | 22822993 | Any equivalent hardware is acceptable |
| XCell SureLock Mini-Cell system | ThermoFisher Scientific | EI0001 | Any equivalent hardware is acceptable |
| Bio-Rad CFX96 Touch Real-Time PCR Detection System | Bio-Rad | 1855195 | Any equivalent hardware is acceptable |
| Amersham UVC 500 Ultraviolet Crosslinker | GE Healthcare Life Sciences | UVC500-115V | Discontinued, any equivalent hardware is acceptable |
| Gel Doc XR+ Gel Documentation System | Bio-Rad | 1708195 | Referred to in the text as gel imager |
| Dark Reader Transilluminator | Clare Chemical Research | DR89 | Referred to in the text as UV transilluminator |
| Ultrasonic Bath | Bransonic | 1207K35 | Any equivalent ultrasonic bath is acceptable. |
Referências
- Mawy, T. Single-cell sequencing. Nat Methods. 11 (1), 18 (2014).
- Gole, J., et al. Massively parallel polymerase cloning and genome sequencing of single cells using nanoliter microwells. Nat Biotech. 31 (12), 1126-1132 (2013).
- Sasagawa, Y., et al. Quartz-Seq: a highly reproducible and sensitive single-cell RNA-Seq reveals non-genetic gene expression heterogeneity. Genome Biol. 14 (4), 31 (2013).
- Ramsköld, D., et al. Full-length mRNA-Seq from single-cell levels of RNA and individual circulating tumor cells. Nat Biotechnol. 30 (8), 777-782 (2012).
- Shapiro, E., Biezuner, T., Linnarsson, S. Single-cell sequencing-based technologies will revolutionize whole-organism science. Nat Rev Genet. 14 (9), 618-630 (2013).
- Navin, N., et al. Tumour evolution inferred by single-cell sequencing. Nature. 472 (7341), 90-94 (2011).
- Hoople, G. D., et al. Gel-seq: whole-genome and transcriptome sequencing by simultaneous low-input DNA and RNA library preparation using semi-permeable hydrogel barriers. Lab Chip. 17, 2619-2630 (2017).
- Macaulay, I. C., et al. G&T-seq: parallel sequencing of single-cell genomes and transcriptomes. Nat Methods. 12 (6), 519-522 (2015).
- Dey, S. S., Kester, L., Spanjaard, B., Bienko, M., van Oudenaarden, A. Integrated genome and transcriptome sequencing of the same cell. Nat Biotechnol. 33 (3), 285-289 (2015).
- Gopal, A., Zhou, Z. H., Knobler, C. M., Gelbart, W. M. Visualizing large RNA molecules in solution. RNA. 18 (2), 284-299 (2012).
- . SDS-PAGE Gel. Cold Spring Harb Protoc. 2015 (7), 087908 (2015).
- Lake, B. B., et al. Neuronal subtypes and diversity revealed by single-nucleus RNA sequencing of the human brain. Science. 352 (6293), (2016).
- Lee, P. Y., Costumbrado, J., Hsu, C. -. Y., Kim, Y. H. Agarose Gel Electrophoresis for the Separation of DNA Fragments. J Vis Exp. (62), e3923 (2012).
- Qubit 3.0 Fluorometer Manual. MAN0010866. Life Technologies Available from: https://tools.thermofisher.com/content/sfs/manuals/qubit_3_fluorometer_man.pd (2017)
- Vitak, S. A., et al. Sequencing thousands of single-cell genomes with combinatorial indexing. Nat Methods. 14 (3), 302-308 (2017).
- Illumina. . Nextera ® XT DNA Library Preparation Kit. , (2017).
- Takara Bio USA Inc. . SMART-Seq v4 Ultra Low Input RNA Kit for Sequencing User Manual. , (2016).
- Kurimoto, K., Yabuta, Y., Ohinata, Y., Saitou, M. Global single-cell cDNA amplification to provide a template for representative high-density oligonucleotide microarray analysis. Nat Protoc. 2 (3), 739-752 (2007).

