Gel-seq: Een methode voor de voorbereiding van de bibliotheek van de gelijktijdige Sequencing van DNA en RNA met Hydrogel Matrices
Summary
Gel-seq kan onderzoekers gelijktijdig bereiden bibliotheken voor beide DNA – en RNA-seq op de te verwaarlozen extra kosten vanaf 100-1000 cellen met behulp van een eenvoudige hydrogel-apparaat. Dit document stelt een gedetailleerde aanpak voor de fabricage van zowel het apparaat als het biologische protocol voor het genereren van gepaarde bibliotheken.
Abstract
Alleen is er de mogelijkheid om te versterken en de opeenvolging van DNA of RNA van kleine startende steekproeven geboekt in de afgelopen vijf jaar. Helaas, de standaardprotocollen voor het genereren van genomic of transcriptomic bibliotheken stroken en onderzoekers moeten kiezen of u wilt de opeenvolging van DNA of RNA dat voor een bepaalde monster. Gel-seq is dit probleem opgelost doordat onderzoekers gelijktijdig voor te bereiden op bibliotheken zowel DNA en RNA beginnen met 100-1000 cellen met behulp van een eenvoudige hydrogel-apparaat. Dit document stelt een gedetailleerde aanpak voor de fabricage van zowel het apparaat als het biologische protocol voor het genereren van gepaarde bibliotheken. We ontworpen Gel-seq zo dat het eenvoudig konden worden geïmplementeerd door andere onderzoekers; vele genetica laboratoria hebben reeds de nodige apparatuur om te reproduceren van de Gel-seq apparaat fabricage. Ons protocol maakt gebruik van veelgebruikte kits voor beide geheel-transcript versterking (WTA) en voorbereiding van de bibliotheek, die ook kunnen worden vertrouwd voor onderzoekers reeds bedreven in genomic genereren en transcriptomic bibliotheken. Onze aanpak maakt het mogelijk onderzoekers aan het uitoefenen van de kracht van zowel DNA en RNA sequencing op één sample zonder splitsing en met te verwaarlozen extra kosten.
Introduction
Volgende generatie sequencing (NGS) heeft een diepgaande invloed gehad op de wijze genetica onderzoek wordt uitgevoerd. Waarin onderzoekers vooral eens gericht op rangschikkend het genoom van een hele diersoort, is het nu mogelijk om het genoom van een enkelvoudige tumor of zelfs een enkele cel in een experiment. 1 NGS heeft ook het kosteneffectief te sequentie van het RNA afschriften gevonden binnen een cel, een verzameling gegevens die als een transcriptome wordt bekend. Alleen is er de mogelijkheid om te versterken en de opeenvolging van DNA of RNA van kleine startende steekproeven geboekt in de afgelopen vijf jaar. 2 , 3 , 4 helaas standaard protocollen zijn niet compatibel en onderzoekers moeten kiezen of u wilt de opeenvolging van DNA of RNA dat voor een gegeven steekproef. Wanneer een eerste steekproef groot genoeg is, kan het in tweeën worden gesplitst. Op kleinere schalen, echter verlies van materiaal als gevolg van splitsing van monsters kan invloed hebben op de kwaliteit van de bibliotheek, en bundeling van monsters kan gemiddelde uit interessante verschillen tussen cellen. 5 voorts onderzoekers zijn steeds meer geïnteresseerd in het onderzoek van de monsters die niet kunnen worden gesplitst, zoals afzonderlijke cellen of kleine heterogene tumor biopsieën. 6
Om aan te pakken dit probleem, drie protocollen zijn onlangs ontwikkeld om zowel DNA en RNA van hetzelfde uitgangspunt monster volgnummer: Gel-seq7, G & T-seq8pt9van de DR-seq. Dit artikel presenteert een gedetailleerd protocol voor Gel-seq, die kan worden gebruikt om gelijktijdig het genereren van DNA en RNA bibliotheken uit zo weinig als 100 cellen op de te verwaarlozen extra kosten. De roman aspect van Gel-seq is de capaciteit om DNA en RNA uitsluitend gebaseerd op grootte met behulp van low-cost hydrogel matrices te scheiden. De innovatie van de kern van het Gel-Seq-protocol is de fysieke scheiding van DNA van RNA. Deze scheiding wordt bereikt electrophoretically met behulp van een combinatie van polyacrylamide membranen die van de verschillen van grootte tussen deze moleculen profiteren. Om deze grootte verschillen in context, overwegen hoe DNA en RNA zijn beeld: terwijl DNA op de micron-schaal bestaat en kan bekeken worden met behulp van traditionele microscopen, RNA bestaat op de nanometerschaal en moet worden beeld met behulp van complexe technieken zoals cryo-elektron microscopie. 10
De aanpak van het scheiden van DNA en RNA in dit protocol is afgebeeld in Figuur 1. Het linker paneel toont DNA en RNA vrij zwevend in de oplossing in de buurt van een membraan. Wanneer een elektrisch veld wordt toegepast, zoals wordt weergegeven in het rechter paneel, ervaring DNA en RNA een elektroforetisch kracht die induceert van migratie door het membraan. Door de eigenschappen van de membraan tuning, hebben we een semi-permeabel membraan die scheidt van DNA van RNA. De DNA moleculen zijn tegen het membraan geduwd, maar geraakt aan de rand vanwege hun grote omvang. Kleine molecules van RNA, aan de andere kant, kunnen configureren en weven hun weg door het membraan. Dit proces, bekend als reptation, is vergelijkbaar met de manier waarop die een slang door gras beweegt. Uiteindelijk deze RNA-moleculen worden gestopt door een tweede, high-density membraan, dat is te moeilijk voor zelfs kleinere polymeren (> 200 basenparen) te onderuit door. Zodra fysiek gescheiden, kan DNA en RNA worden hersteld en verwerkt voor het genereren van informatie over zowel het genoom en de transcriptome. Terwijl we DNA en RNA scheiden kunt, hebben we vonden betere resultaten worden verkregen als de RNA via reverse wordt-herschreven als cDNA vóór scheiding. De cDNA/RNA-hybriden zijn stabieler dan RNA alleen en kunnen nog aan de lage dichtheid membraan passeren.
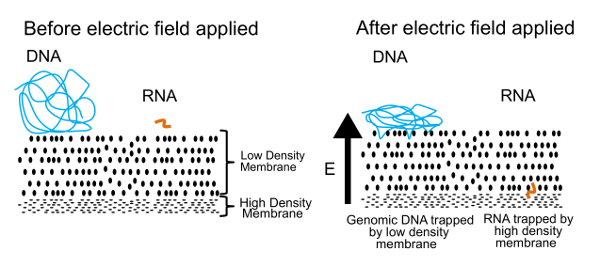
Figuur 1 . Gel-seq operationele principe. Het onderliggende principe gebruikt voor fysiek gescheiden van DNA en RNA. In een toegepast elektrisch veld, kleine molecules van RNA worden gemigreerd door de lage dichtheid membraan maar grote DNA moleculen zitten op het oppervlak. Dit cijfer was van Ref. 7 gereproduceerd met toestemming van de Royal Society of Chemistry. Klik hier voor een grotere versie van dit cijfer.
Deze paper beschrijft in detail zowel de fabricage van de Gel-seq-apparaat en het biologische protocol voor het genereren van gepaarde bibliotheken van DNA en RNA. Een overzicht van beide is afgebeeld in Figuur 2. Het apparaat is vervaardigd door gelaagdheid drie verschillende dichtheid polyacrylamide gels boven op elkaar in een proces gelijkend op het maken van standaard stapelvolgorde gels. 11 het biologische protocol begint met 100-1000 cellen gesuspendeerd in PBS. De cellen zijn lysed en RNA wordt omgezet in cDNA voordat het apparaat wordt gebruikt om te scheiden van de genomic DNA van de cDNA/RNA hybriden. Na de scheiding en terugwinning, genomic en transcriptomic zijn bibliotheken opgesteld op basis van een proces dat volgt op het geheel-genoom-standaardbibliotheek voorbereiding kit protocol. Verdere details over de ontwikkeling en validatie van Gel-seq kan worden gelezen in het Lab op een Chip publicatie “Gel-seq: geheel-genoom en de sequencing van de transcriptome door gelijktijdige lage-input DNA en RNA bibliotheek voorbereiding met behulp van semi-permeabel hydrogel belemmeringen .” 7
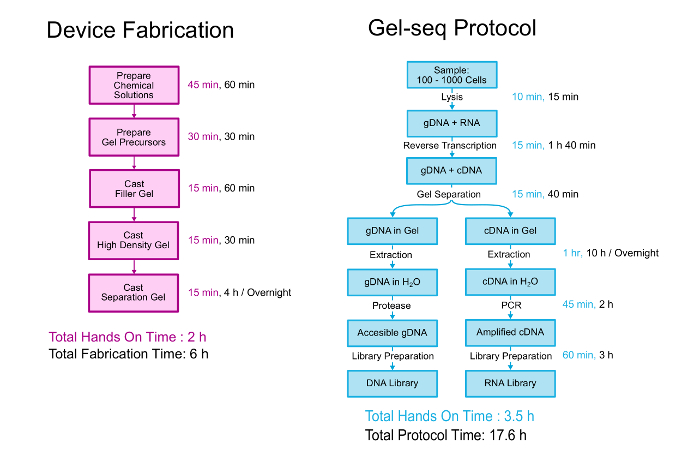
Figuur 2 . Gel-seq Protocol. Een overzicht van de stappen voor het fabriceren van het Gel-seq-apparaat en het protocol bij gegenereerde gepaarde DNA en RNA bibliotheken. Gedeelten van dit cijfer waren van Ref. 7 gereproduceerd met toestemming van de Royal Society of Chemistry. Klik hier voor een grotere versie van dit cijfer.
Voor het genereren van DNA en RNA bibliotheken van afzonderlijke cellen, onderzoekers overwegen G & T-seq of DR-seq. G & T-Seq, zoals Gel-seq te gebruiken, is afhankelijk van een fysieke scheiding van RNA van genomic DNA. Deze aanpak berust op messenger RNA (mRNA) 3 ‘ polyadenylated staart als een pull-down-doel. De mRNA wordt vastgelegd op een magnetische kraal met behulp van een biotinyleerd oligo-dT primer. Zodra het mRNA is gevangen de kralen zijn met een magneet op zijn plaats gehouden en het supernatant met de genomic DNA kan worden verwijderd en kan worden overgedragen aan een andere buis. Na deze fysieke scheiding voltooid is, kunnen de aparte bibliotheken worden gegenereerd vanuit de mRNA en DNA. 8 deze aanpak werkt goed als het RNA van belang polyadenylated, nochtans niet het worden gebruikt om te studeren van afschriften van de niet-polyadenylated, zoals ribosomaal RNA, tRNA, of RNA van prokaryoten.
DR-seq is afhankelijk van een versterking van de pre-stap waar zowel DNA en cDNA afgeleid van RNA worden versterkt in de dezelfde buis. Het monster wordt vervolgens in tweeën gesplitst en verwerkt parallel aan het bereiden van DNA – en RNA-seq bibliotheken. Om deze te onderscheiden van genomic DNA en cDNA afgeleid van RNA, neemt DR-seq een rekenkundige benadering. Sequenties waar alleen exons aanwezig zijn worden rekenkundig onderdrukt in de genomic DNA-gegevens, zoals die zou zijn ontstaan van DNA of RNA. 9 een voordeel van deze aanpak is dat het DNA en cDNA/RNA niet worden fysiek gescheiden moeten zoals is gedaan in Gel-seq en G & T-seq. Het nadeel is echter dat DR-seq een priori kennis van het genoom en de transcriptome (d.w.z., exons en introns vereist), en niet ideaal voor toepassingen zoals het rangschikken van kernen wellicht, waarin vele transcripties nog niet volledig zijn verbinding aan de randen en bevatten nog steeds introns. 12
De roman aspect van Gel-seq is de mogelijkheid om te scheiden van DNA en RNA in honderden cellen uitsluitend gebaseerd op grootte. Deze methode vereist geen a priori kennis van het genoom- of transcriptome, is robuust tegen onvolledig splicing, en is niet beperkt tot de afschriften van de poly-adenylated. Voor toepassingen waar een onderzoeker met ten minste 100 cellen beginnen kan, biedt Gel-seq een eenvoudige aanpak met behulp van goedkope en wijd-beschikbare materialen.
Protocol
Representative Results
Discussion
Er zijn verschillende kritische stappen die zijn gekoppeld aan de Gel-seq apparaat fabricage, alsmede het protocol zelf. Tijdens fabricage, is het raadzaam te beginnen met de voorgeschreven laagdiktes voor de verschillende regio’s van de gel. We tijd aanzienlijke testen verschillende fabricage opties en het protocol hier beschreven produceert de beste apparaten voor de cassettes vermeld in de tabel van materialen en reagentia. Als onderzoekers een alternatieve cassette systeem gebruiken, kunnen zij vinde…
Declarações
The authors have nothing to disclose.
Acknowledgements
Financiering voor dit werk werd door de Universiteit van San Diego, de National Science Foundation Graduate Research Fellowship Program, NIH grant R01-HG007836 en door het Koreaanse Ministerie van wetenschap, ICT en Planning van de toekomst.
Eerdere versies van een aantal cijfers werden voor het eerst gepubliceerd in “Hoople, G. D. et al. Gel-seq: geheel-genoom en de sequencing van de transcriptome door gelijktijdige lage-input DNA en RNA bibliotheek voorbereiding met behulp van semi-permeabel hydrogel belemmeringen. Lab op een Chip 17, 2619-2630, doi:10.1039 / c7lc00430c (2017). ” Lab op een Chip heeft bestraft het hergebruik van de cijfers in deze publicatie.
Materials
| Reagents | |||
| Acrylamide Monomer | Sigma Aldrich | A8887-100G | |
| Ammonium Persulfate | Sigma Aldrich | A3678-25G | |
| Ampure XP Beads | Beckman Coulter | A63880 | Referred to in the text as solid phase reversible immobilization (SPRI) beads |
| DNA Gel Loading Dye (6x) | ThermoFisher Scientific | R0611 | Referred to in the text as 6X loading dye |
| Ethyl alcohol | Sigma Aldrich | E7023-500ML | |
| KAPA SYBR FAST One-Step qRT-PCR Kits | Kapa BioSystems | 7959613001 | Referred to in the text as 2X qPCR mix |
| N,N′-Methylenebis(acrylamide) | Sigma Aldrich | 146072-100G | Also known as bis-acrylamide |
| NexteraXT DNA Library Preparation Kit (referred to in the text as library preparation kit) | Illumina | FC-131-1024 | Includes: TD (referred to in the text as transposase buffer), ATM (referred to in the text as transposase), NT (referred to in the text as transposase stop buffer), and NPM (Referred to in the text as library prep PCR mix) |
| Nuclease Free Water | Millipore | 3098 | |
| Protease | Qiagen | 19155 | |
| SMART-Seq v4 Kit (referred to in the text as whole-transcript amplification (WTA) kit) | Takara/Clontech | 634888 | Includes: Lysis buffer, RNase inhibitor, 3’ SMART-Seq CDS Primer II A (referred to in the text as RT primer), 5X Ultra Low First Strand Buffer (referred to in the text as first strand buffer), SMART-Seq v4 Oligonucleotide (referred to in the text as template switch oligonucleotide (TSO)), SMART-Scribe Reverse Transcriptase (referred to in the text as reverse transcriptase), and PCR Primer II A (referred to in the text as cDNA PCR primer) |
| Random hexamer with WTA adapter | IDT | n/a | 5′-AAGCAGTGGTATCAACGCAGAGTAC-NNNNNN-3′ |
| Sucrose | Sigma Aldrich | S0389-500G | |
| TEMED | Sigma Aldrich | T9281-25ML | |
| DNA AWAY Surface Decontaminant | ThermoFisher Scientific | 7010PK | Referred to in the text as DNA removal product |
| Tris-Borate-EDTA buffer (10X concentration) | Sigma Aldrich | T4415-1L | |
| SYBR Gold Nucleic Acid Gel Stain (10,000X Concentrate in DMSO) | ThermoFisher Scientific | S11494 | Referred to in the text as gel stain |
| Equipment | |||
| BD Precisionglide syringe needles, gauge 18 | Sigma Aldrich | Z192554 | Any equivalent hardware is acceptable |
| Branson CPX series ultrasonic bath | Sigma Aldrich | Z769363 | Any equivalent hardware is acceptable |
| Empty Gel Cassettes, mini, 1.0 mm | ThermoFisher Scientific | NC2010 | Any equivalent hardware is acceptable |
| Mesh Filter Plate – Corning HTS Transwell 96 well permeable supports – 8.0 µm pore size | Sigma Aldrich | CLS3374 | Referred to in the text as 8 um mesh filter plate |
| PowerPac HC Power Supply | Bio-Rad | 1645052 | Any equivalent hardware is acceptable |
| Qubit Fluorometer | ThermoFisher Scientific | Q33216 | Any equivalent hardware is acceptable |
| Vacufuge Concentrator | Eppendorf | 22822993 | Any equivalent hardware is acceptable |
| XCell SureLock Mini-Cell system | ThermoFisher Scientific | EI0001 | Any equivalent hardware is acceptable |
| Bio-Rad CFX96 Touch Real-Time PCR Detection System | Bio-Rad | 1855195 | Any equivalent hardware is acceptable |
| Amersham UVC 500 Ultraviolet Crosslinker | GE Healthcare Life Sciences | UVC500-115V | Discontinued, any equivalent hardware is acceptable |
| Gel Doc XR+ Gel Documentation System | Bio-Rad | 1708195 | Referred to in the text as gel imager |
| Dark Reader Transilluminator | Clare Chemical Research | DR89 | Referred to in the text as UV transilluminator |
| Ultrasonic Bath | Bransonic | 1207K35 | Any equivalent ultrasonic bath is acceptable. |
Referências
- Mawy, T. Single-cell sequencing. Nat Methods. 11 (1), 18 (2014).
- Gole, J., et al. Massively parallel polymerase cloning and genome sequencing of single cells using nanoliter microwells. Nat Biotech. 31 (12), 1126-1132 (2013).
- Sasagawa, Y., et al. Quartz-Seq: a highly reproducible and sensitive single-cell RNA-Seq reveals non-genetic gene expression heterogeneity. Genome Biol. 14 (4), 31 (2013).
- Ramsköld, D., et al. Full-length mRNA-Seq from single-cell levels of RNA and individual circulating tumor cells. Nat Biotechnol. 30 (8), 777-782 (2012).
- Shapiro, E., Biezuner, T., Linnarsson, S. Single-cell sequencing-based technologies will revolutionize whole-organism science. Nat Rev Genet. 14 (9), 618-630 (2013).
- Navin, N., et al. Tumour evolution inferred by single-cell sequencing. Nature. 472 (7341), 90-94 (2011).
- Hoople, G. D., et al. Gel-seq: whole-genome and transcriptome sequencing by simultaneous low-input DNA and RNA library preparation using semi-permeable hydrogel barriers. Lab Chip. 17, 2619-2630 (2017).
- Macaulay, I. C., et al. G&T-seq: parallel sequencing of single-cell genomes and transcriptomes. Nat Methods. 12 (6), 519-522 (2015).
- Dey, S. S., Kester, L., Spanjaard, B., Bienko, M., van Oudenaarden, A. Integrated genome and transcriptome sequencing of the same cell. Nat Biotechnol. 33 (3), 285-289 (2015).
- Gopal, A., Zhou, Z. H., Knobler, C. M., Gelbart, W. M. Visualizing large RNA molecules in solution. RNA. 18 (2), 284-299 (2012).
- . SDS-PAGE Gel. Cold Spring Harb Protoc. 2015 (7), 087908 (2015).
- Lake, B. B., et al. Neuronal subtypes and diversity revealed by single-nucleus RNA sequencing of the human brain. Science. 352 (6293), (2016).
- Lee, P. Y., Costumbrado, J., Hsu, C. -. Y., Kim, Y. H. Agarose Gel Electrophoresis for the Separation of DNA Fragments. J Vis Exp. (62), e3923 (2012).
- Qubit 3.0 Fluorometer Manual. MAN0010866. Life Technologies Available from: https://tools.thermofisher.com/content/sfs/manuals/qubit_3_fluorometer_man.pd (2017)
- Vitak, S. A., et al. Sequencing thousands of single-cell genomes with combinatorial indexing. Nat Methods. 14 (3), 302-308 (2017).
- Illumina. . Nextera ® XT DNA Library Preparation Kit. , (2017).
- Takara Bio USA Inc. . SMART-Seq v4 Ultra Low Input RNA Kit for Sequencing User Manual. , (2016).
- Kurimoto, K., Yabuta, Y., Ohinata, Y., Saitou, M. Global single-cell cDNA amplification to provide a template for representative high-density oligonucleotide microarray analysis. Nat Protoc. 2 (3), 739-752 (2007).

