Gel-seq: Un metodo per la preparazione di libreria simultanea di sequenziamento del DNA e RNA utilizzando matrici di idrogel
Summary
Gel-seq consente ai ricercatori di preparare contemporaneamente le librerie per entrambi DNA – e RNA-seq trascurabile costo aggiunto a partire da 100-1000 cellule utilizzando un dispositivo semplice idrogel. Questa carta presenta un approccio dettagliato per la fabbricazione del dispositivo, nonché il protocollo biologico per generare librerie accoppiate.
Abstract
La capacità di amplificare e sequenza di DNA o RNA da piccoli campioni di partenza è stato raggiunto solo negli ultimi cinque anni. Purtroppo, i protocolli standard per la generazione genomico o librerie di trascrittomica sono incompatibili e i ricercatori devono scegliere se sequenza DNA o RNA per un particolare campione. Gel-seq risolve questo problema consentendo ai ricercatori di preparare contemporaneamente le librerie per sia DNA che RNA a partire con 100-1000 cellule utilizzando un dispositivo semplice idrogel. Questa carta presenta un approccio dettagliato per la fabbricazione del dispositivo, nonché il protocollo biologico per generare librerie accoppiate. Abbiamo progettato di Gel-seq, così che potrebbe essere facilmente implementato da altri ricercatori; molti laboratori di genetica hanno già le attrezzature necessarie per riprodurre la fabbricazione di dispositivi di Gel-seq. Il nostro protocollo impiega Kit comunemente usati per l’amplificazione sia intero-trascrizione (WTA) e preparazione di biblioteca, che sono anche suscettibili di essere familiare ai ricercatori già esperto in generazione genomica e trascrittomica librerie. Il nostro approccio permette ai ricercatori di far per valere il potere di sequenziamento del DNA e di RNA su un singolo campione senza spaccare e con costo aggiuntivo trascurabile.
Introduction
Prossima generazione sequenziamento (NGS) ha avuto un profondo impatto sulla strada la ricerca genetica è condotta. Dove i ricercatori una volta concentrata sul sequenziamento del genoma di un’intera specie, ora è possibile sequenziare il genoma di un singolo tumore o anche una singola cella in un esperimento. 1 NGS ha anche reso conveniente per sequenziare i trascritti di RNA trovati all’interno di una cella, un insieme di dati noto come il trascrittoma. La capacità di amplificare e sequenza di DNA o RNA da piccoli campioni di partenza è stato raggiunto solo negli ultimi cinque anni. 2 , 3 , 4 purtroppo, protocolli standard sono incompatibili e i ricercatori devono decidere di sequenza di DNA o RNA per un dato campione. Quando un campione di partenza è abbastanza grande, può essere diviso a metà. Alle scale più piccole, tuttavia, perdita di materiale dovuto spaccare campioni può influire sulla qualità di biblioteca e pool di campioni possono media di interessanti variazioni fra le cellule. 5 inoltre, i ricercatori sono sempre più interessati ad esaminare i campioni che non possono essere suddivisi, come cellule singole o biopsie di piccolo tumore eterogeneo. 6
Per risolvere questo problema, tre protocolli sono stati recentemente sviluppati per RNA e DNA di sequenza dallo stesso campione iniziale: Gel-seq7, G & T-seq8e DR-seq9. Questo articolo presenta un protocollo dettagliato per Gel-seq, che può essere utilizzato per generare simultaneamente librerie di DNA e RNA da poco più di 100 celle a costi trascurabili aggiunto. L’aspetto di novità di Gel-seq è la capacità di separare DNA e RNA basata esclusivamente sulla dimensione utilizzando matrici di idrogel di basso costo. L’innovazione di nucleo del protocollo Gel-Seq è la separazione fisica del DNA da RNA. Questa separazione si ottiene elettroforeticamente utilizzando una combinazione di membrane di poliacrilammide che sfruttano le differenze di dimensioni tra queste molecole. Per mettere queste differenze di dimensione nel contesto, considera come DNA e RNA sono Imaging: mentre DNA esiste sulla micron-scala e possa essere visualizzato utilizzando microscopi tradizionali, RNA esiste su scala nanometrica e deve essere imaged utilizzando tecniche complesse come la cryo-elettrone microscopia. 10
L’approccio alla separazione del DNA e RNA in questo protocollo è illustrato nella Figura 1. Il pannello di sinistra mostra DNA e RNA gratis galleggianti in soluzione nei pressi di una membrana. Quando viene applicato un campo elettrico, come mostrato nel pannello di destra, DNA e RNA esperienza una forza elettroforetica che induce la migrazione attraverso la membrana. Regolando le proprietà della membrana, abbiamo creato una membrana semipermeabile che separa il DNA dal RNA. Le molecole di DNA vengono spinte contro la membrana, ma rimanere impigliate ai margini a causa delle loro grandi dimensioni. Piccole molecole di RNA, d’altra parte, possono riconfigurare e tessere la loro strada attraverso la membrana. Questo processo, noto come reptation, è simile a quello di che un serpente si muove attraverso l’erba. Alla fine queste molecole di RNA vengono fermate da una seconda membrana ad alta densità che è troppo difficile per i polimeri ancora più piccoli (> 200 paia di basi) a divincolarsi attraverso. Una volta fisicamente separato, DNA e RNA possono essere recuperati ed elaborati per generare informazioni sul genoma e il trascrittoma. Mentre possiamo separare DNA e RNA, abbiamo trovato risultati migliori si ottengono se il RNA è inverso trascritto in cDNA prima della separazione. Gli ibridi di cDNA/RNA sono più stabili di RNA da solo e possono ancora passare attraverso la membrana a bassa densità.
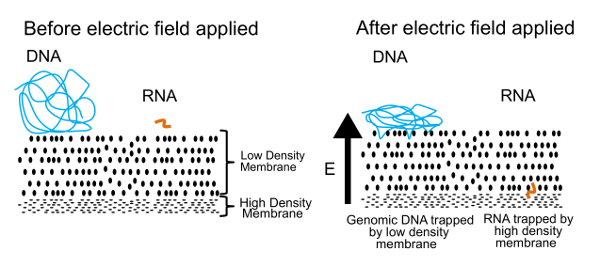
Figura 1 . Principio di funzionamento del gel-seq. Il principio sottostante utilizzato per separare fisicamente il DNA e RNA. In un campo elettrico applicato, piccole molecole di RNA migrano attraverso la membrana a bassa densità ma grandi molecole di DNA sono intrappolati sulla superficie. Questa figura è stata riprodotta da Ref. 7 con il permesso della Royal Society of Chemistry. Clicca qui per visualizzare una versione più grande di questa figura.
Questo articolo descrive in dettaglio sia la fabbricazione del dispositivo Gel-seq e accoppiato il protocollo biologico per generare librerie di DNA e RNA. Una panoramica di entrambi è illustrata nella Figura 2. Il dispositivo è fabbricato da stratificazione tre differenti densità gel di poliacrilammide in cima a vicenda in un processo simile alla creazione di standard gel d’impilamento. 11 il protocollo biologico inizia con 100-1000 cellule sospese in PBS. Le cellule sono lisate e il RNA viene convertito in cDNA prima che il dispositivo viene utilizzato per separare il DNA genomico da ibridi cDNA/RNA. Dopo la separazione e recupero, genomica e trascrittomica librerie vengono preparate utilizzando un processo che segue da vicino il protocollo di kit di preparazione libreria standard intero genoma. Ulteriori dettagli sullo sviluppo e la convalida di Gel-seq possono essere letto in laboratorio su una pubblicazione di Chip “Gel-seq: intero genoma e sequenziamento del trascrittoma di basso input del DNA e RNA libreria preparazione simultanea utilizzando barriere semipermeabile idrogel .” 7
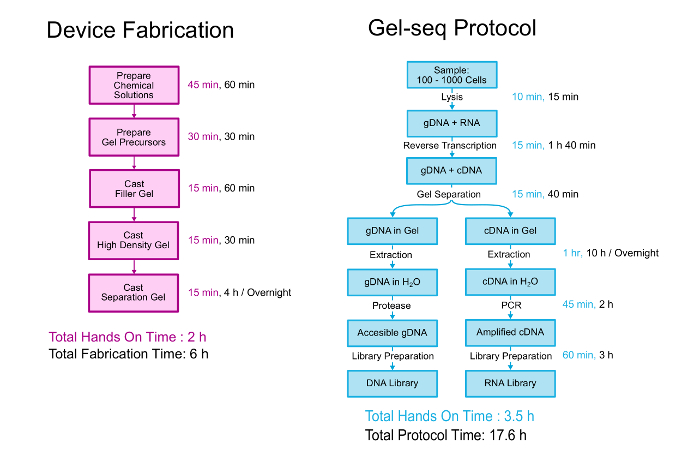
Figura 2 . Protocollo di gel-seq. Una panoramica dei passaggi per fabbricare il dispositivo Gel-seq e il protocollo generare coppia librerie di DNA e RNA. Porzioni di questa figura sono state riprodotte da Ref. 7 con il permesso della Royal Society of Chemistry. Clicca qui per visualizzare una versione più grande di questa figura.
Per generare le librerie di DNA e RNA da cellule singole, i ricercatori dovrebbero considerare l’utilizzo di G & T-seq o DR-segg. G & T-Seq, come Gel-seq, si basa su una separazione fisica di RNA dal DNA genomico. Questo approccio si basa sul RNA messaggero (mRNA) 3 ′ poliadenilazione coda come una menu a discesa destinazione. il mRNA viene catturato il magnetic beads utilizzando un primer oligo-dT biotinilato. Una volta che il mRNA è stato catturato le perline sono tenute in posizione da un magnete e il supernatante contenente il DNA genomico può essere rimosso e trasferito in un’altra provetta. Dopo aver completata questa separazione fisica, librerie separate possono essere generate dal mRNA e DNA. 8 questo approccio funziona bene se il RNA di interesse è poliadenilazione, tuttavia non può essere utilizzato per studiare le trascrizioni non poliadenilazione, come RNA ribosomiale, tRNA o RNA da procarioti.
DR-seq si basa su un passaggio di pre-amplificazione dove sono amplificati sia DNA e cDNA derivate da RNA nello stesso tubo. Il campione è quindi diviso in due ed elaborato in parallelo per preparare librerie di DNA e RNA-seq. Per distinguere tra il DNA di genomic e il cDNA derivate da RNA, DR-seq adotta un approccio computazionale. Sequenze dove sono presenti solo esoni informaticamente vengono soppressi nei dati di DNA genomici, come quelli potrebbe avere avuto origine da DNA o RNA. 9 un vantaggio di questo approccio è che il DNA e cDNA/RNA bisogno di non essere fisicamente separate come avviene in Gel-seq e G & T-segg. Lo svantaggio, tuttavia, è che il DR-seq richiede conoscenza a priori del genoma e del trascrittoma (cioè, esoni e introni) e potrebbe non essere l’ideale per applicazioni come il sequenziamento dei nuclei, in cui molte trascrizioni non sono ancora completamente impiombato e contengono ancora gli introni. 12
L’aspetto di novità di Gel-seq è la capacità di separare il DNA ed il RNA in centinaia di celle basate esclusivamente sulla dimensione. Questo metodo richiede nessuna conoscenza aprioristicamente del genoma o del trascrittoma, è robusta contro splicing incompleta e non è limitata alle trascrizioni di poli-adenylated. Per le applicazioni dove un ricercatore può iniziare con almeno 100 cellule, Gel-seq fornisce un approccio diretto con materiali economici e ampiamente disponibili.
Protocol
Representative Results
Discussion
Ci sono diversi passaggi critici connessi con la fabbricazione di dispositivi di Gel-seq, nonché il protocollo stesso. Durante la produzione, si consiglia di iniziare con gli spessori di strato prescritti per le varie regioni del gel. Abbiamo trascorso un tempo significativo fabbricazione differenti opzioni di test e il protocollo descritto qui produce i migliori dispositivi per le cassette elencati nella tabella materiali e reagenti. Se i ricercatori usano un sistema alternativo di cassetta, trovino ne…
Declarações
The authors have nothing to disclose.
Acknowledgements
Finanziamenti per questo lavoro è stato indicato dall’Università di San Diego, la National Science Foundation Graduate Research Fellowship programma, NIH concede R01-HG007836 e dal ministero coreano della scienza, ICT e pianificazione del futuro.
Versioni precedenti di un parecchie figure furono pubblicate dapprima nel “Hoople, D. G. et al. Gel-seq: intero genoma e sequenziamento del trascrittoma di basso input del DNA e RNA libreria preparazione simultanea utilizzando barriere idrogel semi-permeabile. Laboratorio su un Chip 17, 2619-2630, doi:10.1039 / c7lc00430c (2017). ” Laboratorio su un Chip ha sancito il riutilizzo delle figure in questa pubblicazione.
Materials
| Reagents | |||
| Acrylamide Monomer | Sigma Aldrich | A8887-100G | |
| Ammonium Persulfate | Sigma Aldrich | A3678-25G | |
| Ampure XP Beads | Beckman Coulter | A63880 | Referred to in the text as solid phase reversible immobilization (SPRI) beads |
| DNA Gel Loading Dye (6x) | ThermoFisher Scientific | R0611 | Referred to in the text as 6X loading dye |
| Ethyl alcohol | Sigma Aldrich | E7023-500ML | |
| KAPA SYBR FAST One-Step qRT-PCR Kits | Kapa BioSystems | 7959613001 | Referred to in the text as 2X qPCR mix |
| N,N′-Methylenebis(acrylamide) | Sigma Aldrich | 146072-100G | Also known as bis-acrylamide |
| NexteraXT DNA Library Preparation Kit (referred to in the text as library preparation kit) | Illumina | FC-131-1024 | Includes: TD (referred to in the text as transposase buffer), ATM (referred to in the text as transposase), NT (referred to in the text as transposase stop buffer), and NPM (Referred to in the text as library prep PCR mix) |
| Nuclease Free Water | Millipore | 3098 | |
| Protease | Qiagen | 19155 | |
| SMART-Seq v4 Kit (referred to in the text as whole-transcript amplification (WTA) kit) | Takara/Clontech | 634888 | Includes: Lysis buffer, RNase inhibitor, 3’ SMART-Seq CDS Primer II A (referred to in the text as RT primer), 5X Ultra Low First Strand Buffer (referred to in the text as first strand buffer), SMART-Seq v4 Oligonucleotide (referred to in the text as template switch oligonucleotide (TSO)), SMART-Scribe Reverse Transcriptase (referred to in the text as reverse transcriptase), and PCR Primer II A (referred to in the text as cDNA PCR primer) |
| Random hexamer with WTA adapter | IDT | n/a | 5′-AAGCAGTGGTATCAACGCAGAGTAC-NNNNNN-3′ |
| Sucrose | Sigma Aldrich | S0389-500G | |
| TEMED | Sigma Aldrich | T9281-25ML | |
| DNA AWAY Surface Decontaminant | ThermoFisher Scientific | 7010PK | Referred to in the text as DNA removal product |
| Tris-Borate-EDTA buffer (10X concentration) | Sigma Aldrich | T4415-1L | |
| SYBR Gold Nucleic Acid Gel Stain (10,000X Concentrate in DMSO) | ThermoFisher Scientific | S11494 | Referred to in the text as gel stain |
| Equipment | |||
| BD Precisionglide syringe needles, gauge 18 | Sigma Aldrich | Z192554 | Any equivalent hardware is acceptable |
| Branson CPX series ultrasonic bath | Sigma Aldrich | Z769363 | Any equivalent hardware is acceptable |
| Empty Gel Cassettes, mini, 1.0 mm | ThermoFisher Scientific | NC2010 | Any equivalent hardware is acceptable |
| Mesh Filter Plate – Corning HTS Transwell 96 well permeable supports – 8.0 µm pore size | Sigma Aldrich | CLS3374 | Referred to in the text as 8 um mesh filter plate |
| PowerPac HC Power Supply | Bio-Rad | 1645052 | Any equivalent hardware is acceptable |
| Qubit Fluorometer | ThermoFisher Scientific | Q33216 | Any equivalent hardware is acceptable |
| Vacufuge Concentrator | Eppendorf | 22822993 | Any equivalent hardware is acceptable |
| XCell SureLock Mini-Cell system | ThermoFisher Scientific | EI0001 | Any equivalent hardware is acceptable |
| Bio-Rad CFX96 Touch Real-Time PCR Detection System | Bio-Rad | 1855195 | Any equivalent hardware is acceptable |
| Amersham UVC 500 Ultraviolet Crosslinker | GE Healthcare Life Sciences | UVC500-115V | Discontinued, any equivalent hardware is acceptable |
| Gel Doc XR+ Gel Documentation System | Bio-Rad | 1708195 | Referred to in the text as gel imager |
| Dark Reader Transilluminator | Clare Chemical Research | DR89 | Referred to in the text as UV transilluminator |
| Ultrasonic Bath | Bransonic | 1207K35 | Any equivalent ultrasonic bath is acceptable. |
Referências
- Mawy, T. Single-cell sequencing. Nat Methods. 11 (1), 18 (2014).
- Gole, J., et al. Massively parallel polymerase cloning and genome sequencing of single cells using nanoliter microwells. Nat Biotech. 31 (12), 1126-1132 (2013).
- Sasagawa, Y., et al. Quartz-Seq: a highly reproducible and sensitive single-cell RNA-Seq reveals non-genetic gene expression heterogeneity. Genome Biol. 14 (4), 31 (2013).
- Ramsköld, D., et al. Full-length mRNA-Seq from single-cell levels of RNA and individual circulating tumor cells. Nat Biotechnol. 30 (8), 777-782 (2012).
- Shapiro, E., Biezuner, T., Linnarsson, S. Single-cell sequencing-based technologies will revolutionize whole-organism science. Nat Rev Genet. 14 (9), 618-630 (2013).
- Navin, N., et al. Tumour evolution inferred by single-cell sequencing. Nature. 472 (7341), 90-94 (2011).
- Hoople, G. D., et al. Gel-seq: whole-genome and transcriptome sequencing by simultaneous low-input DNA and RNA library preparation using semi-permeable hydrogel barriers. Lab Chip. 17, 2619-2630 (2017).
- Macaulay, I. C., et al. G&T-seq: parallel sequencing of single-cell genomes and transcriptomes. Nat Methods. 12 (6), 519-522 (2015).
- Dey, S. S., Kester, L., Spanjaard, B., Bienko, M., van Oudenaarden, A. Integrated genome and transcriptome sequencing of the same cell. Nat Biotechnol. 33 (3), 285-289 (2015).
- Gopal, A., Zhou, Z. H., Knobler, C. M., Gelbart, W. M. Visualizing large RNA molecules in solution. RNA. 18 (2), 284-299 (2012).
- . SDS-PAGE Gel. Cold Spring Harb Protoc. 2015 (7), 087908 (2015).
- Lake, B. B., et al. Neuronal subtypes and diversity revealed by single-nucleus RNA sequencing of the human brain. Science. 352 (6293), (2016).
- Lee, P. Y., Costumbrado, J., Hsu, C. -. Y., Kim, Y. H. Agarose Gel Electrophoresis for the Separation of DNA Fragments. J Vis Exp. (62), e3923 (2012).
- Qubit 3.0 Fluorometer Manual. MAN0010866. Life Technologies Available from: https://tools.thermofisher.com/content/sfs/manuals/qubit_3_fluorometer_man.pd (2017)
- Vitak, S. A., et al. Sequencing thousands of single-cell genomes with combinatorial indexing. Nat Methods. 14 (3), 302-308 (2017).
- Illumina. . Nextera ® XT DNA Library Preparation Kit. , (2017).
- Takara Bio USA Inc. . SMART-Seq v4 Ultra Low Input RNA Kit for Sequencing User Manual. , (2016).
- Kurimoto, K., Yabuta, Y., Ohinata, Y., Saitou, M. Global single-cell cDNA amplification to provide a template for representative high-density oligonucleotide microarray analysis. Nat Protoc. 2 (3), 739-752 (2007).

