Gel-seq: Un método para la preparación de biblioteca simultánea de secuenciación de DNA y RNA utilizando Matrices de hidrogel
Summary
Gel-seq permite a los investigadores a preparar simultáneamente las bibliotecas de ambos ADN – y RNA-seq en el insignificante costo adicional a partir de 100-1000 células usando un dispositivo simple de hidrogel. Este artículo presenta un enfoque detallado para la fabricación del dispositivo así como el protocolo biológico para generar bibliotecas emparejadas.
Abstract
La capacidad de amplificar y la secuencia de DNA o RNA de pequeñas muestras partidas sólo se ha logrado en los últimos cinco años. Desafortunadamente, los protocolos estándar para generar genómica o transcriptómicos bibliotecas son incompatibles y los investigadores deben optar por la secuencia de DNA o RNA de una muestra particular. Gel-seq soluciona este problema permitiendo a los investigadores a preparar simultáneamente las bibliotecas de DNA y RNA a partir de 100-1000 células usando un dispositivo simple de hidrogel. Este artículo presenta un enfoque detallado para la fabricación del dispositivo así como el protocolo biológico para generar bibliotecas emparejadas. Hemos diseñado Gel-seq para que pudiera ser fácilmente implementado por otros investigadores; muchos laboratorios de genética ya tienen el equipo necesario para reproducir la fabricación de dispositivo seq Gel. Nuestro protocolo emplea comúnmente utilizado kits para ambos toda transcripción amplificación (WTA) y preparación de la biblioteca, que también es probable que sean familiares a los investigadores ya versados en la generación genómicos y transcriptómicos bibliotecas. Nuestro enfoque permite a los investigadores a llevar el poder de la secuenciación de ADN y ARN en una sola muestra sin dividir y con insignificante coste añadido.
Introduction
Próxima generación sequencing (NGS) ha tenido un impacto profundo en la manera en que se lleva a cabo la investigación genética. Cuando los investigadores se centraron una vez en secuenciar el genoma de una especie entera, ahora es posible secuenciar el genoma de un solo tumor o incluso una sola celda en un experimento. 1 NGS también ha hecho rentable a los transcritos de RNA encontrados dentro de una célula, un conjunto de datos conocidos como el transcriptoma de la secuencia. La capacidad de amplificar y la secuencia de DNA o RNA de pequeñas muestras partidas sólo se ha logrado en los últimos cinco años. 2 , 3 , 4 Desafortunadamente, protocolos estándar son incompatibles y los investigadores deben optar por la secuencia de DNA o RNA de una muestra determinada. Cuando una partida muestra es suficientemente grande, puede dividirse por la mitad. Sin embargo, en escalas más pequeñas, pérdida de material debido a la división de las muestras puede afectar la calidad de la biblioteca, y puesta en común de las muestras puede promedio de variaciones interesantes entre las células. 5 además, los investigadores están cada vez más interesados en el examen de las muestras que no pueden dividirse, como las células o las biopsias de pequeño tumor heterogéneo. 6
Para resolver este problema, tres protocolos recientemente se han desarrollado para la secuencia de ADN y ARN de la misma muestra partida: Gel-seq7, G & T-seq8y DR-seq9. Este artículo presenta un protocolo detallado para Gel-seq, que puede utilizarse al mismo tiempo generar bibliotecas de ADN y ARN desde tan sólo 100 células en el insignificante costo adicional. El aspecto novedoso de Gel-seq es la capacidad para separar el ADN y el ARN basado exclusivamente en el tamaño utilizando matrices de hidrogel de bajo costo. La innovación principal del protocolo Gel-Seq es la separación física de la DNA del RNA. Esta separación se logra utilizando electroforéticamente una combinación de membranas de poliacrilamida que se aprovechan de las diferencias de tamaño entre estas moléculas. Para poner estas diferencias de tamaño en contexto, considerar cómo son imágenes de ADN y ARN: ADN existe en la escala de micrones y puede verse con los microscopios tradicionales, ARN existe en la escala de nanómetros y debe ser reflejada mediante técnicas complejas como cryo-electrón microscopia. 10
El enfoque a la separación de ADN y ARN en este protocolo se muestra en la figura 1. El panel izquierdo muestra ADN y ARN liberan flotando en la solución cerca de una membrana. Cuando se aplica un campo eléctrico, como se muestra en el panel derecho, DNA y RNA experimentan una fuerza electroforética que induce la migración a través de la membrana. Ajuste las propiedades de la membrana, hemos creado una membrana semipermeable que separa el ADN del ARN. Las moléculas de ADN son empujadas contra la membrana, pero se enredaron en el borde debido a su gran tamaño. Pequeñas moléculas de ARN, por otra parte, pueden reconfigurar y tejer su camino a través de la membrana. Este proceso, conocido como reptation, es similar a la forma de que una serpiente se mueve a través de la hierba. Eventualmente estas moléculas de RNA son detenidas por una segunda membrana alta densidad que es demasiado difícil para los polímeros más pequeños (> 200 pares de bases) a retorcerse a través. Una vez separadas físicamente, ADN y ARN pueden recuperado y procesado para generar información sobre el genoma y el transcriptoma. Mientras que podemos separar el ADN y el ARN, hemos encontrado mejores resultados se obtienen si el ARN es revés transcrito a cDNA antes de la separación. Los híbridos de ARN cDNA son más estables que el RNA solo y todavía pueden pasar a través de la membrana de baja densidad.
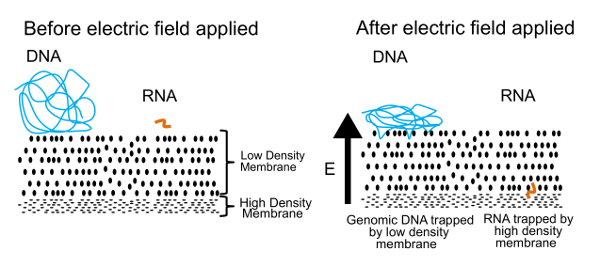
Figura 1 . Principio de funcionamiento de gel-seq. El principio subyacente que se utiliza para separar físicamente los ADN y ARN. En un campo eléctrico aplicado, las moléculas de ARN pequeñas migran a través de la membrana baja densidad pero grandes moléculas de ADN están atrapadas en la superficie. Esta figura fue reproducida de ref. 7 con permiso de la Real Sociedad de química. Haga clic aquí para ver una versión más grande de esta figura.
Este documento describe en detalle tanto la fabricación del dispositivo de Gel-seq y el protocolo biológico para generar emparejado bibliotecas de DNA y RNA. Un resumen de ambos se muestra en la figura 2. El dispositivo es fabricado por capas tres diferentes densidades geles de poliacrilamida uno encima del otro en un proceso similar a la creación de geles de apilamientos estándar. 11 el protocolo biológico comienza con 100-1000 células suspendidas en PBS. Las células son sometidas a lisis y el ARN se convierte en ADNc antes de que el dispositivo se utiliza para separar el ADN genómico de los híbridos de ARN cDNA. Después de la separación y recuperación, genómico y transcriptómicos bibliotecas son preparadas mediante un proceso que sigue de cerca el protocolo de kit de preparación de biblioteca estándar de todo el genoma. Mayores detalles sobre el desarrollo y validación de Gel-seq se pueden leer en el laboratorio en una publicación de Chip “seq Gel: todo el genoma y transcriptoma ordenando por la simultánea baja ADN y ARN biblioteca preparación utilizando barreras semipermeables hidrogel .” 7
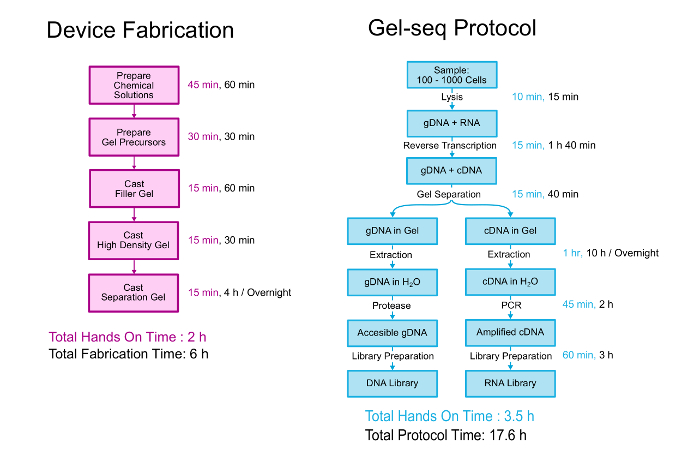
Figura 2 . Protocolo de gel-seq. Un resumen de los pasos para fabricar el dispositivo de Gel-seq y el protocolo generados bibliotecas pares de ADN y ARN. Porciones de esta figura fueron reproducidas de 7 Ref. con permiso de la Real Sociedad de química. Haga clic aquí para ver una versión más grande de esta figura.
Para generar bibliotecas de ADN y ARN de las células, los investigadores deben considerar el uso de G & T-seq o siguientes Dr. G & T-Seq, como Gel-seq, se basa en una separación física del ARN de la DNA genomic. Este enfoque se basa en ARN mensajero (ARNm) 3′ cofia cola como Diana abatible. El mRNA es capturado en una tira magnética utilizando una cartilla de oligo-dT biotinilado. Una vez que el ARNm se ha capturado las cuentas se sujetan con un imán y el sobrenadante que contiene el ADN genómico se puede quitar y transfirió a otro tubo. Después de esta separación física es completa, se pueden generar bibliotecas separadas del mRNA y de ADN. 8 este enfoque funciona bien si el ARN de interés es la cofia, sin embargo no puede utilizarse para el estudio de transcripciones no cofia, como ARN ribosomal, tRNA o ARN de procariotas.
DR-seq se basa en un paso de amplificación previa donde se amplifican ADN y ADNc derivado del RNA en el mismo tubo. La muestra es entonces dividida en dos y procesada paralelamente preparar bibliotecas de DNA y RNA-seq. Para distinguir entre ADN genómico y cDNA derivada del RNA, DR-seq tiene un enfoque computacional. Secuencias que están presentes sólo los exones computacionalmente se suprimen en los datos de ADN genómicos, como los pudieran haber originado de DNA o RNA. 9 una ventaja de este enfoque es que el DNA y cDNA del ARN necesitan no ser físicamente separados como se hace en Gel-seq y G & T-SS. El inconveniente, sin embargo, es que DR-seq requiere un conocimiento a priori del genoma y transcriptoma (es decir, exons y introns), puede no ser ideal para aplicaciones como secuenciación de núcleos, en que muchas transcripciones no son aún completamente empalmado y todavía contienen intrones. 12
El aspecto novedoso de Gel-seq es la capacidad para separar el ADN y el ARN en cientos de células basadas exclusivamente en el tamaño. Este método requiere de ningún conocimiento a priori del genoma o transcriptoma, es robusta contra empalme incompleto y no se limita a las transcripciones de poli-adenylated. Para aplicaciones donde un investigador puede comenzar con por lo menos 100 células, seq Gel proporciona un enfoque sencillo utilizando materiales baratos y extensamente disponible.
Protocol
Representative Results
Discussion
Hay varios pasos críticos asociados con la fabricación de dispositivo seq Gel así como el propio protocolo. Durante la fabricación, recomendamos a partir de los espesores de capa prescritos para las distintas regiones del gel. Pasamos mucho tiempo prueba opciones de fabricación diferentes y el protocolo descrito aquí produce los mejores dispositivos para los cassettes enumerados en la tabla de materiales y reactivos. Si los investigadores utilizan un sistema de cassette alternativos, pueden encontr…
Declarações
The authors have nothing to disclose.
Acknowledgements
Fondos para este trabajo fue proporcionado por la Universidad de San Diego, el programa nacional de ciencia Fundación postgrado investigación beca, concesión de NIH R01-HG007836 y por el Ministerio Coreano de la ciencia, TIC y planeación de futuro.
Versiones anteriores de varias figuras fueron publicadas primero en “Hoople, G. D. et al. Gel-seq: todo el genoma y transcriptoma ordenando por la simultánea baja ADN y ARN biblioteca preparación utilizando barreras semipermeables hidrogel. Laboratorio en un Chip 17, 2619-2630, doi:10.1039 / c7lc00430c (2017). ” Laboratorio en un Chip ha sancionado la reutilización de figuras en esta publicación.
Materials
| Reagents | |||
| Acrylamide Monomer | Sigma Aldrich | A8887-100G | |
| Ammonium Persulfate | Sigma Aldrich | A3678-25G | |
| Ampure XP Beads | Beckman Coulter | A63880 | Referred to in the text as solid phase reversible immobilization (SPRI) beads |
| DNA Gel Loading Dye (6x) | ThermoFisher Scientific | R0611 | Referred to in the text as 6X loading dye |
| Ethyl alcohol | Sigma Aldrich | E7023-500ML | |
| KAPA SYBR FAST One-Step qRT-PCR Kits | Kapa BioSystems | 7959613001 | Referred to in the text as 2X qPCR mix |
| N,N′-Methylenebis(acrylamide) | Sigma Aldrich | 146072-100G | Also known as bis-acrylamide |
| NexteraXT DNA Library Preparation Kit (referred to in the text as library preparation kit) | Illumina | FC-131-1024 | Includes: TD (referred to in the text as transposase buffer), ATM (referred to in the text as transposase), NT (referred to in the text as transposase stop buffer), and NPM (Referred to in the text as library prep PCR mix) |
| Nuclease Free Water | Millipore | 3098 | |
| Protease | Qiagen | 19155 | |
| SMART-Seq v4 Kit (referred to in the text as whole-transcript amplification (WTA) kit) | Takara/Clontech | 634888 | Includes: Lysis buffer, RNase inhibitor, 3’ SMART-Seq CDS Primer II A (referred to in the text as RT primer), 5X Ultra Low First Strand Buffer (referred to in the text as first strand buffer), SMART-Seq v4 Oligonucleotide (referred to in the text as template switch oligonucleotide (TSO)), SMART-Scribe Reverse Transcriptase (referred to in the text as reverse transcriptase), and PCR Primer II A (referred to in the text as cDNA PCR primer) |
| Random hexamer with WTA adapter | IDT | n/a | 5′-AAGCAGTGGTATCAACGCAGAGTAC-NNNNNN-3′ |
| Sucrose | Sigma Aldrich | S0389-500G | |
| TEMED | Sigma Aldrich | T9281-25ML | |
| DNA AWAY Surface Decontaminant | ThermoFisher Scientific | 7010PK | Referred to in the text as DNA removal product |
| Tris-Borate-EDTA buffer (10X concentration) | Sigma Aldrich | T4415-1L | |
| SYBR Gold Nucleic Acid Gel Stain (10,000X Concentrate in DMSO) | ThermoFisher Scientific | S11494 | Referred to in the text as gel stain |
| Equipment | |||
| BD Precisionglide syringe needles, gauge 18 | Sigma Aldrich | Z192554 | Any equivalent hardware is acceptable |
| Branson CPX series ultrasonic bath | Sigma Aldrich | Z769363 | Any equivalent hardware is acceptable |
| Empty Gel Cassettes, mini, 1.0 mm | ThermoFisher Scientific | NC2010 | Any equivalent hardware is acceptable |
| Mesh Filter Plate – Corning HTS Transwell 96 well permeable supports – 8.0 µm pore size | Sigma Aldrich | CLS3374 | Referred to in the text as 8 um mesh filter plate |
| PowerPac HC Power Supply | Bio-Rad | 1645052 | Any equivalent hardware is acceptable |
| Qubit Fluorometer | ThermoFisher Scientific | Q33216 | Any equivalent hardware is acceptable |
| Vacufuge Concentrator | Eppendorf | 22822993 | Any equivalent hardware is acceptable |
| XCell SureLock Mini-Cell system | ThermoFisher Scientific | EI0001 | Any equivalent hardware is acceptable |
| Bio-Rad CFX96 Touch Real-Time PCR Detection System | Bio-Rad | 1855195 | Any equivalent hardware is acceptable |
| Amersham UVC 500 Ultraviolet Crosslinker | GE Healthcare Life Sciences | UVC500-115V | Discontinued, any equivalent hardware is acceptable |
| Gel Doc XR+ Gel Documentation System | Bio-Rad | 1708195 | Referred to in the text as gel imager |
| Dark Reader Transilluminator | Clare Chemical Research | DR89 | Referred to in the text as UV transilluminator |
| Ultrasonic Bath | Bransonic | 1207K35 | Any equivalent ultrasonic bath is acceptable. |
Referências
- Mawy, T. Single-cell sequencing. Nat Methods. 11 (1), 18 (2014).
- Gole, J., et al. Massively parallel polymerase cloning and genome sequencing of single cells using nanoliter microwells. Nat Biotech. 31 (12), 1126-1132 (2013).
- Sasagawa, Y., et al. Quartz-Seq: a highly reproducible and sensitive single-cell RNA-Seq reveals non-genetic gene expression heterogeneity. Genome Biol. 14 (4), 31 (2013).
- Ramsköld, D., et al. Full-length mRNA-Seq from single-cell levels of RNA and individual circulating tumor cells. Nat Biotechnol. 30 (8), 777-782 (2012).
- Shapiro, E., Biezuner, T., Linnarsson, S. Single-cell sequencing-based technologies will revolutionize whole-organism science. Nat Rev Genet. 14 (9), 618-630 (2013).
- Navin, N., et al. Tumour evolution inferred by single-cell sequencing. Nature. 472 (7341), 90-94 (2011).
- Hoople, G. D., et al. Gel-seq: whole-genome and transcriptome sequencing by simultaneous low-input DNA and RNA library preparation using semi-permeable hydrogel barriers. Lab Chip. 17, 2619-2630 (2017).
- Macaulay, I. C., et al. G&T-seq: parallel sequencing of single-cell genomes and transcriptomes. Nat Methods. 12 (6), 519-522 (2015).
- Dey, S. S., Kester, L., Spanjaard, B., Bienko, M., van Oudenaarden, A. Integrated genome and transcriptome sequencing of the same cell. Nat Biotechnol. 33 (3), 285-289 (2015).
- Gopal, A., Zhou, Z. H., Knobler, C. M., Gelbart, W. M. Visualizing large RNA molecules in solution. RNA. 18 (2), 284-299 (2012).
- . SDS-PAGE Gel. Cold Spring Harb Protoc. 2015 (7), 087908 (2015).
- Lake, B. B., et al. Neuronal subtypes and diversity revealed by single-nucleus RNA sequencing of the human brain. Science. 352 (6293), (2016).
- Lee, P. Y., Costumbrado, J., Hsu, C. -. Y., Kim, Y. H. Agarose Gel Electrophoresis for the Separation of DNA Fragments. J Vis Exp. (62), e3923 (2012).
- Qubit 3.0 Fluorometer Manual. MAN0010866. Life Technologies Available from: https://tools.thermofisher.com/content/sfs/manuals/qubit_3_fluorometer_man.pd (2017)
- Vitak, S. A., et al. Sequencing thousands of single-cell genomes with combinatorial indexing. Nat Methods. 14 (3), 302-308 (2017).
- Illumina. . Nextera ® XT DNA Library Preparation Kit. , (2017).
- Takara Bio USA Inc. . SMART-Seq v4 Ultra Low Input RNA Kit for Sequencing User Manual. , (2016).
- Kurimoto, K., Yabuta, Y., Ohinata, Y., Saitou, M. Global single-cell cDNA amplification to provide a template for representative high-density oligonucleotide microarray analysis. Nat Protoc. 2 (3), 739-752 (2007).

