इम्यूनोफ्लोरेसेंस द्वारा डीएनए मरम्मत प्रोटीन इंटरैक्शन का दृश्य
Summary
डीएनए क्षति के बाद, मानव कोशिकाओं को उनके जीनोम की अखंडता को बहाल करने के लिए आवश्यक मरम्मत रास्तों को सक्रिय । यहां, हम डीएनए मरम्मत प्रोटीन का पता लगाने, उनके स्थानिक और लौकिक भर्ती का विश्लेषण करने और डीएनए क्षति की साइटों पर प्रोटीन-प्रोटीन बातचीत से पूछताछ करने में मदद करने के साधन के रूप में अप्रत्यक्ष इम्यूनोफ्लोरेसेंस की विधि का वर्णन करते हैं ।
Abstract
स्तनधारी कोशिकाओं को लगातार रसायनों, विकिरणों, और स्वाभाविक रूप से होने वाले मेटाबोलिक बाय-उत्पादों के संपर्क में आते हैं, जो विशिष्ट प्रकार के डीएनए अपमान बनाते हैं। जेनोटॉक्सिक एजेंट डीएनए बैकबोन को नुकसान पहुंचा सकते हैं, इसे तोड़ सकते हैं, या व्यक्तिगत ठिकानों की रासायनिक प्रकृति को संशोधित कर सकते हैं। डीएनए अपमान के बाद, डीएनए क्षति प्रतिक्रिया (डीडीआर) रास्ते सक्रिय होते हैं और मरम्मत में शामिल प्रोटीन की भर्ती की जाती है । नुकसान के प्रकार को भांपने और उचित मरम्मत प्रतिक्रिया को सक्रिय करने में कारकों की अधिकता शामिल है। डीडीआर कारकों को सही ढंग से सक्रिय करने और भर्ती करने में विफलता जीनोमिक अस्थिरता का कारण बन सकती है, जो कैंसर सहित कई मानव विकृतियों को रेखांकित करती है। डीडीआर प्रोटीन के अध्ययन जेनोटॉक्सिक दवा प्रतिक्रिया और दवा प्रतिरोध के सेलुलर तंत्र में अंतर्दृष्टि प्रदान कर सकते हैं।
वीवो मेंप्रोटीन की कल्पना करने के दो प्रमुख तरीके हैं: प्रत्यक्ष अवलोकन, फ्लोरोसेंट प्रोटीन के साथ ब्याज के प्रोटीन को टैग करके और निश्चित नमूनों पर लाइव इमेजिंग, या अप्रत्यक्ष इम्यूनोफ्लोरेसेंस द्वारा इसका पालन करके। जबकि फ्लोरोसेंटली टैग किए गए प्रोटीन का विज़ुअलाइज़ेशन समय के साथ सटीक निगरानी की अनुमति देता है, एन-या सी-टर्मिनस में प्रत्यक्ष टैगिंग प्रोटीन स्थानीयकरण या कार्य में हस्तक्षेप कर सकती है। उनके असंशोधित, अंतर्जात संस्करण में प्रोटीन का अवलोकन पसंद किया जाता है। जब डीएनए मरम्मत प्रोटीन डीएनए अपमान करने के लिए भर्ती कर रहे हैं, उनकी एकाग्रता स्थानीय रूप से बढ़ जाती है और वे समूहों, या “foci” फार्म, कि विशिष्ट एंटीबॉडी का उपयोग कर अप्रत्यक्ष इम्यूनोफ्लोरेसेंस द्वारा कल्पना की जा सकती है ।
हालांकि प्रोटीन फोसी का पता लगाने से प्रत्यक्ष बातचीत का एक निश्चित प्रमाण प्रदान नहीं होता है, कोशिकाओं में प्रोटीन का सह-स्थानीयकरण इंगित करता है कि वे क्षति की साइट पर फिर से समूहीकृत होते हैं और जटिल गठन के लिए आवश्यक घटनाओं के अनुक्रम के बारे में सूचित कर सकते हैं। एक प्रोटीन के जंगली प्रकार या उत्परिवर्ती संस्करणों को व्यक्त करने वाली कोशिकाओं में फोसी स्थानिक ओवरलैप का सावधानीपूर्वक विश्लेषण डीएनए मरम्मत कार्य के लिए महत्वपूर्ण कार्यात्मक डोमेन पर कीमती सुराग प्रदान कर सकता है। अंतिम, प्रोटीन का सह-स्थानीयकरण संभावित प्रत्यक्ष बातचीत को इंगित करता है जिसे कोशिकाओं में सह-इम्यूनोप्रिपिपिटेशन द्वारा सत्यापित किया जा सकता है, या शुद्ध प्रोटीन का उपयोग करके प्रत्यक्ष पुलडाउन।
Introduction
मानव कोशिकाओं को लगातार विभिन्न मूल के डीएनए हानिकारक एजेंटों की एक किस्म के संपर्क में हैं। बहिर्जात स्रोतों में ज्यादातर विकिरण, रसायन (कीमोथैरेपी एजेंटों और कुछ एंटीबायोटिक दवाओं सहित) और वायरस के संपर्क में होते हैं, जबकि मुख्य अंतर्जात स्रोतों में डीएनए प्रतिकृति और ऑक्सीडेटिव तनाव में त्रुटियां शामिल हैं। जेनोटॉक्सिक एक्सपोजर का सीधा प्रभाव तनाव और एक्सपोजर खुराक के आधार पर एक संशोधित आधार से संभावित घातक डीएनए डबल-स्ट्रैंड ब्रेक (डीएसबी) तक हो सकता है। अंततः, बिना मरम्मत या गलत मरम्मत किए गए डीएनए क्षति से उत्परिवर्तन, जीनोमिक पुनर्व्यवस्था, जीनोम अस्थिरता का संचय हो सकता है और अंततः कार्सिनोजेनेसिस 1 का कारणबन सकताहै। स्तनधारी कोशिकाओं ने विशिष्ट प्रकार के डीएनए क्षति2, 3,को पहचानने और उन्हें समय पर फैशन में मरम्मत करने के लिए जटिलरास्ते विकसित किए हैं, जो सेल चक्र प्रगति के साथ सिंक्रोनाइज्ड हैं।
आयनीकरण विकिरण (आईआर) डीएनए डबल हेलिक्स को नुकसान पहुंचाता है और डीएनए क्षति के सबसे हानिकारक रूपों में से एक डबल-स्ट्रैंड ब्रेक (डीएसबी) बनाता है। एमआरएन (एमआरएन (एमआरए11, आरए 50, एनबीएस 1) जटिल कार्य डीएनए के सेंसर के रूप में समाप्त होता है और प्रोटीन किनेज़ एटैक्सिया तेलंगिए म्यूटेड (एटीएम)4,,5को सक्रिय करता है। डीएनए समाप्त होता है द्वारा एटीएम के प्रारंभिक सक्रियण के बाद, एटीएम को तोड़ने की साइट पर DDR घटनाओं का झरना चलाता है, एक महत्वपूर्ण घटना के साथ शुरू, हिस्टोन संस्करण H2AX6के फॉस्फोरिलेशन । अवशेष S139 पर एच2एक्स फॉस्फोरिलेशन इसे γH2AX में सक्रिय करता है, जो डीएनए घाव,,6, 7,8,9 के आसपास मेगाबेस तक फैले,7हुएहैं। इस घटना से डीएनए पहुंच बढ़ जाती है, जिससे अन्य डीएनए मरम्मत प्रोटीन7की भर्ती और संचय होता है । क्योंकि γH2AX बहुतायत से और विशेष रूप से DSBs आसपास प्रेरित है, यह आसानी से विशिष्ट एंटीबॉडी का उपयोग कर कल्पना की जा सकती है, और आमतौर पर डीएनए मरम्मत क्षेत्र में DSBs के लिए एक किराए मार्कर के रूप में प्रयोग किया जाता है । एक बार तोड़ संकेत दिया है, कोशिकाओं को अपने डीएनए की मरंमत के रास्ते सक्रिय और डीएनए क्षति की प्रक्रिया । प्रोटीन एमडीसी1 (डीएनए क्षति चेकपॉइंट प्रोटीन 1 का मध्यस्थ) सीधे10γH2AX बांधता है , एटीएम11 के साथ और एनबीएस112,13के साथ भी बातचीत करता है । यह डीएसबी में एमआरएन कॉम्प्लेक्स की एकाग्रता बढ़ाने और एक सकारात्मक एटीएम फीडबैक लूप शुरू करने में योगदान देता है। ब्रेक की मरम्मत होने के बाद γH2AX को तेजी से हटा दिया जाता है, नतीजतन, डीएसबी क्लीयरेंस की निगरानी की अनुमति दी जाती है। माइक्रोस्कोपी के बाद, समय के साथ γH2AX में कमी अवशिष्ट टूटता है और डीएनए मरम्मत दक्षता का एक अप्रत्यक्ष माप प्रदान करता है ।
यूकेरियोटिक कोशिकाएं कई रास्तों से डीएसबी की मरम्मत कर सकती हैं, दो मुख्य गैर-मुताबिक़ एंड-जॉइनिंग (एनएचईजे) और मुताबिक़ पुनर्संयोजन (एचआर)(चित्रा 1)जा रहे हैं। एनएचईजे अनिवार्य रूप से विस्तारित होमोलॉजी के उपयोग के बिना डीएनए डबल-स्ट्रैंड समाप्त होता है और पूरे सेल चक्र14,,15में संचालित होता है। मानव संसाधन एस और जी 2 चरणों के दौरान प्रमुख हो जाता है, और अन्यथा दमित होता है, क्योंकि इसे मरम्मत14,,16के लिए एक समरूप टेम्पलेट के रूप में एक बहन क्रोमटिड की आवश्यकता होती है। एनएचईजे और एचआर के बीच पाथवे विकल्प न केवल सिस्टर क्रोमैटिड की शारीरिक निकटता पर निर्भर करता है, बल्कि डीएनए एंड रिसेक्शन17के विस्तार पर भी निर्भर करता है, जो एनएचईजे को रोकता है।
होमोलॉजी-निर्भर डीएसबी मरम्मत 3 ‘सिंगल-स्ट्रैंड डीएनए (एसएसडीएनए) पूंछ उत्पन्न करने के लिए समाप्त होता है, एक प्रक्रिया जिसे 5′-3’ रिसेक्शन के रूप में संदर्भित किया जाता है, ब्रेक से 5 ‘स्ट्रैंड के न्यूक्लियोलिटिकिक क्षरण द्वारा शुरू किया जाता है। एमआरएन परिसर डीएनए एंड रिसेक्शन शुरू करता है और आगे की रीसेक्शन को बीएलएम/एक्सओ1 (ब्लूम सिंड्रोम प्रोटीन/एक्सोन्यूलेस 1) या बीएलएम/डीएनए2 (डीएनए प्रतिकृति,एटीपी-निर्भर हेलीकेस/न्यूक्लियेज)18,19,20,,,21, 22के संयोजन में संसाधित कियाजाताहै ।, डीएनए एंड रिसेक्शन को सीटीआईपी (सीटीबीपी-इंटरैक्टिंग प्रोटीन) द्वारा एमआरएन कॉम्प्लेक्स 23 के साथअपनी सीधी बातचीत और बीआरसीए1 (स्तन कैंसर टाइप 1 संवेदनशीलता प्रोटीन)24, 25,के साथ अपनी भर्ती के माध्यम से बढ़ायाजाताहै । प्रतिकृति प्रोटीन ए (आरपीए) तुरंत उजागर एसएसडीएनए से बांधता है और फिर पुनः संयोजन प्रोटीन RAD51 द्वारा विस्थापित किया जाता है ताकि एक न्यूकोप्रोटीन फिलामेंट बन,सके जो26, 27,28केमुताबिक़ खोज और कतरा आक्रमण को उत्प्रेरित करता है।28
resection की शुरुआत मरम्मत मार्ग विकल्प के लिए एक महत्वपूर्ण कदम है। एक बार resection शुरू हो गया है, डीएनए समाप्त होता है Ku70/Ku80 heterodimer (NHEJ मार्ग के घटक) द्वारा बाध्यकारी के लिए गरीब सब्सट्रेट्स बन जाते हैं और कोशिकाएं एचआर17,29,,30के लिए प्रतिबद्ध हैं ।, Ku70/Ku80 heterodimer DSB समाप्त होता है, डीएनए-PKcs और p53 बाध्यकारी प्रोटीन 1 (53BP1)29,30की भर्ती करनेकेलिए बांधता है । 53BP1 जी 1 में पुनर्सेक्शन के लिए एक बाधा के रूप में कार्य करता है, इस प्रकार एनएचईजे31, 32,को बढ़ावा देतेहुएमानव संसाधन को अवरुद्ध करता है, लेकिन इसे एस चरण में बीआरसीए1-निर्भर तरीके से हटा दिया जाता है, नतीजतन रिसेक्शन33, 34,होनेकीअनुमति देता है। इसलिए, 53BP1 और BRCA1 डीएसबी मरम्मत में विरोधी भूमिकाएं निभाते हैं, जिसमें 53बीपी1 एनएचईजे फैसिलिटेटर है जबकि बीआरसीए1 मानव संसाधन के माध्यम से मरम्मत के लिए ब्रेक को सक्षम करने के लिए कार्य करता है।
प्रयोगशाला में, डीएसबी गठन आयनीकरण विकिरण (आईआर) द्वारा प्रेरित किया जा सकता है। जबकि यह उदाहरण 4 जीवाई, 1 जीवाई और 2 जीवाई की उच्च खुराक का उपयोग करता है, जो प्रचुर मात्रा में प्रोटीन द्वारा फोसी गठन के विश्लेषण के लिए उपयुक्त डीएसबी की एक महत्वपूर्ण मात्रा भी बनाते हैं। यह ध्यान रखना महत्वपूर्ण है कि उपयोग किए जाने वाले विकिरण के प्रकार और खुराक डीएनए और कोशिका में विभिन्न घावों का कारण बन सकते हैं: जबकि आईआर डीएसबी को प्रेरित करता है, यह एकल स्ट्रैंड ब्रेक या बेस संशोधन का कारण बन सकता है (विकिरण रैखिक ऊर्जा हस्तांतरण (एलईटी) और डीएनए क्षति के प्रकार पर संदर्भ के लिए35,,36 देखें)। आयनीकरण विकिरण-प्रेरित फोसी (आईआरआईएफ) गठन और उनकी मंजूरी के काइनेटिक्स का निर्धारण करने के लिए, जो सक्रिय डीडीआर,8, 9,37, 38के नुकसान और उत्क्रमण की मरम्मत का संकेत देता है, आयनीकरण विकिरण के बाद विभिन्न समय बिंदुओं पर फोसीगठनकी निगरानी की जा सकती है।,9, सक्रियण और सभी प्रमुख डीएनए क्षति प्रोटीन की मंजूरी के समय३९जाना जाता है, और कई प्रमुख घटनाओं के किराए मार्कर के रूप में जांच कर रहे हैं । उदाहरण के लिए, PRPA, जो ssDNA के लिए उच्च आत्मीयता के पास तोड़ resection के एक किराए के रूप में प्रयोग किया जाता है, MRN प्रोटीन (MRE11, RAD50, NBS1) और exonucleases भी resection दक्षता का आकलन करने के लिए इस्तेमाल किया जा सकता है । जबकि RAD51, BRCA1, BRCA2 (स्तन कैंसर प्रकार 2 संवेदनशीलता प्रोटीन), और PALB2 (साथी और BRCA2 के स्थानीयता) मानव संसाधन दक्षता का मूल्यांकन करने के लिए निगरानी की जा सकती है, केयू प्रोटीन या 53BP1 की उपस्थिति, NHEJ(चित्रा 1)के मार्कर के रूप में उपयोग किया जाता है ।
डीएनए मरम्मत मशीनरी के प्रोटीन के रूप में एक दूसरे को तोड़ने के लिए भर्ती और सुपर परिसरों में इकट्ठा, डीएनए प्रोटीन और प्रोटीन प्रोटीन बातचीत समय के साथ अपने व्यक्तिगत स्थानीयकरण का पालन करके अनुमानित किया जा सकता है और प्रोटीन के सह स्थानीयकरण का विश्लेषण, के रूप में सेल४०,४१,,४२में ओवरलैपिंग संकेतों द्वारा कल्पना ।, सेल लाइनों में, जीनोम संपादन के माध्यम से या प्लाज्मिड-आधारित म्यूटेंट की अधिकव्यक्तता के माध्यम से विशिष्ट डीएनए मरम्मत जीन में बिंदु उत्परिवर्तन या विलोपन की शुरूआत, विशिष्ट अवशेषों की जांच और डीएनए क्षति की मान्यता में उनकी संभावित भूमिका की अनुमति देती है (उदाहरण के लिए, γH2AX के साथ सह-स्थानीयकरण) या जटिल असेंबली (सह-स्थानीयकरण दूसरे, या कई, प्रोटीन के साथ), साथ ही डीएनए मरम्मत पर उनके प्रभाव। यहां, हम समय के साथ γH2AX फोसी का पालन करके डीएसबी के गठन और संकल्प की जांच करने के लिए एक मतलब के रूप में अप्रत्यक्ष इम्यूनोफ्लोरेसेंस का उपयोग करते हैं। हम डीएसबी मरम्मत में एक प्रमुख खिलाड़ी द्वारा फोसी गठन और सह-स्थानीयकरण विश्लेषण का एक उदाहरण भी प्रस्तुत करते हैं: p53 बाध्यकारी प्रोटीन 1 (53BP1)32। जैसा कि पहले उल्लेख किया गया है, 53BP1 को डीएनए मरम्मत मार्ग विकल्प के लिए केंद्रीय माना जाता है। 53BP1 संचय और γH2AX के साथ इसके सह-स्थानीयकरण के बाद सेल चक्र चरण, डीएनए क्षति संचय, और DSBs की मरम्मत के लिए इस्तेमाल किया मार्ग के बारे में कीमती जानकारी प्रदान करता है । अप्रत्यक्ष इम्यूनोलोकेलाइजेशन का उद्देश्य सेल लाइनों में डीएनए क्षति की मरम्मत की दक्षता का आकलन करना है, इस अध्ययन में आईआर के बाद, या कोशिका में विभिन्न तनावों के संपर्क में आने के बाद, डीएनए क्रॉसलिंकिंग से प्रतिकृति कांटा की रुकावट तक (डीएनए हानिकारक एजेंटों की एक सूची तालिका 1में प्रदान की जाती है)।
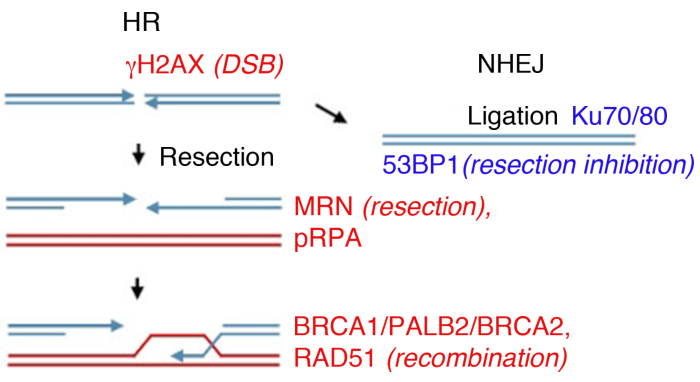
चित्रा 1: डीएनए डबल स्ट्रैंड ब्रेक (डीएसबी) मरम्मत के रास्ते।
डीएसबी मरम्मत में दो प्रमुख मार्ग शामिल हैं: मुताबिक़ पुनर्संयोजन (एचआर, बाएं) और गैर-मुताबिक़ एंड-जॉइनिंग (एनएचईजे, दाएं)। ब्रेक के बाद, प्रोटीन ब्रेक (γH2AX) को चिह्नित करने के लिए सक्रिय हो जाते हैं, अंत रिसेक्शन (एमआरएन) में भाग लेते हैं, पुनः प्राप्त एसएसडीएनए (PRPA) कोट करते हैं, पुनर्संयोजन को बढ़ावा देते हैं (बीआरसीए1, पाल्ब 2, बीआरसीए2, RAD51) या सीमा resection और NHEJ (53BP1) को बढ़ावा देते हैं। अन्य प्रोटीन क्षति मरम्मत में भाग लेते हैं, लेकिन सूचीबद्ध प्रोटीन नियमित रूप से अप्रत्यक्ष इम्यूनोफ्लोरेसेंस के बाद होते हैं। कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें ।
| डीएनए हानिकारक एजेंट | कार्रवाई का तंत्र | अनुशंसित खुराक |
| γ-रे/एक्स-रे | विकिरण कुछ अनियंत्रित सेलुलर प्रभावों के साथ डबल-फंसे ब्रेक का गठन |
1-4 जीए |
| 36 एआर आयन | विकिरण डबल फंसे ब्रेक का गठन |
270 केवी/माइक्रोन |
| α-कण | विकिरण डबल फंसे ब्रेक का गठन |
116 केवी/माइक्रोन |
| ब्लेओमाइसिन | डीएनए संश्लेषण का अवरोधक | 0.4-2 μg/mL |
| कैम्पटोथेसिन | टोपोसोमरेसी I का अवरोधक | 10-200 एनएम |
| सिस्प्लैटिन | एल्किलेटिंग एजेंट (उत्प्रेरण इंट्रास्ट्रैंड क्रॉसलिंक) |
0.25-2 माइक्रोन |
| डॉक्सोरुबिसिन | इंटरकैलिंग एजेंट टोपोसोमरेस द्वितीय का अवरोधक |
10-200 एनएम |
| ईटीओपोसाइड | टोपोसोमरेस द्वितीय का अवरोधक | 10 माइक्रोन |
| हाइड्रोक्सीयूरिया | डीएनए संश्लेषण का अवरोधक (राइबोन्यूक्लियोटाइड रिडक्शन द्वारा) |
10-200 माइक्रोन |
| मिथाइल मीथेनसुलफोनेट | एल्किलेटिंग एजेंट | 0.25-2 mM |
| माइटोमाइसिन सी | एल्किलेटिंग एजेंट | 0.25-2 माइक्रोन |
| पराबैंगनी (यूवी) प्रकाश | थाइमिडीन डिमर्स का गठन (डीएनए श्रृंखला की विकृति पैदा करना) |
50-100 mJ/सेमी2 |
तालिका 1: जेनोटॉक्सिक एजेंट। डीएनए हानिकारक एजेंटों के उदाहरण, कार्रवाई के उनके तंत्र और सुझाए गए काम एकाग्रता के आधार पर प्रेरित नुकसान ।
Protocol
Representative Results
Discussion
माइक्रोस्कोपी द्वारा डीएनए क्षति की मरम्मत के समय और दक्षता का विश्लेषण यह समझने के लिए आवश्यक साबित हुआ है कि डीएनए मरम्मत मशीनरी सामान्य कोशिकाओं में और कैंसर जैसी मानव विकृतियों में कैसे कार्य कर?…
Declarações
The authors have nothing to disclose.
Acknowledgements
इस काम को सैन एंटोनियो एरिया फाउंडेशन से अनुदान द्वारा समर्थित किया गया था। Mays कैंसर केंद्र एनसीआई कैंसर सेंटर समर्थन कोर अनुदान P30 CA054174 द्वारा समर्थित है । हम स्टीफन होलोवे को उनकी मदद के लिए शुक्रिया अदा करना चाहते हैं, जो अभिकर्षकों को सोर्सिंग करते हैं, और अंतरिक्ष और माइक्रोस्कोपी क्षमता प्रदान करने के लिए सुंग प्रयोगशाला ।
Materials
| 16% (v/v) paraformaldehyde (PFA) aqueous solution | Electron Microscopy Sciences | 15710 | Microscopy quality of the PFA ensures best images. If using "home-made PFA", filter prior to use. |
| Bovine serum albumin (BSA) | Sigma-Aldrich | A3059 | Heat-shock fraction is recommended, to avoid precipitation/background. |
| Coverglass #1, 18 mm round (coverslips) | Neuvitro | NC0308920 | Coverslips need to be cleaned and sterilized prior using, with HCl or ethanol. |
| DMEM, High Glucose [(+) 4.5 g/L D-Glucose, (+) L-Glutamine, (-) Sodium Pyruvate] | Gibco | 11965118 | Adjust the growing media to the needs of cell line used. |
| DPBS, no calcium, no magnesium | Gibco | 14190144 | PBS for tissue culture. |
| Ethylene glycol-bis(β-aminoethyl ether)-N,N,N′,N′-tetraacetic acid (EGTA) | Research Products International | E57060 | Nuclear extraction buffer. |
| Fetal Bovine Serum (FBS) | Life Technologies | 104370028 | The quality of FBS can be assessed by testing gH2AX foci formation. If traces of genotoxic endotoxin are present in the batch, gH2AX will be positive in the absence of stress. |
| Magnesium chloride (MgCl2) | Research Products International | M24000 | Nuclear extraction buffer. |
| Piperazine-N,N′-bis(2-ethanesulfonic acid) (PIPES) | Research Products International | P40150 | Nuclear extraction buffer. |
| SlowFade Diamond Antifade Mountant with DAPI | Invitrogen | S36973 | 300 nM DAPI with VECTASHIELD Antifade Mounting Medium can be used instead. |
| Sodium chloride (NaCl) | Research Products International | S23020 | Nuclear extraction buffer. |
| Sucrose | Research Products International | S24060 | Nuclear extraction buffer. |
| Superfrost Plus Microscope Slides | Fisherbrand | 1255015 | Polysine Slides can be used instead. |
| TC-Treated Multiple Well Plates, size 12 wells | Costar | 3513 | Seeding on coverslips is done in 12-wells plate. |
| Triton X-100 | AmericanBio | AB02025 | Nuclear extraction buffer. |
| TrypLE Express Enzyme (1X), No Phenol Red | Gibco | 12604021 | Trypsin-EDTA can be used instead. |
| Trypsin-EDTA (0.5%), No Phenol Red | Gibco | 15400054 | TrypLE can be used instead. |
Referências
- Prakash, R., Zhang, Y., Feng, W., Jasin, M. Homologous recombination and human health: the roles of BRCA1, BRCA2, and associated proteins. Cold Spring Harbor Perspectives in Biology. 7 (4), 016600 (2015).
- Jalan, M., Olsen, K. S., Powell, S. N. Emerging Roles of RAD52 in Genome Maintenance. Cancers (Basel). 11 (7), (2019).
- Oh, J., Symington, L. S. Role of the Mre11 Complex in Preserving Genome Integrity. Genes (Basel). 9 (12), (2018).
- Uziel, T., et al. Requirement of the MRN complex for ATM activation by DNA damage. The EMBO Journal. 22 (20), 5612-5621 (2003).
- Lee, J. H., Paull, T. T. ATM activation by DNA double-strand breaks through the Mre11-Rad50-Nbs1 complex. Science. 308 (5721), 551-554 (2005).
- Rogakou, E. P., Pilch, D. R., Orr, A. H., Ivanova, V. S., Bonner, W. M. DNA double-stranded breaks induce histone H2AX phosphorylation on serine 139. Journal of Biological Chemistry. 273, 5858-5868 (1998).
- Kinner, A., Wu, W., Staudt, C., Iliakis, G. γ-H2AX in recognition and signaling of DNA double-strand breaks in the context of chromatin. Nucleic Acids Research. 36 (17), 5678-5694 (2008).
- Martin, O. A., Pilch, D. R., Redon, C., Bonner, W. M. Histone H2AX in DNA damage repair. Cancer Biology & Therapy. 2 (3), 233-235 (2003).
- Rogakou, E. P., Boon, C., Redon, C., Bonner, W. M. Megabase chromatin domains involved in DNA double-strand breaks in vivo. Journal of Cell Biology. 146 (5), 905-916 (1999).
- Stucki, M., et al. MDC1 directly binds phosphorylated histone H2AX to regulate cellular responses to DNA double-strand breaks. Cell. 123 (7), 1213-1226 (2005).
- Lou, Z., et al. MDC1 maintains genomic stability by participating in the amplification of ATM-dependent DNA damage signals. Molecular Cell. 21 (2), 187-200 (2006).
- Chapman, J. R., Jackson, S. P. Phospho-dependent interaction between NBS1 and MDC1 mediate chromatin retention of the MRN complex at sites of DNA damage. EMBO Reports. 9 (8), 795-801 (2008).
- Melander, F., et al. Phosphorylation of SDT repeats in the MDC1 N terminus triggers retention of NBS1 at the DNA damage-modified chromatin. Journal of Cell Biology. 181 (2), 213-226 (2008).
- Branzei, D., Foiani, M. Regulation of DNA repair throughout the cell cycle. Nature Review. Molecular Cell Biology. 9 (4), 297-308 (2008).
- Chiruvella, K. K., Liang, Z., Wilson, T. E. Repair of double-strand breaks by end joining. Cold Spring Harbor Perspectives in Biology. 5 (5), 012757 (2013).
- Mehta, A., Haber, J. E. Sources of DNA double-strand breaks and models of recombinational DNA repair. Cold Spring Harbor Perspectives in Biology. 6 (9), 016428 (2014).
- Symington, L. S., Gautier, J. Double-strand break end resection and repair pathway choice. Annual Review of Genetics. 45, 247-271 (2011).
- Huertas, P. DNA resection in eukaryotes: deciding how to fix the break. Nature Structural & Molecular Biology. 17 (1), 11-16 (2010).
- Nimonkar, A. V., et al. BLM-DNA2-RPA-MRN and EXO1-BLM-RPA-MRN constitute two DNA end resection machineries for human DNA break repair. Genes & Development. 25 (4), 350-362 (2011).
- Garcia, V., Phelps, S. E. L., Gray, S., Neale, M. J. Bidirectional resection of DNA double-strand breaks by Mre11 and Exo1. Nature. 479 (7372), 241-244 (2011).
- Sturzenegger, A., et al. DNA2 cooperates with the WRN and BLM RecQ helicases to mediate long-range DNA end resection in human cells. Journal of Biological Chemistry. 289 (39), 27314-27326 (2014).
- Daley, J. M., Niu, H., Miller, A. S., Sung, P. Biochemical mechanism of DSB end resection and its regulation. DNA Repair. 32, 66-74 (2015).
- Sartori, A. A., et al. Human CtIP promotes DNA end resection. Nature. 450 (7169), 509-514 (2007).
- Chen, L., Nievera, C. J., Lee, A. Y. L., Wu, X. Cell cycle-dependent complex formation of BRCA1-CtIP-MRN is important for DNA double-strand break repair. Journal of Biological Chemistry. 283, 7713-7720 (2008).
- Yun, M. H., Hiom, K. CtIP-BRCA1 modulates the choice of DNA double-strand break repair pathway throughout the cell cycle. Nature. 459 (7245), 460-463 (2009).
- Sung, P., Klein, H. Mechanism of homologous recombination: mediators and helicases take on regulatory functions. Nature Review. Molecular Cell Biology. 7, 739-750 (2006).
- San Filippo, J., Sung, P., Klein, H. Mechanisms of eukaryotic homologous recombination. Annual Review of Biochemistry. 77, 229-257 (2008).
- Jasin, M., Rothstein, R. Repair of strand breaks by homologous recombination. Cold Spring Harbor Perspectives in Biology. 5 (11), 012740 (2013).
- Dynan, W. S., Yoo, S. Interaction of Ku protein and DNA-dependent protein kinase catalytic subunit with nucleic acids. Nucleic Acids Research. 26 (7), 1551-1559 (1998).
- Lieber, M. R. The mechanism of double-strand DNA break repair by the nonhomologous DNA end-joining pathway. Annual Review of Biochemistry. 79, 181-211 (2010).
- Cejka, P. DNA end resection: nucleases team up with the right partners to initiate homologous recombination. Journal of Biological Chemistry. 290 (38), 22931-22938 (2015).
- Mirman, Z., de Lange, T. 53BP1: a DSB escort. Genes & Development. 34, 7-23 (2020).
- Cao, L., et al. A selective requirement for 53BP1 in the biological response to genomic instability induces by BRCA1 deficiency. Molecular Cell. 35 (4), 534-541 (2009).
- Zimmermann, M., de Lange, T. 53BP1: Pro choice in DNA repair. Trends in Cell Biology. 24 (2), 108-117 (2014).
- Mavragani, I. V., Nikitaki, Z., Kalospyros, S. A., Georgakilas, A. G. Ionizing Radiation and Complex DNA Damage: From Prediction to Detection Challenges and Biological Significance. Cancers (Basel). 11 (11), (2019).
- Nikitaki, Z., et al. Measurement of complex DNA damage induction and repair in human cellular systems after exposure to ionizing radiations of varying linear energy transfer (LET). Free Radical Research. 50, 64-78 (2016).
- Redon, C., et al. Histone H2A variants H2AX and H2AZ. Current Opinion in Genetics & Development. 12 (2), 162-169 (2002).
- Fernandez-Capetillo, O., Lee, A., Nussenzweig, M., Nussenzweig, A. H2AX: the histone guardian of the genome. DNA Repair. 3 (8-9), 959-967 (2004).
- Paull, T. T., et al. A critical role for histone H2AX in recruitment of repair factors to nuclear foci after DNA damage. Current Biology. 10 (15), 886-895 (2000).
- Sy, S. M. H., Huen, M. S. Y., Chen, J. PALB2 is an integral component of the BRCA complex required for homologous recombination repair. Proceedings of the National Academy of Sciences. 106 (17), 7155-7160 (2009).
- Buisson, R., Masson, J. Y. PALB2 self-interaction controls homologous recombination. Nucleic Acids Research. 40 (20), 10312-10323 (2012).
- Belotserkovskaya, R., et al. PALB2 chromatin recruitment restores homologous recombination in BRCA1-deficient cells depleted of 53BP1. Nature Communications. 11 (1), 819 (2020).
- Betts, J. A., et al. Long noncoding RNAs CUPID1 and CUPID2 mediate breast cancer risk at 11q13 by modulating the response to DNA damage. American Journal of Human Genetics. 101 (2), 255-266 (2017).
- Dray, E., et al. Molecular basis for enhancement of the meiotic DMC1 recombinase by RAD51 associated protein 1 (RAD51AP1). Proceedings of the National Academy of Sciences. 108 (9), 3560-3565 (2011).

