免疫荧光DNA修复蛋白相互作用的可视化
Summary
在DNA损伤之后,人类细胞激活必要的修复途径,以恢复其基因组的完整性。在这里,我们描述了间接免疫荧光的方法,作为检测DNA修复蛋白,分析其空间和时间招募,并帮助查询DNA损伤点蛋白质-蛋白质相互作用的方法。
Abstract
哺乳动物细胞经常暴露在化学物质、辐射和自然产生的代谢产物中,这些产物会造成特定类型的DNA侮辱。基因毒性剂可以破坏DNA骨干,破坏它,或修改单个碱基的化学性质。在DNA侮辱之后,DNA损伤反应(DDR)途径被激活,参与修复的蛋白质被招募。在感知损坏类型和激活适当的维修响应时,涉及大量因素。未能正确激活和招募 DDR 因子可能导致基因组不稳定,这是包括癌症在内的许多人类疾病的基础。DDR蛋白的研究可以提供对基因毒性药物反应和细胞耐药性机制的见解。
体内蛋白质可视化有两 种主要方法:直接观察,通过用荧光蛋白标记感兴趣的蛋白质,然后通过活体成像或固定样品上的间接免疫荧光跟踪。虽然荧光标记蛋白质的可视化允许随着时间的推移进行精确监测,但 N-或 C-术语中的直接标记可能会干扰蛋白质的定位或功能。观察蛋白质的未修改,内源版本是首选。当DNA修复蛋白被招募到DNA侮辱中时,它们的浓度会局部增加,它们形成组,或”foci”,这些组可以通过使用特异性抗体的间接免疫荧光来可视化。
虽然蛋白质叶酸的检测不能提供直接相互作用的明确证据,但细胞中蛋白质的共同定位表明它们重新组合到损伤发生地,并可以告知复杂形成所需的事件序列。仔细分析表达野生类型或蛋白质突变版本的细胞的 foci 空间重叠,可以为对 DNA 修复功能重要的功能域提供宝贵的线索。最后,蛋白质的共同定位表明可能的直接相互作用,可以通过细胞中的共免疫沉淀或使用纯化蛋白质直接下拉来验证。
Introduction
人类细胞不断暴露在各种来源的DNA破坏剂中。外源主要包括接触辐射、化学品(包括化疗剂和某些抗生素)和病毒,而主要的内源包括DNA复制和氧化应激方面的错误。基因毒性暴露的直接影响范围从改良的碱基到潜在的致命DNA双链断裂(DSB),这取决于压力和暴露剂量。最终,未修复或修复不当的DNA损伤可导致突变的积累、基因组重组、基因组不稳定,并最终导致致癌1。哺乳动物细胞已经进化出复杂的途径来识别特定类型的DNA损伤2,2,3,并及时修复它们,与细胞周期进展同步。
电离辐射(IR)损害DNA双螺旋,并造成双链断裂(DSB),这是最有害的DNA损伤形式之一。MRN (MRE11, RAD50, NBS1) 复合功能作为 DNA 末端的传感器,并激活蛋白激酶 ataxia telangiectasia 突变 (ATM)4,5。在DNA端对ATM的初始激活后,ATM在断点触发一系列DDR事件,启动一个关键事件,即组蛋白变种H2AX6的磷酸化。在残留物S139上的H2AX磷酸化激活它到+H2AX,跨越区域,高达围绕DNA病变6,7,8,97,8的巨型碱。6这一事件增加了DNA的可访问性,导致其他DNA修复蛋白的招募和积累7。由于 +H2AX 是丰富和具体诱导周围的 DSB,它可以很容易地可视化使用特定的抗体,并通常用作 DSB 在 DNA 修复领域的代理标记。一旦中断被发出信号,细胞激活其DNA修复途径并处理DNA损伤。蛋白质MDC1(DNA损伤检查点蛋白1的中介)直接结合+H2AX10,与ATM11和NBS1 12,13,相互作用。它有助于提高 MRN 复合物在 DSB 的浓度,并启动正 ATM 反馈回路。•一旦修复断裂,快速清除H2AX,从而允许监控DSB间隙。其次是显微镜,随着时间的推移,μH2AX 的减少提供了残余断裂和 DNA 修复效率的间接测量。
真核细胞可以通过多种途径修复DSB,两个主要途径是非同源端连接(NHEJ)和同源重组(HR)(图1)。NHEJ基本上连接DNA双链结束不使用扩展同源和在整个细胞周期14,15运行。14,HR在S和G2阶段成为占主导地位,否则被压抑,因为它需要一个姐妹色度作为一个同源模板修复14,16。14,NHEJ和HR之间的路径选择不仅取决于姐妹色度的物理接近性,还取决于DNA端切除17的延伸,它抑制了NHEJ。
同源性依赖的DSB修复启动核解降解的5’链从断裂端产生3’单链DNA(ssDNA)尾部,这个过程被称为5′-3’切除。MRN复合物启动DNA端切除术,并结合BLM/EXO1(布洛姆综合征蛋白/外核酶1)或BLM/DNA2(DNA复制ATP依赖性直升机酸酶,/核酸酶)18、19、20、21、22,19,20进行进一,步切除。21CtIP(CtBP相互作用蛋白)通过与 MRN 复合物23的直接相互作用和 BRCA1(乳腺癌 1 型易感性蛋白)24,25的招募来增强 DNA 末切。复制蛋白A(RPA)迅速结合到暴露的ssDNA,然后被重组酶蛋白RAD51取代,形成核蛋白丝,催化同源搜索和链入侵26,27,28。26,27,28
切除的启动是修复路径选择的关键步骤。一旦切除开始,DNA末端成为由Ku70/Ku80异质剂(NHEJ通路的组成部分)结合的不良基质,细胞被承诺到HR 17,29,30。17,29,30Ku70/Ku80异质剂结合到DSB末端,招募DNA-PKcs和p53结合蛋白,1(53BP1)29,2930。53BP1在G1中起到切除屏障的作用,从而在推广NHEJ31、32,32的同时阻断HR,但在S相中以BRCA1依赖的方式去除,从而允许切除发生33,34,34。因此,53BP1 和 BRCA1 在 DSB 维修中扮演相反的角色,53BP1 是 NHEJ 促进者,而 BRCA1 则通过 HR 进行中断修复。
在实验室中,DSB形成可由电离辐射(IR)引起。虽然本示例利用高剂量的 4 Gy、1 Gy 和 2 Gy 也创建大量的 DSB,适合通过丰富的蛋白质分析叶层形成。需要注意的是,使用的辐射类型和剂量可导致DNA和细胞中不同的病变:虽然IR诱导DSB,它也可能导致单股断裂或碱基修饰(见35,36,,36关于辐照线性能量转移(LET)和DNA损伤类型的参考)。为了确定电离辐射诱导的源(IRIF)形成的动力学及其间隙,表明修复激活的DDR8、9、37、38,9,37,的损伤和反转,在电离辐射后的不同时间点可以监测源层。所有主要DNA损伤蛋白的激活和清除时间已知为39种,许多被调查为关键事件的代理标记。例如,对 ssDNA 具有高亲和力的 pRPA 用作断分切除的代理,MRN 蛋白(MRE11、RAD50、NBS1)和外核酶也可用于评估切除效率。虽然RAD51、BRCA1、BRCA2(乳腺癌2型易感性蛋白)和PALB2(BRCA2的合作伙伴和本地化者)可以监测,以评估人力资源效率,而Ku蛋白或53BP1的存在则用作NHEJ的标记(图1)。
当DNA修复机械的蛋白质相互招募到超级复合物中分离和组装时,DNA蛋白和蛋白质-蛋白质的相互作用可以通过跟踪它们各自的定位并分析蛋白质的共同定位来推断,如细胞40、41、42,41,42中的重叠信号。在细胞系中,通过基因组编辑或对质粒突变体过度表达,在特定DNA修复基因中引入点突变或删除,允许调查特定残留物及其在识别DNA损伤(例如,与+H2AX)或复杂组装(与其他蛋白质或几种蛋白质共同定位)中可能发挥的作用,以及它们对DNA修复的影响。在这里,我们使用间接免疫荧光作为一种均值,通过跟踪 +2AX foci 来研究 DSB 的形成和分辨率。我们还提出了一个示例,由 DSB 修复的主要参与者进行 foci 形成和共同定位分析:p53 结合蛋白 1 (53BP1)32。如前所述,53BP1 被认为是 DNA 修复路径选择的核心。在 53BP1 积累及其与 +2AX 的共定位后,提供了有关细胞周期相、DNA损伤积累和用于修复 DSB 的路径的宝贵信息。间接免疫定位的目的是评估细胞系DNA损伤修复的效率,如本研究中的IR之后,或在接触细胞中的各种应力后,从DNA交联到复制分叉的阻塞(表1提供了DNA损伤剂列表)。
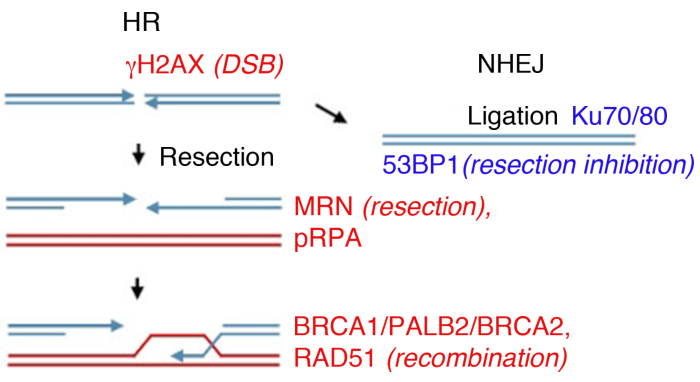
图1:DNA双链断裂(DSB)修复途径。
DSB 修复涉及两个主要途径:同源重组(HR、左)和非同源端连接(NHEJ,右)。断裂后,蛋白质被激活以标记断裂(+2AX),参与最终切除(MRN),涂上被切除的ssDNA(pRPA),促进重组(BRCA1,PALB2,BRCA2,RAD51)或限制切除,促进NHEJ(53BP1)。其他蛋白质参与损伤修复,但列出的蛋白质通常遵循间接免疫荧光。 请单击此处查看此图的较大版本。
| DNA损伤剂 | 行动机制 | 推荐剂量 |
| • 射线/X 射线 | 辐射 形成双链断裂与一些不受控制的细胞效应 |
1-4 Gy |
| 36Ar 离子 | 辐射 双链断裂的形成 |
270千瓦/μm |
| • 粒子 | 辐射 双链断裂的形成 |
116 keV/μm |
| 博莱 霉 素 | DNA合成的抑制剂 | 0.4-2 μg/mL |
| 喜 树 碱 | 托托莫默酶 I 的抑制剂 | 10-200 nM |
| 顺 铂 | 阿尔基拉特剂 (诱导海峡内部交联) |
0.25-2 μM |
| 阿 霉 素 | 中间代理 托托莫默酶II.的抑制剂 |
10-200 nM |
| 埃托波赛德 | 托托莫默酶II.的抑制剂 | 10 μM |
| 羟基尤雷亚 | DNA合成的抑制剂 (由核糖核酸还原酶) |
10-200 μM |
| 甲基甲烷硫化甲酸酯 | 阿尔基拉特剂 | 0.25-2 mM |
| 米托米霉素 C | 阿尔基拉特剂 | 0.25-2 μM |
| 紫外线(UV)光 | 甲状腺素二丁的形成 (产生DNA链失真) |
50-100 mJ/厘米2 |
表1:基因毒性剂.DNA损伤剂的例子,其作用机制和根据建议的工作浓度引起的损伤。
Protocol
Representative Results
Discussion
通过显微镜分析DNA损伤修复的时机和效率,已证明对了解DNA修复机械在正常细胞和人类疾病(如癌症)中如何工作至关重要。
特异性抗体的培养,允许检测活性蛋白在其磷酸化版本(如+H2AX,pRPA,pRAD50和其他10,23,39,43),39,43在更好地了解DNA修复时间的事件及其与细胞周期的同步。10<su…
Declarações
The authors have nothing to disclose.
Acknowledgements
这项工作得到了圣安东尼奥地区基金会的赠款的支持。Mays 癌症中心由 NCI 癌症中心支持核心赠款 P30 CA054174 提供支持。我们要感谢斯蒂芬·霍洛韦帮助采购试剂,感谢宋实验室提供空间和显微镜容量。
Materials
| 16% (v/v) paraformaldehyde (PFA) aqueous solution | Electron Microscopy Sciences | 15710 | Microscopy quality of the PFA ensures best images. If using "home-made PFA", filter prior to use. |
| Bovine serum albumin (BSA) | Sigma-Aldrich | A3059 | Heat-shock fraction is recommended, to avoid precipitation/background. |
| Coverglass #1, 18 mm round (coverslips) | Neuvitro | NC0308920 | Coverslips need to be cleaned and sterilized prior using, with HCl or ethanol. |
| DMEM, High Glucose [(+) 4.5 g/L D-Glucose, (+) L-Glutamine, (-) Sodium Pyruvate] | Gibco | 11965118 | Adjust the growing media to the needs of cell line used. |
| DPBS, no calcium, no magnesium | Gibco | 14190144 | PBS for tissue culture. |
| Ethylene glycol-bis(β-aminoethyl ether)-N,N,N′,N′-tetraacetic acid (EGTA) | Research Products International | E57060 | Nuclear extraction buffer. |
| Fetal Bovine Serum (FBS) | Life Technologies | 104370028 | The quality of FBS can be assessed by testing gH2AX foci formation. If traces of genotoxic endotoxin are present in the batch, gH2AX will be positive in the absence of stress. |
| Magnesium chloride (MgCl2) | Research Products International | M24000 | Nuclear extraction buffer. |
| Piperazine-N,N′-bis(2-ethanesulfonic acid) (PIPES) | Research Products International | P40150 | Nuclear extraction buffer. |
| SlowFade Diamond Antifade Mountant with DAPI | Invitrogen | S36973 | 300 nM DAPI with VECTASHIELD Antifade Mounting Medium can be used instead. |
| Sodium chloride (NaCl) | Research Products International | S23020 | Nuclear extraction buffer. |
| Sucrose | Research Products International | S24060 | Nuclear extraction buffer. |
| Superfrost Plus Microscope Slides | Fisherbrand | 1255015 | Polysine Slides can be used instead. |
| TC-Treated Multiple Well Plates, size 12 wells | Costar | 3513 | Seeding on coverslips is done in 12-wells plate. |
| Triton X-100 | AmericanBio | AB02025 | Nuclear extraction buffer. |
| TrypLE Express Enzyme (1X), No Phenol Red | Gibco | 12604021 | Trypsin-EDTA can be used instead. |
| Trypsin-EDTA (0.5%), No Phenol Red | Gibco | 15400054 | TrypLE can be used instead. |
Referências
- Prakash, R., Zhang, Y., Feng, W., Jasin, M. Homologous recombination and human health: the roles of BRCA1, BRCA2, and associated proteins. Cold Spring Harbor Perspectives in Biology. 7 (4), 016600 (2015).
- Jalan, M., Olsen, K. S., Powell, S. N. Emerging Roles of RAD52 in Genome Maintenance. Cancers (Basel). 11 (7), (2019).
- Oh, J., Symington, L. S. Role of the Mre11 Complex in Preserving Genome Integrity. Genes (Basel). 9 (12), (2018).
- Uziel, T., et al. Requirement of the MRN complex for ATM activation by DNA damage. The EMBO Journal. 22 (20), 5612-5621 (2003).
- Lee, J. H., Paull, T. T. ATM activation by DNA double-strand breaks through the Mre11-Rad50-Nbs1 complex. Science. 308 (5721), 551-554 (2005).
- Rogakou, E. P., Pilch, D. R., Orr, A. H., Ivanova, V. S., Bonner, W. M. DNA double-stranded breaks induce histone H2AX phosphorylation on serine 139. Journal of Biological Chemistry. 273, 5858-5868 (1998).
- Kinner, A., Wu, W., Staudt, C., Iliakis, G. γ-H2AX in recognition and signaling of DNA double-strand breaks in the context of chromatin. Nucleic Acids Research. 36 (17), 5678-5694 (2008).
- Martin, O. A., Pilch, D. R., Redon, C., Bonner, W. M. Histone H2AX in DNA damage repair. Cancer Biology & Therapy. 2 (3), 233-235 (2003).
- Rogakou, E. P., Boon, C., Redon, C., Bonner, W. M. Megabase chromatin domains involved in DNA double-strand breaks in vivo. Journal of Cell Biology. 146 (5), 905-916 (1999).
- Stucki, M., et al. MDC1 directly binds phosphorylated histone H2AX to regulate cellular responses to DNA double-strand breaks. Cell. 123 (7), 1213-1226 (2005).
- Lou, Z., et al. MDC1 maintains genomic stability by participating in the amplification of ATM-dependent DNA damage signals. Molecular Cell. 21 (2), 187-200 (2006).
- Chapman, J. R., Jackson, S. P. Phospho-dependent interaction between NBS1 and MDC1 mediate chromatin retention of the MRN complex at sites of DNA damage. EMBO Reports. 9 (8), 795-801 (2008).
- Melander, F., et al. Phosphorylation of SDT repeats in the MDC1 N terminus triggers retention of NBS1 at the DNA damage-modified chromatin. Journal of Cell Biology. 181 (2), 213-226 (2008).
- Branzei, D., Foiani, M. Regulation of DNA repair throughout the cell cycle. Nature Review. Molecular Cell Biology. 9 (4), 297-308 (2008).
- Chiruvella, K. K., Liang, Z., Wilson, T. E. Repair of double-strand breaks by end joining. Cold Spring Harbor Perspectives in Biology. 5 (5), 012757 (2013).
- Mehta, A., Haber, J. E. Sources of DNA double-strand breaks and models of recombinational DNA repair. Cold Spring Harbor Perspectives in Biology. 6 (9), 016428 (2014).
- Symington, L. S., Gautier, J. Double-strand break end resection and repair pathway choice. Annual Review of Genetics. 45, 247-271 (2011).
- Huertas, P. DNA resection in eukaryotes: deciding how to fix the break. Nature Structural & Molecular Biology. 17 (1), 11-16 (2010).
- Nimonkar, A. V., et al. BLM-DNA2-RPA-MRN and EXO1-BLM-RPA-MRN constitute two DNA end resection machineries for human DNA break repair. Genes & Development. 25 (4), 350-362 (2011).
- Garcia, V., Phelps, S. E. L., Gray, S., Neale, M. J. Bidirectional resection of DNA double-strand breaks by Mre11 and Exo1. Nature. 479 (7372), 241-244 (2011).
- Sturzenegger, A., et al. DNA2 cooperates with the WRN and BLM RecQ helicases to mediate long-range DNA end resection in human cells. Journal of Biological Chemistry. 289 (39), 27314-27326 (2014).
- Daley, J. M., Niu, H., Miller, A. S., Sung, P. Biochemical mechanism of DSB end resection and its regulation. DNA Repair. 32, 66-74 (2015).
- Sartori, A. A., et al. Human CtIP promotes DNA end resection. Nature. 450 (7169), 509-514 (2007).
- Chen, L., Nievera, C. J., Lee, A. Y. L., Wu, X. Cell cycle-dependent complex formation of BRCA1-CtIP-MRN is important for DNA double-strand break repair. Journal of Biological Chemistry. 283, 7713-7720 (2008).
- Yun, M. H., Hiom, K. CtIP-BRCA1 modulates the choice of DNA double-strand break repair pathway throughout the cell cycle. Nature. 459 (7245), 460-463 (2009).
- Sung, P., Klein, H. Mechanism of homologous recombination: mediators and helicases take on regulatory functions. Nature Review. Molecular Cell Biology. 7, 739-750 (2006).
- San Filippo, J., Sung, P., Klein, H. Mechanisms of eukaryotic homologous recombination. Annual Review of Biochemistry. 77, 229-257 (2008).
- Jasin, M., Rothstein, R. Repair of strand breaks by homologous recombination. Cold Spring Harbor Perspectives in Biology. 5 (11), 012740 (2013).
- Dynan, W. S., Yoo, S. Interaction of Ku protein and DNA-dependent protein kinase catalytic subunit with nucleic acids. Nucleic Acids Research. 26 (7), 1551-1559 (1998).
- Lieber, M. R. The mechanism of double-strand DNA break repair by the nonhomologous DNA end-joining pathway. Annual Review of Biochemistry. 79, 181-211 (2010).
- Cejka, P. DNA end resection: nucleases team up with the right partners to initiate homologous recombination. Journal of Biological Chemistry. 290 (38), 22931-22938 (2015).
- Mirman, Z., de Lange, T. 53BP1: a DSB escort. Genes & Development. 34, 7-23 (2020).
- Cao, L., et al. A selective requirement for 53BP1 in the biological response to genomic instability induces by BRCA1 deficiency. Molecular Cell. 35 (4), 534-541 (2009).
- Zimmermann, M., de Lange, T. 53BP1: Pro choice in DNA repair. Trends in Cell Biology. 24 (2), 108-117 (2014).
- Mavragani, I. V., Nikitaki, Z., Kalospyros, S. A., Georgakilas, A. G. Ionizing Radiation and Complex DNA Damage: From Prediction to Detection Challenges and Biological Significance. Cancers (Basel). 11 (11), (2019).
- Nikitaki, Z., et al. Measurement of complex DNA damage induction and repair in human cellular systems after exposure to ionizing radiations of varying linear energy transfer (LET). Free Radical Research. 50, 64-78 (2016).
- Redon, C., et al. Histone H2A variants H2AX and H2AZ. Current Opinion in Genetics & Development. 12 (2), 162-169 (2002).
- Fernandez-Capetillo, O., Lee, A., Nussenzweig, M., Nussenzweig, A. H2AX: the histone guardian of the genome. DNA Repair. 3 (8-9), 959-967 (2004).
- Paull, T. T., et al. A critical role for histone H2AX in recruitment of repair factors to nuclear foci after DNA damage. Current Biology. 10 (15), 886-895 (2000).
- Sy, S. M. H., Huen, M. S. Y., Chen, J. PALB2 is an integral component of the BRCA complex required for homologous recombination repair. Proceedings of the National Academy of Sciences. 106 (17), 7155-7160 (2009).
- Buisson, R., Masson, J. Y. PALB2 self-interaction controls homologous recombination. Nucleic Acids Research. 40 (20), 10312-10323 (2012).
- Belotserkovskaya, R., et al. PALB2 chromatin recruitment restores homologous recombination in BRCA1-deficient cells depleted of 53BP1. Nature Communications. 11 (1), 819 (2020).
- Betts, J. A., et al. Long noncoding RNAs CUPID1 and CUPID2 mediate breast cancer risk at 11q13 by modulating the response to DNA damage. American Journal of Human Genetics. 101 (2), 255-266 (2017).
- Dray, E., et al. Molecular basis for enhancement of the meiotic DMC1 recombinase by RAD51 associated protein 1 (RAD51AP1). Proceedings of the National Academy of Sciences. 108 (9), 3560-3565 (2011).

