Visualisering af DNA Reparation Proteiner Interaktion ved Immunofluorescence
Summary
Efter DNA-skader aktiverer humane celler vigtige reparationsveje for at genoprette integriteten af deres genom. Her beskriver vi metoden til indirekte immunfluorescens som et middel til at detektere DNA-reparationsproteiner, analysere deres rumlige og tidsmæssige rekruttering og hjælpe med at afhøre proteinproteininteraktion på steder med DNA-skader.
Abstract
Pattedyrceller er konstant udsat for kemikalier, stråling, og naturligt forekommende metaboliske biprodukter, som skaber specifikke typer af DNA-fornærmelser. Genotoksiske midler kan beskadige DNA-rygraden, bryde den eller ændre den kemiske karakter af individuelle baser. Efter DNA-fornærmelse aktiveres DNA-skadesresponsveje (DDR), og proteiner, der er involveret i reparationen, rekrutteres. En overflod af faktorer er involveret i sensing typen af skader og aktivere passende reparation svar. Undladelse af korrekt aktivere og rekruttere DDR faktorer kan føre til genomisk ustabilitet, som ligger til grund for mange menneskelige patologier, herunder kræft. Undersøgelser af DDR-proteiner kan give indsigt i genotoksisk lægemiddelrespons og cellulære mekanismer for lægemiddelresistens.
Der er to vigtige måder at visualisere proteiner in vivo:direkte observation, ved at mærke protein af interesse med et fluorescerende protein og efter det ved levende billeddannelse, eller indirekte immunfluorescens på faste prøver. Mens visualisering af fluorescerende mærkede proteiner giver mulighed for præcis overvågning over tid, direkte mærkning i N- eller C-endestation kan forstyrre protein lokalisering eller funktion. Observation af proteiner i deres uændrede, endogene version foretrækkes. Når DNA reparation proteiner rekrutteres til DNA-fornærmelse, deres koncentration stiger lokalt, og de danner grupper, eller “foci”, der kan visualiseres ved indirekte immunfluorescens ved hjælp af specifikke antistoffer.
Selv om påvisning af protein foci ikke giver et endeligt bevis for direkte interaktion, co-lokalisering af proteiner i celler viser, at de omgruppere til stedet for skader og kan informere om rækkefølgen af begivenheder, der kræves for kompleks dannelse. Omhyggelig analyse af foci rumlige overlapning i celler, der udtrykker vilde type eller mutant versioner af et protein kan give dyrebare spor på funktionelle domæner vigtigt for DNA reparation funktion. Sidst, co-lokalisering af proteiner indikerer mulige direkte interaktioner, der kan verificeres ved co-immunoprecipation i celler, eller direkte pulldown ved hjælp af rensede proteiner.
Introduction
Menneskelige celler er konstant udsat for en række DNA skadelige stoffer af forskellig oprindelse. Eksogene kilder består for det meste af eksponering for stråling, kemikalier (herunder kemoterapeutiske midler og visse antibiotika) og virus, mens de vigtigste endogene kilder omfatter fejl i DNA-replikation og oxidativt stress. De direkte virkninger af genotoksisk eksponering kan variere fra en modificeret base til en potentielt dødelig DNA dobbelt-streng pause (DSB), afhængigt af stress og eksponeringsdosis. I sidste ende, urepareret eller fejlrepareret DNA-skader kan føre til ophobning af mutationer, genomiske rearrangements, genom ustabilitet og i sidste ende føre til carcinogenese1. Pattedyrceller har udviklet komplekse veje til at genkende specifikke typer afDNA-skader 2,,3 og reparere dem i tide, synkroniseret med cellecyklus progression.
Iioniserende stråling (IR) skader DNA dobbelt helix og skaber dobbelt-streng pauser (DSBs), en af de mest skadelige former for DNA-skader. MRN (MRE11, RAD50, NBS1) kompleks fungerer som en sensor af DNA ender og aktiverer protein kinase ataxia telangiectasia muteret (ATM)4,5. Efter den første aktivering af ATM ved DNA ender, ATM udløser en kaskade af DDR begivenheder på det sted, hvor pausen, indlede med en vigtig begivenhed, fosforylering af histone variant H2AX6. H2AX-fosforylering på restprodukt S139 aktiverer det i γH2AX, der spænder over områder op til megabaser omkring DNA-læsionen6,7,8,9. Denne hændelse øger DNA-tilgængeligheden, hvilket fører til rekruttering og akkumulering af andre DNA-reparationsproteiner7. Fordi γH2AX er rigeligt og specifikt induceret omkringliggende DSBs, det kan let visualiseres ved hjælp af specifikke antistoffer, og er almindeligt anvendt som en surrogat markør for DSBs i DNA reparation området. Når bruddet er signaleret, celler aktivere deres DNA reparation veje og behandle DNA-skader. Proteinet MDC1 (mægler af DNA-skader checkpoint protein 1) binder direkte γH2AX10, interagerer med ATM11 og også med NBS112,13. Det bidrager til at øge koncentrationen af MRN-komplekset i DSB og indleder et positivt atm-feedback-loop. γH2AX fjernes hurtigt, når bruddet er repareret, hvilket gør det muligt at overvåge DSB-frigangen. Efterfulgt af mikroskopi giver faldet i γH2AX over tid en indirekte måling af resterende brud og DNA-reparationseffektivitet.
Eukaryote celler kan reparere DSBs ved flere veje, de to vigtigste er ikke-homologe end-sammenføjning (NHEJ) og homolog rekombination (HR) (Figur 1). NHEJ i det væsentlige ligaerer DNA dobbelt-streng ender uden brug af udvidet homologi og opererer i hele cellecyklus 14,15. HR bliver fremherskende i S- og G2-faser, og undertrykkes ellers, da det kræver en søsterkromet som en homolog skabelon til reparation14,16. Pathway valg mellem NHEJ og HR ikke kun afhænger af den fysiske nærhed af søsterchromatid, men også af udvidelsen af DNA ende resektion17, som hæmmer NHEJ.
Homologi-afhængige DSB reparation initiere ved nukleolytisk nedbrydning af 5 ‘streng fra pausen ender til at generere 3 ‘single-streng DNA (ssDNA) haler, en proces benævnt 5’-3 ‘resektion. MRN-komplekset indleder DNA-slutresektion, og yderligere resektion behandles i kombination med BLM/EXO1 (Bloom syndrom protein/exonuclease 1) eller BLM/DNA2 (DNA replikation ATP-afhængig helicase/nuklease)18,19,20,21,22. DNA-slutresektion forstærkes af CtIP (CtBP-interagerende protein) gennem dets direkte interaktion med MRN-kompleks23 og rekruttering af BRCA1 (brystkræfttype 1 modtagelighedsprotein)24,25. Replikering protein A (RPA) straks binder sig til ssDNA eksponeret og derefter fordrevet af recombinase protein RAD51 til at danne en nukleoprotein glødetråd, der katalyserer homolog søgning og strand invasion26,27,28.
Indledningen af resektion er et kritisk skridt for reparation pathway valg. Når resektion er indledt, DNA ender bliver fattige substrater til binding af Ku70/Ku80 heterodimer (komponent af NHEJ vej) og celler er forpligtet til HR17,,29,30. Den Ku70/Ku80 heterodimer binder sig til DSB ender, rekruttere DNA-pc’er og p53 Bindende Protein 1 (53BP1)29,30. 53BP1 fungerer som en hindring for resektion i G1 og blokerer således HR og fremmer samtidig NHEJ31,32, men fjernes på en BRCA1-afhængig måde i S-fasen , hvilket gør det muligt at genskrive33,34. Derfor spiller 53BP1 og BRCA1 modsatrettede roller i DSB-reparationen, hvor 53BP1 er NHEJ-facilitator, mens BRCA1 handler, der gør det muligt at reparere gennem HR.
I laboratoriet kan DSB-formationen induceres ved iioniseringsstråling (IR). Mens dette eksempel udnytter en høj dosis på 4 Gy, 1 Gy og 2 Gy også skabe en betydelig mængde af DSBs, egnet til analyse af foci dannelse af rigelige proteiner. Det er vigtigt at bemærke, at den anvendte type og dosis af stråling kan føre til forskellige læsioner i DNA og i cellen: mens IR inducerer DSBs, kan det også forårsage enkelt streng pauser eller base modifikation (se35,36 for en reference på bestråling lineær energioverførsel (LET) og type DNA-skader). For at bestemme kinetik af iionizing stråling-induceret foci (IRIF) dannelse og deres clearance, som indikerer reparation af skader og tilbageførsel af den aktiverede DDR8,9,37,38, foci dannelse kan overvåges på forskellige tidspunkter efter iionizing stråling.9 Timing af aktivering og clearance af alle større DNA-skader proteiner er kendt39, og mange er undersøgt som surrogat markører for vigtige begivenheder. For eksempel, pRPA, som har høj affinitet for ssDNA bruges som et surrogat for pause resektion, MRN proteiner (MRE11, RAD50, NBS1) og exonucleaser kan bruges til at vurdere resektion effektivitet også. Mens RAD51, BRCA1, BRCA2 (brystkræft type 2 modtagelighed protein), og PALB2 (partner og localizer af BRCA2) kan overvåges for at evaluere HR effektivitet, tilstedeværelsen af Ku proteiner eller 53BP1, anvendes som markører for NHEJ (Figur 1).
Som proteiner af DNA reparation maskiner rekruttere hinanden til pause og samle i super-komplekser, DNA-protein og protein-protein interaktioner kan udledes ved at følge deres individuelle lokalisering over tid og analysere co-lokalisering af proteiner, som visualiseret af overlappende signaler i celle40,41,42. I cellelinjer giver indførelsen af punktmutationer eller sletning i specifikke DNA-reparationsgener enten gennem genomredigering eller ved overekspression af plasmidbaserede mutanter mulighed for undersøgelse af specifikke restkoncentrationer og deres mulige rolle i genkendelse af DNA-skader (f.eks. co-lokalisering med γH2AX) eller kompleks samling (sam lokalisering med en anden eller flere proteiner) samt deres indvirkning på DNA-reparationen. Her bruger vi indirekte immunfluorescens som et middel til at undersøge dannelsen og opløsningen af DSB’er ved at følge γH2AX foci over tid. Vi præsenterer også et eksempel på foci formation og co-lokalisering analyse af en stor aktør i DSB reparation: p53 Bindende Protein 1 (53BP1)32. Som tidligere nævnt, 53BP1 anses for centralt for DNA reparation pathway valg. Efter 53BP1 ophobning og dens co-lokalisering med γH2AX giver værdifulde oplysninger om celle cyklus fase, DNA-skader ophobning, og vej bruges til at reparere DSBs. Formålet med indirekte immunolokalkisering er at vurdere effektiviteten af DNA-skade reparation i cellelinjer, efter IR som i denne undersøgelse, eller efter eksponering for forskellige belastninger i celle, fra DNA crosslinking til blokering af replikation gaffel (en liste over DNA skadelige stoffer er fastsat i tabel 1).
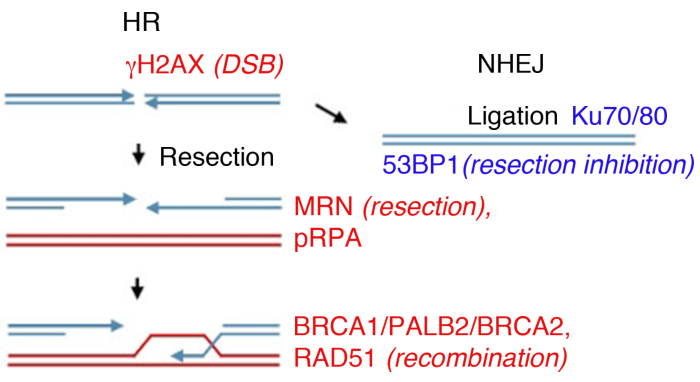
Figur 1: DNA dobbelt streng pauser (DSB) reparation veje.
DSB reparation indebærer to store veje: Homolog rekombination (HR, venstre) og ikke-homologe End-Joining (NHEJ, højre). Efter pausen aktiveres proteiner for at markere bruddet (γH2AX), deltager i slutresektionen (MRN), belægger den resected ssDNA (pRPA), fremmer rekombination (BRCA1, PALB2, BRCA2, RAD51) eller begrænser resektion og fremmer NHEJ (53BP1). Andre proteiner deltager i skadesreparation, men de anførte proteiner efterfølges rutinemæssigt af indirekte immunfluorescens. Klik her for at se en større version af dette tal.
| DNA-skademiddel | Virkningsmekanisme | Anbefalet dosis |
| γ-stråler/røntgenstråler | Stråling Dannelse af dobbeltstrengede brud med nogle ukontrollerede cellulære effekter |
1-4 Gy |
| kr. Ar ioner | Stråling Dannelse af dobbeltstrengede pauser |
270 keV/μm |
| α-partikler | Stråling Dannelse af dobbeltstrengede pauser |
116 keV/μm |
| Bleomycin | Hæmmer af DNA-syntese | 0,4-2 μg/ml |
| Camptothecin | Hæmmer af topoisomerase I | 10-200 nM |
| Cisplatin | Alkylerende middel (inducerende intrastrand krydslinks) |
0,25-2 μM |
| Doxorubicin | Intercaleringsmiddel Hæmmer af topoisomerase II |
10-200 nM |
| Etoposide | Hæmmer af topoisomerase II | 10 μM |
| Hydroxyurea | Hæmmer af DNA-syntese (ved ribonucleotid reduktase) |
10-200 μM |
| Methylmethanulfonat | Alkylerende middel | 0,25-2 mM |
| Mitomycin C | Alkylerende middel | 0,25-2 μM |
| Ultraviolet (UV) lys | Dannelse af thymidindæmpere (genererer forvrængning af DNA-kæden) |
50-100 mJ/cm2 |
Tabel 1: Genotoksiske midler. Eksempler på DNA-skadelige stoffer, deres virkningsmekanisme og de skader, der er forårsaget på grundlag af foreslået arbejdskoncentration.
Protocol
Representative Results
Discussion
Analyse af timingen og effektiviteten af DNA-skader reparation ved mikroskopi har vist sig afgørende for at forstå, hvordan DNA reparation maskiner funktioner, i normale celler og i menneskelige patologier såsom kræft.
Udviklingen af specifikke antistoffer, der gør det muligt at detektionere aktiverede proteiner i deres fosforylerede version (såsom γH2AX, pRPA, pRAD50 og andre10,,23,39,</s…
Declarações
The authors have nothing to disclose.
Acknowledgements
Dette arbejde blev støttet af et tilskud fra San Antonio Area Foundation. Mays Cancer Center er støttet af NCI kræft center støtte kerne tilskud P30 CA054174. Vi vil gerne takke Stephen Holloway for hans hjælp sourcing reagenser, og Sung laboratorium for at give plads og mikroskopi kapacitet.
Materials
| 16% (v/v) paraformaldehyde (PFA) aqueous solution | Electron Microscopy Sciences | 15710 | Microscopy quality of the PFA ensures best images. If using "home-made PFA", filter prior to use. |
| Bovine serum albumin (BSA) | Sigma-Aldrich | A3059 | Heat-shock fraction is recommended, to avoid precipitation/background. |
| Coverglass #1, 18 mm round (coverslips) | Neuvitro | NC0308920 | Coverslips need to be cleaned and sterilized prior using, with HCl or ethanol. |
| DMEM, High Glucose [(+) 4.5 g/L D-Glucose, (+) L-Glutamine, (-) Sodium Pyruvate] | Gibco | 11965118 | Adjust the growing media to the needs of cell line used. |
| DPBS, no calcium, no magnesium | Gibco | 14190144 | PBS for tissue culture. |
| Ethylene glycol-bis(β-aminoethyl ether)-N,N,N′,N′-tetraacetic acid (EGTA) | Research Products International | E57060 | Nuclear extraction buffer. |
| Fetal Bovine Serum (FBS) | Life Technologies | 104370028 | The quality of FBS can be assessed by testing gH2AX foci formation. If traces of genotoxic endotoxin are present in the batch, gH2AX will be positive in the absence of stress. |
| Magnesium chloride (MgCl2) | Research Products International | M24000 | Nuclear extraction buffer. |
| Piperazine-N,N′-bis(2-ethanesulfonic acid) (PIPES) | Research Products International | P40150 | Nuclear extraction buffer. |
| SlowFade Diamond Antifade Mountant with DAPI | Invitrogen | S36973 | 300 nM DAPI with VECTASHIELD Antifade Mounting Medium can be used instead. |
| Sodium chloride (NaCl) | Research Products International | S23020 | Nuclear extraction buffer. |
| Sucrose | Research Products International | S24060 | Nuclear extraction buffer. |
| Superfrost Plus Microscope Slides | Fisherbrand | 1255015 | Polysine Slides can be used instead. |
| TC-Treated Multiple Well Plates, size 12 wells | Costar | 3513 | Seeding on coverslips is done in 12-wells plate. |
| Triton X-100 | AmericanBio | AB02025 | Nuclear extraction buffer. |
| TrypLE Express Enzyme (1X), No Phenol Red | Gibco | 12604021 | Trypsin-EDTA can be used instead. |
| Trypsin-EDTA (0.5%), No Phenol Red | Gibco | 15400054 | TrypLE can be used instead. |
Referências
- Prakash, R., Zhang, Y., Feng, W., Jasin, M. Homologous recombination and human health: the roles of BRCA1, BRCA2, and associated proteins. Cold Spring Harbor Perspectives in Biology. 7 (4), 016600 (2015).
- Jalan, M., Olsen, K. S., Powell, S. N. Emerging Roles of RAD52 in Genome Maintenance. Cancers (Basel). 11 (7), (2019).
- Oh, J., Symington, L. S. Role of the Mre11 Complex in Preserving Genome Integrity. Genes (Basel). 9 (12), (2018).
- Uziel, T., et al. Requirement of the MRN complex for ATM activation by DNA damage. The EMBO Journal. 22 (20), 5612-5621 (2003).
- Lee, J. H., Paull, T. T. ATM activation by DNA double-strand breaks through the Mre11-Rad50-Nbs1 complex. Science. 308 (5721), 551-554 (2005).
- Rogakou, E. P., Pilch, D. R., Orr, A. H., Ivanova, V. S., Bonner, W. M. DNA double-stranded breaks induce histone H2AX phosphorylation on serine 139. Journal of Biological Chemistry. 273, 5858-5868 (1998).
- Kinner, A., Wu, W., Staudt, C., Iliakis, G. γ-H2AX in recognition and signaling of DNA double-strand breaks in the context of chromatin. Nucleic Acids Research. 36 (17), 5678-5694 (2008).
- Martin, O. A., Pilch, D. R., Redon, C., Bonner, W. M. Histone H2AX in DNA damage repair. Cancer Biology & Therapy. 2 (3), 233-235 (2003).
- Rogakou, E. P., Boon, C., Redon, C., Bonner, W. M. Megabase chromatin domains involved in DNA double-strand breaks in vivo. Journal of Cell Biology. 146 (5), 905-916 (1999).
- Stucki, M., et al. MDC1 directly binds phosphorylated histone H2AX to regulate cellular responses to DNA double-strand breaks. Cell. 123 (7), 1213-1226 (2005).
- Lou, Z., et al. MDC1 maintains genomic stability by participating in the amplification of ATM-dependent DNA damage signals. Molecular Cell. 21 (2), 187-200 (2006).
- Chapman, J. R., Jackson, S. P. Phospho-dependent interaction between NBS1 and MDC1 mediate chromatin retention of the MRN complex at sites of DNA damage. EMBO Reports. 9 (8), 795-801 (2008).
- Melander, F., et al. Phosphorylation of SDT repeats in the MDC1 N terminus triggers retention of NBS1 at the DNA damage-modified chromatin. Journal of Cell Biology. 181 (2), 213-226 (2008).
- Branzei, D., Foiani, M. Regulation of DNA repair throughout the cell cycle. Nature Review. Molecular Cell Biology. 9 (4), 297-308 (2008).
- Chiruvella, K. K., Liang, Z., Wilson, T. E. Repair of double-strand breaks by end joining. Cold Spring Harbor Perspectives in Biology. 5 (5), 012757 (2013).
- Mehta, A., Haber, J. E. Sources of DNA double-strand breaks and models of recombinational DNA repair. Cold Spring Harbor Perspectives in Biology. 6 (9), 016428 (2014).
- Symington, L. S., Gautier, J. Double-strand break end resection and repair pathway choice. Annual Review of Genetics. 45, 247-271 (2011).
- Huertas, P. DNA resection in eukaryotes: deciding how to fix the break. Nature Structural & Molecular Biology. 17 (1), 11-16 (2010).
- Nimonkar, A. V., et al. BLM-DNA2-RPA-MRN and EXO1-BLM-RPA-MRN constitute two DNA end resection machineries for human DNA break repair. Genes & Development. 25 (4), 350-362 (2011).
- Garcia, V., Phelps, S. E. L., Gray, S., Neale, M. J. Bidirectional resection of DNA double-strand breaks by Mre11 and Exo1. Nature. 479 (7372), 241-244 (2011).
- Sturzenegger, A., et al. DNA2 cooperates with the WRN and BLM RecQ helicases to mediate long-range DNA end resection in human cells. Journal of Biological Chemistry. 289 (39), 27314-27326 (2014).
- Daley, J. M., Niu, H., Miller, A. S., Sung, P. Biochemical mechanism of DSB end resection and its regulation. DNA Repair. 32, 66-74 (2015).
- Sartori, A. A., et al. Human CtIP promotes DNA end resection. Nature. 450 (7169), 509-514 (2007).
- Chen, L., Nievera, C. J., Lee, A. Y. L., Wu, X. Cell cycle-dependent complex formation of BRCA1-CtIP-MRN is important for DNA double-strand break repair. Journal of Biological Chemistry. 283, 7713-7720 (2008).
- Yun, M. H., Hiom, K. CtIP-BRCA1 modulates the choice of DNA double-strand break repair pathway throughout the cell cycle. Nature. 459 (7245), 460-463 (2009).
- Sung, P., Klein, H. Mechanism of homologous recombination: mediators and helicases take on regulatory functions. Nature Review. Molecular Cell Biology. 7, 739-750 (2006).
- San Filippo, J., Sung, P., Klein, H. Mechanisms of eukaryotic homologous recombination. Annual Review of Biochemistry. 77, 229-257 (2008).
- Jasin, M., Rothstein, R. Repair of strand breaks by homologous recombination. Cold Spring Harbor Perspectives in Biology. 5 (11), 012740 (2013).
- Dynan, W. S., Yoo, S. Interaction of Ku protein and DNA-dependent protein kinase catalytic subunit with nucleic acids. Nucleic Acids Research. 26 (7), 1551-1559 (1998).
- Lieber, M. R. The mechanism of double-strand DNA break repair by the nonhomologous DNA end-joining pathway. Annual Review of Biochemistry. 79, 181-211 (2010).
- Cejka, P. DNA end resection: nucleases team up with the right partners to initiate homologous recombination. Journal of Biological Chemistry. 290 (38), 22931-22938 (2015).
- Mirman, Z., de Lange, T. 53BP1: a DSB escort. Genes & Development. 34, 7-23 (2020).
- Cao, L., et al. A selective requirement for 53BP1 in the biological response to genomic instability induces by BRCA1 deficiency. Molecular Cell. 35 (4), 534-541 (2009).
- Zimmermann, M., de Lange, T. 53BP1: Pro choice in DNA repair. Trends in Cell Biology. 24 (2), 108-117 (2014).
- Mavragani, I. V., Nikitaki, Z., Kalospyros, S. A., Georgakilas, A. G. Ionizing Radiation and Complex DNA Damage: From Prediction to Detection Challenges and Biological Significance. Cancers (Basel). 11 (11), (2019).
- Nikitaki, Z., et al. Measurement of complex DNA damage induction and repair in human cellular systems after exposure to ionizing radiations of varying linear energy transfer (LET). Free Radical Research. 50, 64-78 (2016).
- Redon, C., et al. Histone H2A variants H2AX and H2AZ. Current Opinion in Genetics & Development. 12 (2), 162-169 (2002).
- Fernandez-Capetillo, O., Lee, A., Nussenzweig, M., Nussenzweig, A. H2AX: the histone guardian of the genome. DNA Repair. 3 (8-9), 959-967 (2004).
- Paull, T. T., et al. A critical role for histone H2AX in recruitment of repair factors to nuclear foci after DNA damage. Current Biology. 10 (15), 886-895 (2000).
- Sy, S. M. H., Huen, M. S. Y., Chen, J. PALB2 is an integral component of the BRCA complex required for homologous recombination repair. Proceedings of the National Academy of Sciences. 106 (17), 7155-7160 (2009).
- Buisson, R., Masson, J. Y. PALB2 self-interaction controls homologous recombination. Nucleic Acids Research. 40 (20), 10312-10323 (2012).
- Belotserkovskaya, R., et al. PALB2 chromatin recruitment restores homologous recombination in BRCA1-deficient cells depleted of 53BP1. Nature Communications. 11 (1), 819 (2020).
- Betts, J. A., et al. Long noncoding RNAs CUPID1 and CUPID2 mediate breast cancer risk at 11q13 by modulating the response to DNA damage. American Journal of Human Genetics. 101 (2), 255-266 (2017).
- Dray, E., et al. Molecular basis for enhancement of the meiotic DMC1 recombinase by RAD51 associated protein 1 (RAD51AP1). Proceedings of the National Academy of Sciences. 108 (9), 3560-3565 (2011).

