הדמיה של אינטראקציה חלבוני תיקון DNA על ידי אימונופלואורסץ
Summary
בעקבות נזק DNA, תאים אנושיים להפעיל מסלולי תיקון חיוניים כדי לשחזר את שלמות הגנום שלהם. כאן, אנו מתארים את השיטה של חיסון עקיף כאמצעי לזיהוי חלבוני תיקון DNA, לנתח את הגיוס המרחבי והזמני שלהם, ולעזור לחקור אינטראקציה חלבון-חלבון באתרים של נזק DNA.
Abstract
תאים יונקים נחשפים כל הזמן כימיקלים, קרינה, ומוצרי משנה מטבוליים המתרחשים באופן טבעי, אשר יוצרים סוגים ספציפיים של עלבונות DNA. סוכנים גנוטוקסיים יכולים לגרום נזק לעמוד השדרה של הדנ”א, לשבור אותו, או לשנות את האופי הכימי של בסיסים בודדים. בעקבות עלבון DNA, מסלולי תגובה נזק DNA (DDR) מופעלים חלבונים המעורבים בתיקון מגויסים. שפע של גורמים מעורבים בחישת סוג הנזק והפעלת תגובת התיקון המתאימה. כישלון בהפעלה נכונה וגיוס גורמי DDR יכול להוביל לחוסר יציבות גנומית, אשר בבסיס פתולוגיות אנושיות רבות כולל סרטן. מחקרים של חלבוני DDR יכולים לספק תובנות לתגובה לתרופות גנוטוקסיות ומנגנונים תאיים של עמידות לתרופות.
ישנן שתי דרכים עיקריות לדמיין חלבונים בvivio: תצפית ישירה, על ידי תיוג החלבון של עניין עם חלבון פלורסנט ואחריו על ידי הדמיה חיה, או חיסון עקיף על דגימות קבועות. בעוד הדמיה של חלבונים מתויגים פלורסנט מאפשר ניטור מדויק לאורך זמן, תיוג ישיר N- או C-terminus יכול להפריע למיקום מחדש של חלבון או פונקציה. התבוננות בחלבונים בגרסה אנדוגנית שלא שונה שלהם עדיפה. כאשר חלבוני תיקון DNA מגויסים עלבון ה-DNA, הריכוז שלהם גדל באופן מקומי והם יוצרים קבוצות, או “foci”, שניתן לדמיין על ידי אימונו-לודיזם עקיף באמצעות נוגדנים ספציפיים.
למרות זיהוי של חלבון foci אינו מספק הוכחה מוחלטת של אינטראקציה ישירה, התאמה חברתית של חלבונים בתאים מצביעה על כך שהם להתארגן מחדש לאתר של נזק יכול להודיע על רצף האירועים הנדרשים להיווצרות מורכבת. ניתוח זהיר של חפיפה מרחבית foci בתאים המביעים סוג בר או גרסאות מוטנט של חלבון יכול לספק רמזים יקרים על תחומים פונקציונליים חשובים עבור פונקציית תיקון DNA. לבסוף, התאמה ישירה של חלבונים מצביעה על אינטראקציות ישירות אפשריות שניתן לאמת על ידי שיתוף ערך חיסוני בתאים, או משיכה ישירה באמצעות חלבונים מטוהרים.
Introduction
תאים אנושיים נחשפים כל הזמן למגוון של סוכני DNA מזיקים ממקורות שונים. מקורות אקסוגניים מורכבים בעיקר מחשיפה לקרינה, כימיקלים (כולל חומרים כימותרפיים וכמה אנטיביוטיקה), ווירוסים, בעוד המקורות האנדורניים העיקריים כוללים שגיאות בשכפול DNA ומתח חמצוני. ההשפעות הישירות של חשיפה גנוטוקסית יכול לנוע מבסיס שונה לשבור DNA קטלני פוטנציאלית דו גדיל (DSB), בהתאם ללחץ ואת מינון החשיפה. בסופו של דבר, נזק DNA לא מתוקן או שלא תוקן כהלכה יכול להוביל להצטברות של מוטציות, סידורים מחדש גנומיים, חוסר יציבות הגנום ובסופו של דבר להוביל קרסינוגנזה1. תאים יונקים פיתחו מסלולים מורכבים כדי לזהות סוגים ספציפיים של נזק DNA2,,3 ולתקן אותם בזמן, מסונכרן עם התקדמות מחזור התא.
קרינה מיננת (IR) פוגעת בסליל ה-DNA הכפול ויוצרת הפסקות דו-גדיליות (DSBs), אחת הצורות המבזות ביותר של נזק לדנ”א. MRN (MRE11, RAD50, NBS1) פונקציות מורכבות כמו חיישן של ה-DNA מסתיים ומפעיל את החלבון קינאז ataxia telangiectasia מוטציה (ATM)4,,5. לאחר ההפעלה הראשונית של ATM על ידי קצוות DNA, ATM מפעיל מפל של אירועי DDR באתר ההפסקה, ייזום עם אירוע מפתח, phosphorylation של משתנה histone H2AX6. H2AX phosphorylation על שאריות S139 מפעיל אותו לתוך ơH2AX, משתרע על פני אזורים עד megabases סביב נגע ה-DNA6,7,,8,9. אירוע זה מגביר את הנגישות DNA, המוביל גיוס והצטברות של חלבוני תיקון DNA אחרים7. מכיוון 8H2AX הוא בשפע ובמיוחד מושרה סביב DSBs, זה יכול להיות חזותי בקלות באמצעות נוגדנים ספציפיים, והוא משמש בדרך כלל כסמן פונדקאית עבור DSBs בשדה תיקון ה-DNA. ברגע שההפסקה מאותתת, תאים מפעילים את נתיבי תיקון הדנ”א שלהם ומעבדים את נזקי הדנ”א. החלבון MDC1 (מגשר של חלבון מחסום נזק DNA 1) ישירות נקשר ơH2AX10, אינטראקציה עם ATM11 וגם עם NBS112,13. זה תורם להגדלת הריכוז של מתחם MRN ב DSB וייזום לולאת משוב חיובית כספומט. ơH2AX מוסר במהירות לאחר תיקון השבר, כתוצאה מכך, המאפשר ניטור של סיווג DSB. ואחריו מיקרוסקופיה, הירידה ב 0H2AX לאורך זמן מספק מדידה עקיפה של הפסקות שיורית ויעילות תיקון DNA.
תאים אאוקריוטיים יכולים לתקן DSBs על ידי מספר מסלולים, שני העיקריים להיות לא הומולוגי סוף הצטרפות (NHEJ) ו שילוב הומולוגי (HR)(איור 1). NHEJ למעשה רצועות DNA כפול גדיל מסתיים ללא שימוש הומולוגיה מורחבת ופועללאורך כל מחזור התא 14,15. משאבי אנוש הופכים לדומיננטיים בשלבי S ו-G2, ומודחקים באופן אחר, שכן היא דורשת כרומטיד אחות כתבנית הומולוגיתלתיקון 14,16. בחירת מסלול בין NHEJ ו HR לא רק תלוי הקרבה הפיזית של כרומט אחותו, אלא גם על הרחבת ה-DNAסוף חותך 17, אשר מעכב NHEJ.
תיקון DSB תלוי הומולוגיה יוזם על ידי השפלה נוקליאוליטית של 5 ‘ גדיל מן קצוות ההפסקה כדי ליצור 3 ‘ זנבות DNA גדיל יחיד (ssDNA), תהליך המכונה 5′-3’ ריתוק. תסביך MRN יוזם את החיתוך קצה DNA וכתור נוסף מעובד בשילוב עם BLM / EXO1 (תסמונת בלום חלבון / exonuclease 1) או BLM / DNA2 (שכפול DNA ATP תלוי הליקאז / nuclease)18, 19,,20,21,22.19 ניתוח קצה DNA משופרת על ידי CtIP (חלבון אינטראקציה CtBP) באמצעות האינטראקציה הישירה שלה עם MRNמורכב 23 וגיוס של BRCA1 (סרטן השד סוג 1 חלבון רגישות)24,25. חלבון שכפול A (RPA) נקשר מיד ssDNA חשוף ולאחר מכן נעקר על ידי חלבון רקומבינאז RAD51 כדי ליצור חוט נוקליאופרוטאין כי מזרז חיפוש הומולוגיופלישה גדיל 26,27,28.
החניכה של ניתוח הוא צעד קריטי לבחירה במסלול תיקון. לאחר ההתקנה, מסתיים ה-DNA להיות מצעים עניים עבור איגוד על ידי Ku70/Ku80 הטרודימר (רכיב של מסלול NHEJ) ותאים מחויבים HR17,29,30. Ku70/Ku80 הטרודימר נקשר לקצוות DSB, גיוס DNA-PKcs ו p53 מחייב חלבון 1 (53BP1)29,,30. 53BP1 משמש כמחסום לניתוק ב-G1, ובכך חוסם את משאבי האנוש תוך קידום NHEJ31,32, אך הוא מוסר באופן תלוי BRCA1 בשלב S, וכתוצאה מכך מאפשר לניתוחפעולה 33,,34. לכן, 53BP1 ו BRCA1 לשחק תפקידים מנוגדים בתיקון DSB, עם 53BP1 להיות מנחה NHEJ בעוד פעולות BRCA1 המאפשר הפסקות לתקן באמצעות משאבי אנוש.
במעבדה, היווצרות DSB יכול להיות מושרה על ידי קרינה מיונן (IR). בעוד דוגמה זו מנצלת מינון גבוה של 4 Gy, 1 Gy ו 2 Gy גם ליצור כמות משמעותית של DSBs, מתאים לניתוח של היווצרות foci על ידי חלבונים בשפע. חשוב לציין כי הסוג והמינון של קרינה בשימוש יכול להוביל נגעים שונים ב-DNA ובתא: בעוד IR גורם DSBs, זה יכול גם לגרום הפסקות גדיל יחיד או שינוי בסיס(ראה 35,36 לעיון על העברת אנרגיה ליניארית קרינה (LET) וסוג של נזק DNA). כדי לקבוע את הקינטיקה של היווצרות foci (IRIF) הנגרמת על-ידי קרינה מיוננת ואת הסיווג שלהם, אשר מצביעים על תיקון של הנזק והיפוך של DDRמופעל 8,9,37,38,היווצרותfoci ניתן לפקח בנקודות זמן שונות לאחר קרינה מיוננת. תזמון ההפעלה והאישור של כל החלבונים העיקריים לנזקי DNAידוע 39, ורבים נחקרים כסמן חלופי של אירועי מפתח. לדוגמה, pRPA, אשר בעל זיקה גבוהה עבור ssDNA משמש כתחליף של ניתוק לשבור, חלבוני MRN (MRE11, RAD50, NBS1) ו exonucleases יכול לשמש כדי להעריך יעילות השבירה מדי. בעוד RAD51, BRCA1, BRCA2 (סרטן השד סוג 2 חלבון רגישים), ו PALB2 (שותף ו localizer של BRCA2) ניתן לפקח כדי להעריך את יעילות משאבי אנוש, הנוכחות של חלבוני Ku או 53BP1, משמשים סמנים של NHEJ (איור 1).
כמו חלבונים של מכונות תיקון DNA לגייס אחד את השני לשבור ולהרכיב בסופר קומפלקסים, אינטראקציות DNA-חלבון וחלבון-חלבון ניתן להסיק על ידי ביצוע מקומיות הפרט שלהם לאורך זמן וניתוח התאמה חברתית של חלבונים, כפי שניתן לראות על ידי אותותחופפים בתא 40,41,42. בקווי תאים, המבוא של מוטציות נקודתיות או מחיקה בגנים ספציפיים לתיקון DNA או באמצעות עריכת הגנום, או על ידי דיכוי יתר של מוטציות מבוססות פלסמיד, מאפשר חקירה של שאריות ספציפיות ותפקידם האפשרי בהכרה בנזקי DNA (למשל, התאמה משותפת עם ΩH2AX) או הרכבה מורכבת (התאמה משותפת עם אחר, או מספר, חלבונים), כמו גם השפעתם על תיקון DNA. כאן, אנו משתמשים בחיסונים עקיף כדרך לחקור את היווצרות ורזולוציה של DSBs על ידי ביצוע 8H2AX foci לאורך זמן. אנו מציגים גם דוגמה אחת של היווצרות foci וניתוח התאמה למיקום אחר-תום-מקומי על-ידי שחקן מרכזי בתיקון DSB: p53 Binding Protein 1 (53BP1)32. כפי שהוזכר קודם לכן, 53BP1 נחשב מרכזי לבחירה מסלול תיקון DNA. לאחר הצטברות 53BP1 והתמצאות התקצוב ההפוך שלה עם ơH2AX מספק מידע יקר על שלב מחזור התא, הצטברות נזק DNA, מסלול המשמש לתיקון DSBs. המטרה של אימונוקולציה עקיפה היא להעריך את היעילות של תיקון נזק DNA בקווי תאים, בעקבות IR כמו במחקר זה, או לאחר חשיפה ללחצים שונים בתא, מן הצלבת DNA לחסימה של שקע שכפול (רשימה של סוכנים מזיקים DNA מסופק בטבלה 1).
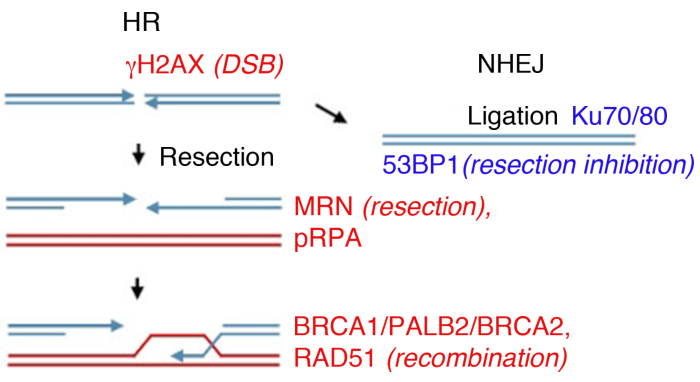
איור 1: נתיבי תיקון של שוברי גדילים כפולים של דנ”א (DSB).
תיקון DSB כולל שני מסלולים עיקריים: שילוב מחדש הומולוגי (HR, שמאל) והצטרפות לסוף לא הומולוגית (NHEJ, נכון). לאחר ההפסקה, חלבונים מופעלים כדי לסמן את ההפסקה (ơH2AX), להשתתף כריתה סוף (MRN), לכסות את ssDNA החתוכים (pRPA), לקדם שילוב מחדש (BRCA1, PALB2, BRCA2, RAD51) או להגביל את כריתת ולקדם NHEJ (53BP1). חלבונים אחרים להשתתף בתיקון נזק, אבל חלבונים המפורטים באופן שגרתי מלווה אימונו-לודיזם עקיף. לחץ כאן כדי להציג גירסה גדולה יותר של נתון זה.
| סוכן מזיק DNA | מנגנון פעולה | מינון מומלץ |
| ơ-קרני רנטגן/ קרני רנטגן | קרינה היווצרות של הפסקות דו-גדיליות עם השפעות תאיות בלתי מבוקרות |
1-4 ג’י |
| 36 00:00: יונים | קרינה היווצרות של הפסקות דו-גדיליות |
270 קילוו/μm |
| α-חלקיקים | קרינה היווצרות של הפסקות דו-גדיליות |
116 keV/μm |
| בלומיצין (Bleomycin) | מעכב סינתזת דנ”א | 0.4-2 μg/mL |
| (קמפטוסין, שוויץ) | מעכב של טופואיסומראז אני | 10-200 00 00 00 00:00:00,00 |
| צ’יספלטין (1999) | סוכן אלקילטינג (גורם קישורים תוך סטריים והצלבה) |
0.25-2 μM |
| דוקסורביצין (11 | סוכן התבות מעכב של טופואיסומראז II |
10-200 00 00 00 00:00:00,00 |
| אטופוסייד (אטופוסייד) | מעכב של טופואיסומראז II | 10 μM |
| הידרוקסיורה (הידרוקסיורה) | מעכב סינתזת דנ”א (על ידי רדוקטסה ריבונוקלאוטיד) |
10-200 μM |
| מתיל מתנסולפונט | סוכן אלקילטינג | 0.25-2 מ”מ |
| מיטומיצין C | סוכן אלקילטינג | 0.25-2 μM |
| אור אולטרה סגול (UV) | היווצרות דימי דימידין (יצירת עיוות של שרשרת ה-DNA) |
50-100 מ”ג/ס”מ2 |
שולחן 1: סוכנים גנוטוקסיים. דוגמאות של סוכני DNA מזיקים, מנגנון הפעולה שלהם ואת הנזק המושרה בהתבסס על ריכוז עבודה המוצע.
Protocol
Representative Results
Discussion
ניתוח התזמון והיעילות של תיקון נזקי DNA על ידי מיקרוסקופיה הוכיח חיוני כדי להבין איך מכונות תיקון DNA מתפקדות, בתאים נורמליים בפתולוגיות אנושיות כגון סרטן.
הפיתוח של נוגדנים ספציפיים המאפשרים זיהוי של חלבונים פעילים בגרסה הזרחן שלהם (כגון ơH2AX, pRPA, pRAD50ואחרים 10,</…
Declarações
The authors have nothing to disclose.
Acknowledgements
עבודה זו נתמכה על ידי מענק מקרן סן אנטוניו. מרכז הסרטן מינס נתמך על ידי מרכז הסרטן NCI תמיכה מענק הליבה P30 CA054174. ברצוננו להודות לסטיבן הולווי על עזרתו במיקור הריאגנטים, ועל מעבדת סונג על מתן מרחב וקיבולת מיקרוסקופיה.
Materials
| 16% (v/v) paraformaldehyde (PFA) aqueous solution | Electron Microscopy Sciences | 15710 | Microscopy quality of the PFA ensures best images. If using "home-made PFA", filter prior to use. |
| Bovine serum albumin (BSA) | Sigma-Aldrich | A3059 | Heat-shock fraction is recommended, to avoid precipitation/background. |
| Coverglass #1, 18 mm round (coverslips) | Neuvitro | NC0308920 | Coverslips need to be cleaned and sterilized prior using, with HCl or ethanol. |
| DMEM, High Glucose [(+) 4.5 g/L D-Glucose, (+) L-Glutamine, (-) Sodium Pyruvate] | Gibco | 11965118 | Adjust the growing media to the needs of cell line used. |
| DPBS, no calcium, no magnesium | Gibco | 14190144 | PBS for tissue culture. |
| Ethylene glycol-bis(β-aminoethyl ether)-N,N,N′,N′-tetraacetic acid (EGTA) | Research Products International | E57060 | Nuclear extraction buffer. |
| Fetal Bovine Serum (FBS) | Life Technologies | 104370028 | The quality of FBS can be assessed by testing gH2AX foci formation. If traces of genotoxic endotoxin are present in the batch, gH2AX will be positive in the absence of stress. |
| Magnesium chloride (MgCl2) | Research Products International | M24000 | Nuclear extraction buffer. |
| Piperazine-N,N′-bis(2-ethanesulfonic acid) (PIPES) | Research Products International | P40150 | Nuclear extraction buffer. |
| SlowFade Diamond Antifade Mountant with DAPI | Invitrogen | S36973 | 300 nM DAPI with VECTASHIELD Antifade Mounting Medium can be used instead. |
| Sodium chloride (NaCl) | Research Products International | S23020 | Nuclear extraction buffer. |
| Sucrose | Research Products International | S24060 | Nuclear extraction buffer. |
| Superfrost Plus Microscope Slides | Fisherbrand | 1255015 | Polysine Slides can be used instead. |
| TC-Treated Multiple Well Plates, size 12 wells | Costar | 3513 | Seeding on coverslips is done in 12-wells plate. |
| Triton X-100 | AmericanBio | AB02025 | Nuclear extraction buffer. |
| TrypLE Express Enzyme (1X), No Phenol Red | Gibco | 12604021 | Trypsin-EDTA can be used instead. |
| Trypsin-EDTA (0.5%), No Phenol Red | Gibco | 15400054 | TrypLE can be used instead. |
Referências
- Prakash, R., Zhang, Y., Feng, W., Jasin, M. Homologous recombination and human health: the roles of BRCA1, BRCA2, and associated proteins. Cold Spring Harbor Perspectives in Biology. 7 (4), 016600 (2015).
- Jalan, M., Olsen, K. S., Powell, S. N. Emerging Roles of RAD52 in Genome Maintenance. Cancers (Basel). 11 (7), (2019).
- Oh, J., Symington, L. S. Role of the Mre11 Complex in Preserving Genome Integrity. Genes (Basel). 9 (12), (2018).
- Uziel, T., et al. Requirement of the MRN complex for ATM activation by DNA damage. The EMBO Journal. 22 (20), 5612-5621 (2003).
- Lee, J. H., Paull, T. T. ATM activation by DNA double-strand breaks through the Mre11-Rad50-Nbs1 complex. Science. 308 (5721), 551-554 (2005).
- Rogakou, E. P., Pilch, D. R., Orr, A. H., Ivanova, V. S., Bonner, W. M. DNA double-stranded breaks induce histone H2AX phosphorylation on serine 139. Journal of Biological Chemistry. 273, 5858-5868 (1998).
- Kinner, A., Wu, W., Staudt, C., Iliakis, G. γ-H2AX in recognition and signaling of DNA double-strand breaks in the context of chromatin. Nucleic Acids Research. 36 (17), 5678-5694 (2008).
- Martin, O. A., Pilch, D. R., Redon, C., Bonner, W. M. Histone H2AX in DNA damage repair. Cancer Biology & Therapy. 2 (3), 233-235 (2003).
- Rogakou, E. P., Boon, C., Redon, C., Bonner, W. M. Megabase chromatin domains involved in DNA double-strand breaks in vivo. Journal of Cell Biology. 146 (5), 905-916 (1999).
- Stucki, M., et al. MDC1 directly binds phosphorylated histone H2AX to regulate cellular responses to DNA double-strand breaks. Cell. 123 (7), 1213-1226 (2005).
- Lou, Z., et al. MDC1 maintains genomic stability by participating in the amplification of ATM-dependent DNA damage signals. Molecular Cell. 21 (2), 187-200 (2006).
- Chapman, J. R., Jackson, S. P. Phospho-dependent interaction between NBS1 and MDC1 mediate chromatin retention of the MRN complex at sites of DNA damage. EMBO Reports. 9 (8), 795-801 (2008).
- Melander, F., et al. Phosphorylation of SDT repeats in the MDC1 N terminus triggers retention of NBS1 at the DNA damage-modified chromatin. Journal of Cell Biology. 181 (2), 213-226 (2008).
- Branzei, D., Foiani, M. Regulation of DNA repair throughout the cell cycle. Nature Review. Molecular Cell Biology. 9 (4), 297-308 (2008).
- Chiruvella, K. K., Liang, Z., Wilson, T. E. Repair of double-strand breaks by end joining. Cold Spring Harbor Perspectives in Biology. 5 (5), 012757 (2013).
- Mehta, A., Haber, J. E. Sources of DNA double-strand breaks and models of recombinational DNA repair. Cold Spring Harbor Perspectives in Biology. 6 (9), 016428 (2014).
- Symington, L. S., Gautier, J. Double-strand break end resection and repair pathway choice. Annual Review of Genetics. 45, 247-271 (2011).
- Huertas, P. DNA resection in eukaryotes: deciding how to fix the break. Nature Structural & Molecular Biology. 17 (1), 11-16 (2010).
- Nimonkar, A. V., et al. BLM-DNA2-RPA-MRN and EXO1-BLM-RPA-MRN constitute two DNA end resection machineries for human DNA break repair. Genes & Development. 25 (4), 350-362 (2011).
- Garcia, V., Phelps, S. E. L., Gray, S., Neale, M. J. Bidirectional resection of DNA double-strand breaks by Mre11 and Exo1. Nature. 479 (7372), 241-244 (2011).
- Sturzenegger, A., et al. DNA2 cooperates with the WRN and BLM RecQ helicases to mediate long-range DNA end resection in human cells. Journal of Biological Chemistry. 289 (39), 27314-27326 (2014).
- Daley, J. M., Niu, H., Miller, A. S., Sung, P. Biochemical mechanism of DSB end resection and its regulation. DNA Repair. 32, 66-74 (2015).
- Sartori, A. A., et al. Human CtIP promotes DNA end resection. Nature. 450 (7169), 509-514 (2007).
- Chen, L., Nievera, C. J., Lee, A. Y. L., Wu, X. Cell cycle-dependent complex formation of BRCA1-CtIP-MRN is important for DNA double-strand break repair. Journal of Biological Chemistry. 283, 7713-7720 (2008).
- Yun, M. H., Hiom, K. CtIP-BRCA1 modulates the choice of DNA double-strand break repair pathway throughout the cell cycle. Nature. 459 (7245), 460-463 (2009).
- Sung, P., Klein, H. Mechanism of homologous recombination: mediators and helicases take on regulatory functions. Nature Review. Molecular Cell Biology. 7, 739-750 (2006).
- San Filippo, J., Sung, P., Klein, H. Mechanisms of eukaryotic homologous recombination. Annual Review of Biochemistry. 77, 229-257 (2008).
- Jasin, M., Rothstein, R. Repair of strand breaks by homologous recombination. Cold Spring Harbor Perspectives in Biology. 5 (11), 012740 (2013).
- Dynan, W. S., Yoo, S. Interaction of Ku protein and DNA-dependent protein kinase catalytic subunit with nucleic acids. Nucleic Acids Research. 26 (7), 1551-1559 (1998).
- Lieber, M. R. The mechanism of double-strand DNA break repair by the nonhomologous DNA end-joining pathway. Annual Review of Biochemistry. 79, 181-211 (2010).
- Cejka, P. DNA end resection: nucleases team up with the right partners to initiate homologous recombination. Journal of Biological Chemistry. 290 (38), 22931-22938 (2015).
- Mirman, Z., de Lange, T. 53BP1: a DSB escort. Genes & Development. 34, 7-23 (2020).
- Cao, L., et al. A selective requirement for 53BP1 in the biological response to genomic instability induces by BRCA1 deficiency. Molecular Cell. 35 (4), 534-541 (2009).
- Zimmermann, M., de Lange, T. 53BP1: Pro choice in DNA repair. Trends in Cell Biology. 24 (2), 108-117 (2014).
- Mavragani, I. V., Nikitaki, Z., Kalospyros, S. A., Georgakilas, A. G. Ionizing Radiation and Complex DNA Damage: From Prediction to Detection Challenges and Biological Significance. Cancers (Basel). 11 (11), (2019).
- Nikitaki, Z., et al. Measurement of complex DNA damage induction and repair in human cellular systems after exposure to ionizing radiations of varying linear energy transfer (LET). Free Radical Research. 50, 64-78 (2016).
- Redon, C., et al. Histone H2A variants H2AX and H2AZ. Current Opinion in Genetics & Development. 12 (2), 162-169 (2002).
- Fernandez-Capetillo, O., Lee, A., Nussenzweig, M., Nussenzweig, A. H2AX: the histone guardian of the genome. DNA Repair. 3 (8-9), 959-967 (2004).
- Paull, T. T., et al. A critical role for histone H2AX in recruitment of repair factors to nuclear foci after DNA damage. Current Biology. 10 (15), 886-895 (2000).
- Sy, S. M. H., Huen, M. S. Y., Chen, J. PALB2 is an integral component of the BRCA complex required for homologous recombination repair. Proceedings of the National Academy of Sciences. 106 (17), 7155-7160 (2009).
- Buisson, R., Masson, J. Y. PALB2 self-interaction controls homologous recombination. Nucleic Acids Research. 40 (20), 10312-10323 (2012).
- Belotserkovskaya, R., et al. PALB2 chromatin recruitment restores homologous recombination in BRCA1-deficient cells depleted of 53BP1. Nature Communications. 11 (1), 819 (2020).
- Betts, J. A., et al. Long noncoding RNAs CUPID1 and CUPID2 mediate breast cancer risk at 11q13 by modulating the response to DNA damage. American Journal of Human Genetics. 101 (2), 255-266 (2017).
- Dray, E., et al. Molecular basis for enhancement of the meiotic DMC1 recombinase by RAD51 associated protein 1 (RAD51AP1). Proceedings of the National Academy of Sciences. 108 (9), 3560-3565 (2011).

