Laser-Capture Microdissection RNA-sekventering for rumlige og tidsmæssige vævsspecifikke genekspressionsanalyser i planter
Summary
Præsenteret her er en protokol for laser-capture microdissection (LCM) af plantevæv. LCM er en mikroskopisk teknik til isolering af vævsområder på en forureningsfri måde. Proceduren omfatter vævsfiksering, paraffinindlejring, sektionsindtrækning, LCM- og RNA-ekstraktion. RNA anvendes i den downstream-vævsspecifikke, tidsmæssigt løste analyse af transskriptomer.
Abstract
Udviklingen af en kompleks flercellet organisme styres af forskellige celletyper, der har forskellige transskriptionelle profiler. For at identificere transskriptionelle reguleringsnetværk, der styrer udviklingsprocesser, er det nødvendigt at måle de rumlige og tidsmæssige genekspressionsprofiler for disse individuelle celletyper. Derfor er indsigt i spatio-tidsmæssig kontrol af genekspression afgørende for at få forståelse for, hvordan biologiske og udviklingsmæssige processer reguleres. Her beskriver vi en laser-capture microdissection (LCM) metode til at isolere et lille antal celler fra tre byg embryo organer over en tid-kursus under spiring efterfulgt af udskrift profilering. Metoden består af vævsfiksering, vævsbehandling, paraffinindlejring, sektionsoptrækning, LCM- og RNA-ekstraktion efterfulgt af pcr eller RNA-seq i realtid. Denne metode har gjort det muligt for os at opnå rumlige og tidsmæssige profiler af frø organ transkriptomer fra varierende antal celler (tiere til hundredvis), hvilket giver langt større væv-specificitet end typiske bulk-væv analyser. Ud fra disse data var vi i stand til at definere og sammenligne transskriptive regulerende netværk samt forudsige kandidat lovgivningsmæssige transskription faktorer for de enkelte væv. Metoden bør anvendes på andre plantevæv med minimal optimering.
Introduction
Planteudvikling og vækst indebærer en koordineret indsats af transskriptionelle regulerende netværk inden for forskellige celler, der findes i et komplekst cellulært miljø. For at forstå aktiviteten af disse regulatoriske netværk, kræver vi viden om rumlige og tidsmæssige genekspression inden for forskellige celletyper på tværs af udviklingsstadier. Men, analyser af genekspression er mere almindeligt udført i hele organer eller bulk vævsprøver på grund af den tekniske udfordring at isolere og analysere et lille antal celler. Den metode, vi beskriver her, har gjort det muligt at opnå rumlig og tidsspecifik transkriptionsanalyse ved at koble LCM med RNA-seq.
LCM blev udviklet for to årtier siden af Emmert-Buck og kolleger1. Teknikken gjorde det muligt for forskere præcist at isolere enkeltceller eller klynger af celler fra deres miljø ved hjælp af direkte mikroskopisk visualisering og manipulation med en smal strålelaser1. Siden da har metoden været meget anvendt i kræftbiologi og patologi2,3. Mange planteforskningsgrupper har også tilpasset LCM til brug med forskellige plantearterogforskellige vævstyper4, 5,66,7,8,9,10,11. For nylig har flere papirer også brugt LCM på eudicot og monocot frø til at studere embryo, endosperms og andre frø strukturer under frø udvikling og spiring10,12,13. De fleste af de andre almindeligt anvendte encellede isolationsmetoder såsom mikrorør, cellesortering, magnetisk separation og mikrofluidiske platforme afhænger af enzymatisk fordøjelse eller mekanisk homogenisering for at adskille celler. Dette kan forstyrre genekspression, indføre tekniske artefakter, der forvirrede data fortolkning14,15. Disse metoder kræver også tidligere kendskab til markørgener for hver celletype for at relatere de dissocierede celler til deres rumlige placering og sande celletype. En yderligere gruppe af teknikker afhænger af affinitetsbaseret isolering af delcellulære strukturer i stedet for hele celler, for eksempel INTAKT (Isolering af nuklei mærket i celletyper) og TRAP (Oversætte Ribosome Affinity Rensning)16,17. Men affinitet mærkning og rensning af kerner eller ribosomer er teknisk udfordrende i plantearter, der ikke har veletablerede transformation protokoller. LCM udnytter hurtig vævsfiksering til at bevare transskriptionsniveauer og konventionel histologisk identifikation ved direkte visualisering af celler i deres normale vævs-/organsammenhæng, hvilket gør det muligt at isolere diskrete cellerpå kort tid 18,19.
Den protokol, der præsenteres her, er en optimeret metode til isolering af specifikke celler eller celletyper fra vævsafsnittene af kornfrø, som kan anvendes på de fleste af de celler, der kan histologisk identificeres. LCM giver en kontaktfri metode til celleisolering, hvilket i høj grad reducerer forureningen og øger integriteten af genvundet RNA. Desuden illustrerer metoden styrken af LCM på store genom brede undersøgelser startende med små mængder af biologiske materialer. Vi beskriver også lineær forstærkning af RNA til generering af tilstrækkeligt inputmateriale til downstream-transskription/transskriptionsanalyser.
Der er ti hovedtrin i denne LCM RNA-seq-protokol for rumlige og tidsmæssige vævsspecifikke transskriptioner, herunder fiksering af vævsprøver, dehydrering, paraffinfiltration, indlejring, sektionsopdelt, LCM- RNA-ekstraktion, RNA-forstærkning, RNA-kvantificering og qRT-PCR og/eller RNA-seq (Figur 1).
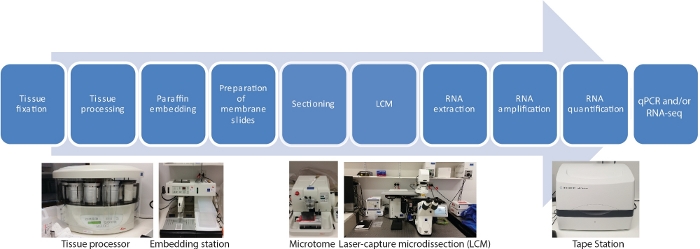
Figur 1: Rutediagram over LCM efterfulgt af RNA-seq eller qRT-PCR. LCM er en rumlig præcis og kontaktfri teknik til at indsamle celler fra faste vævssektioner ved hjælp af en laserstråle under mikroskopisk visualisering. Processen starter med fiksering af vævsprøver, efterfulgt af dehydrering ved hjælp af en gradient serie af ethanol og xylen, og færdig med paraffin infiltration. Processen kan være fuldt automatiseret ved hjælp af en vævsprocessor. Når vævet er infiltreret med paraffin, er det indlejret i en støbeform med smeltet paraffin ved hjælp af en indlejring station. Sektionssnit udføres ved hjælp af mikrotom indstillet til den ønskede tykkelse. Lysbilleder fremstilles, og LCM udføres umiddelbart før RNA skal udvindes fra tilfangetagne celler. RNA-ekstraktion efterfølges direkte af to runder RNA-forstærkning før qRT-PCR og/eller RNA-seq. Klik her for at se en større version af dette tal.
Protocol
Representative Results
Discussion
Mange vævsspecifikke genudtryksundersøgelser er blevet begrænset ved hånddissektion af prøver, som er tidskrævende, arbejdskrævende, har en høj risiko for kontaminering og kan kun udnytte prøver, som en menneskelig agent er tilstrækkelig behændning til at høste. LCM er en præcis og kontaktfri teknik til at indsamle celler fra faste vævssektioner ved hjælp af en mekanisk betjent laserstråle under mikroskopisk visualisering.
God prøve forberedelse er afgørende for LCM. Processe…
Declarações
The authors have nothing to disclose.
Acknowledgements
Dette arbejde blev støttet af Australian Research Council Centre of Excellence in Plant Energy Biology (CE140100008) til JW. M.G.L blev støttet af en La Trobe University start tilskud. Vi takker La Trobe Genomics Platform for deres støtte i høj-throughput sekventering og dataanalyse. Vi takker lektor Matthew Tucker for ekspertrådgivning om oprettelse af LCM i vores laboratorium.
Materials
| Acetic acid 100 % ACS/R. | AnalaR NORMAPUR (BioStrategies) | VWRC20104.323 | |
| AdhesiveCap 200 opaque | Zeiss | 415190-9181-000 | |
| Clear base moulds 8 X 10 | Leica | 3803015 | |
| Diethyl pyrocarbonate | Sigma-Aldrich | 40718-25ML | |
| High Sensitivity RNA ScreenTape | Agilent | 5067-5579 | |
| Lowprofile disp.blades DB80LS | Leica | 14035843489 | |
| MembraneSlide 1.0 PEN | Zeiss | 415190-9041-000 | |
| MessageAmp II aRNA Amplification Kit | Ambion (ThermoFisher) | AMB17515 | |
| On-Column DNase I Digestion Set | Sigma-Aldrich | DNASE70 | |
| Ovation RNA-Seq System V2 | NuGen (Integrated Science) | 7102-08 | |
| Paraffin (Surgipath Paraplast) | Leica | 39601006 | |
| PicoPure RNA Isolation Kit | ABI (ThermoFisher) | KIT0214 | |
| RNaseZap RNase Decontamination Solution | Ambion (ThermoFisher) | AM9780 | |
| Xylene | AnalaR NORMAPUR (BioStrategies) | VWRC28975.360 | |
| Leica Benchtop Tissue Processor | Leica Biosystems | TP1020 | |
| Leica Heated Paraffin Embedding Module | Leica Biosystems | EG1150H | |
| Leica Cold Plate | Leica Biosystems | EG1150C | |
| Safemate Class 2 Biological Safety Cabinets | LAF Technologies Pty Ltd | Safemate 1.5 | |
| Leica Fully Automated Rotary Microtome | Leica Biosystems | RM2265 | with PALMRobo v 4.6 software |
| Zeiss PALM MicroBeam LCM system | Zeiss miscroscopy | ||
| TapeStation | Agilent | TapeStation 2200 |
Referências
- Emmert-Buck, M. R., et al. Laser capture microdissection. Science. 274 (5289), 998-1001 (1996).
- Alevizos, I., et al. Oral cancer in vivo gene expression profiling assisted by laser capture microdissection and microarray analysis. Oncogene. 20 (43), 6196-6204 (2001).
- Cong, P., et al. In situ localization of follicular lymphoma: description and analysis by laser capture microdissection. Blood, The Journal of the American Society of Hematology. 99 (9), 3376-3382 (2002).
- Blokhina, O., et al. Laser capture microdissection protocol for xylem tissues of woody plants. Frontiers in Plant Science. 7, 1965 (2017).
- Casson, S., Spencer, M., Walker, K., Lindsey, K. Laser capture microdissection for the analysis of gene expression during embryogenesis of Arabidopsis. The Plant Journal. 42 (1), 111-123 (2005).
- Chen, Z., et al. LCM-seq reveals the crucial role of LsSOC1 in heat-promoted bolting of lettuce (Lactuca sativa L.). The Plant Journal. 95 (3), 516-528 (2018).
- Jiao, Y., et al. A transcriptome atlas of rice cell types uncovers cellular, functional and developmental hierarchies. Nature Genetics. 41 (2), 258-263 (2009).
- Kivivirta, K., et al. A protocol for laser microdissection (LMD) followed by transcriptome analysis of plant reproductive tissue in phylogenetically distant angiosperms. Plant Methods. 15 (1), 1-11 (2019).
- Li, P., et al. The developmental dynamics of the maize leaf transcriptome. Nature Genetics. 42 (12), 1060-1067 (2010).
- Liew, L. C., et al. Temporal tissue-specific regulation of transcriptomes during barley (Hordeum vulgare) seed germination. The Plant Journal. 101 (3), 700-715 (2020).
- Matas, A. J., et al. Tissue-and cell-type specific transcriptome profiling of expanding tomato fruit provides insights into metabolic and regulatory specialization and cuticle formation. The Plant Cell. 23 (11), 3893-3910 (2011).
- Sakai, K., et al. Combining laser-assisted microdissection (LAM) and RNA-seq allows to perform a comprehensive transcriptomic analysis of epidermal cells of Arabidopsis embryo. Plant Methods. 14 (1), 10 (2018).
- Zhan, J., et al. RNA Sequencing of Laser-Capture Microdissected compartments of the maize kernel identifies regulatory modules associated with endosperm cell differentiation. The Plant Cell. 27 (3), 513-531 (2015).
- Hwang, B., Lee, J. H., Bang, D. Single-cell RNA sequencing technologies and bioinformatics pipelines. Experimental & Molecular Medicine. 50 (8), 1-14 (2018).
- Zeb, Q., Wang, C., Shafiq, S., Liu, L. . Single-Cell Omics. , 101-135 (2019).
- Deal, R. B., Henikoff, S. The INTACT method for cell type-specific gene expression and chromatin profiling in Arabidopsis thaliana. Nature Protocols. 6 (1), 56 (2011).
- Heiman, M., Kulicke, R., Fenster, R. J., Greengard, P., Heintz, N. Cell type-specific mRNA purification by translating ribosome affinity purification (TRAP). Nature Protocols. 9 (6), 1282 (2014).
- Bevilacqua, C., Ducos, B. Laser microdissection: A powerful tool for genomics at cell level. Molecular Aspects of Medicine. 59, 5-27 (2018).
- Nelson, T., Tausta, S. L., Gandotra, N., Liu, T. Laser microdissection of plant tissue: what you see is what you get. Annual Reviews in Plant Biology. 57, 181-201 (2006).
- Day, R. C., Grossniklaus, U., Macknight, R. C. Be more specific! Laser-assisted microdissection of plant cells. Trends in Plant Science. 10 (8), 397-406 (2005).
- Takahashi, H., et al. A method for obtaining high quality RNA from paraffin sections of plant tissues by laser microdissection. Journal of Plant Research. 123 (6), 807-813 (2010).
- Schroeder, A., et al. The RIN: an RNA integrity number for assigning integrity values to RNA measurements. BMC Molecular Biology. 7 (1), 3 (2006).
- Ferreira, E. N., et al. Linear mRNA amplification approach for RNAseq from limited amount of RNA. Gene. 564 (2), 220-227 (2015).
- Schneider, J., et al. Systematic analysis of T7 RNA polymerase based in vitro linear RNA amplification for use in microarray experiments. BMC Genomics. 5 (1), 29 (2004).
- Shanker, S., et al. Evaluation of commercially available RNA amplification kits for RNA sequencing using very low input amounts of total RNA. Journal of Biomolecular Techniques. 26 (1), 4 (2015).
- Bhattacherjee, V., et al. Laser capture microdissection of fluorescently labeled embryonic cranial neural crest cells. Genesis. 39 (1), 58-64 (2004).
- Clément-Ziza, M., Munnich, A., Lyonnet, S., Jaubert, F., Besmond, C. Stabilization of RNA during laser capture microdissection by performing experiments under argon atmosphere or using ethanol as a solvent in staining solutions. RNA. 14 (12), 2698-2704 (2008).
- Blokhina, O., et al. Parenchymal Cells Contribute to Lignification of Tracheids in Developing Xylem of Norway Spruce. Plant Physiology. 181 (4), 1552-1572 (2019).
- Schad, M., Lipton, M. S., Giavalisco, P., Smith, R. D., Kehr, J. Evaluation of two-dimensional electrophoresis and liquid chromatography-tandem mass spectrometry for tissue-specific protein profiling of laser-microdissected plant samples. Electrophoresis. 26 (14), 2729-2738 (2005).
- Schad, M., Mungur, R., Fiehn, O., Kehr, J. Metabolic profiling of laser microdissected vascular bundles of Arabidopsis thaliana. Plant Methods. 1 (1), 2 (2005).
- Latrasse, D., et al. The quest for epigenetic regulation underlying unisexual flower development in Cucumis melo. Epigenetics & Chromatin. 10 (1), 22 (2017).
- Turco, G. M., et al. DNA methylation and gene expression regulation associated with vascularization in Sorghum bicolor. The New Phytologist. 214 (3), 1213-1229 (2017).
- Gomez, S. K., Harrison, M. J. Laser microdissection and its application to analyze gene expression in arbuscular mycorrhizal symbiosis. Pest Management Science: Formerly Pesticide Science. 65 (5), 504-511 (2009).
- Roux, B., et al. An integrated analysis of plant and bacterial gene expression in symbiotic root nodules using laser-capture microdissection coupled to RNA sequencing. The Plant Journal. 77 (6), 817-837 (2014).
- Tang, W., Coughlan, S., Crane, E., Beatty, M., Duvick, J. The application of laser microdissection to in planta gene expression profiling of the maize anthracnose stalk rot fungus Colletotrichum graminicola. Molecular Plant-Microbe Interactions. 19 (11), 1240-1250 (2006).

