Evaluación funcional de variantes BRCA1 utilizando editores base mediados por CRISPR
Summary
Las personas con mutaciones BRCA1 tienen un mayor riesgo de desarrollar cáncer, lo que garantiza una evaluación precisa de la función de las variantes brca1. En este documento, describimos un protocolo para la evaluación funcional de variantes BRCA1 utilizando editores base de citosina mediadas por CRISPR que permiten la conversión de C:G a T:A dirigida en células vivas.
Abstract
Estudios recientes han investigado los riesgos asociados con las mutaciones del gen BRCA1 utilizando varios métodos de evaluación funcional, como ensayos de reporteros fluorescentes, ensayos embrionarios de viabilidad de células madre y ensayos terapéuticos de sensibilidad basada en fármacos. Aunque han aclarado muchas variantes brca1, estos ensayos que implican el uso de variantes BRCA1 expresadas exógenamente están asociados con problemas de sobreexpresión y no se pueden aplicar a la regulación post-transcripcional. Para resolver estas limitaciones, anteriormente informamos de un método de análisis funcional de variantes BRCA1 a través del editor base de citosina mediada por CRISPR que inducen la sustitución de nucleótidos dirigidos en células vivas. Utilizando este método, identificamos variantes cuyas funciones siguen siendo ambiguas, incluyendo c.-97C>T, c.154C>T, c.3847C>T, c.5056C>T y c.4986+5G>A, y confirmó que los editores base mediados por CRISPR son herramientas útiles para reclasificar las variantes de importancia incierta en BRCA1. Aquí, describimos un protocolo para el análisis funcional de variantes BRCA1 utilizando el editor base de citosina basado en CRISPR. Este protocolo proporciona directrices para la selección de sitios de destino, análisis funcional y evaluación de variantes BRCA1.
Introduction
El gen de susceptibilidad tipo 1 del cáncer de mama(BRCA1)es un gen supresor tumoral ampliamente conocido. Debido a que el gen BRCA1 está relacionado con la reparación del daño del ADN, las mutaciones en este gen conducirían a un mayor riesgo de desarrollo del cáncer en un individuo1. Los cánceres de mama, ovario, próstata y páncreas están relacionados con mutaciones hereditarias de pérdida de función (LOF) del gen BRCA1 2. La evaluación funcional y la identificación de las variantes brca1 pueden ayudar a prevenir y diagnosticar las diversas enfermedades. Para abordar la función de las variantes BRCA1, se han desarrollado varios métodos y utilizados ampliamente para investigar la patogenicidad de variantes BRCA1 como ensayos embrionarios de viabilidad de células madre, ensayos de reportero fluorescente y ensayos terapéuticos de sensibilidad basada en fármacos3,4,5,6. Aunque estos métodos han evaluado la función de una gran cantidad de variantes BRCA1, los métodos que implican variantes BRCA1 expresadas exógenamente plantean limitaciones en términos de sobreexpresión que podrían afectar a la regulación aguas abajo, dosis de genes, y plegado de proteínas7. Además, estos ensayos no pueden aprovecharse para el reglamento posttranscriptional, como el empalme del ARNM, la estabilidad de la transcripción y el efecto de la región no traducida8,9.
El sistema CRISPR-Cas9 permite la edición selectiva del genoma en células y organismos vivos10. A través de un ARN mono guía, Cas9 puede inducir roturas de doble hebra (DSB) en adn cromosómico en loci genómico específico con el fin de activar dos vías de reparación del ADN: vía de unión final nohomologous (NHEJ) propensa a errores y vía11de reparación dirigida por homología sin errores (HDR). HDR es un mecanismo de reparación preciso; sin embargo, los DSB inducidos por la nucleasa Cas9 para HDR a menudo resultan en una mutación no deseada de inserción y eliminación (indel). Además, necesita plantillas homologosas de ADN de donantes para reparar el daño del ADN y tiene una eficiencia relativamente baja. Recientemente, Cas9 nickase (nCas9) se han fusionado con dominios desaminases de citidina para apuntar a sustituciones de C:G a T:A, sin necesidad de plantillas de ADN homologosas y roturas de doble hebra de ADN12,13,14,15. Utilizando el editor base de citosina, desarrollamos un nuevo método para el análisis funcional de las variantes BRCA116.
En este estudio, utilizamos el editor base de citosina mediada por CRISPR, BE314, que induce mutaciones eficientes de punto C:G a T:A, para implementar la evaluación funcional de las variantes BRCA1 e identificar con éxito las funciones de varias variantes BRCA1 (Figura 1).
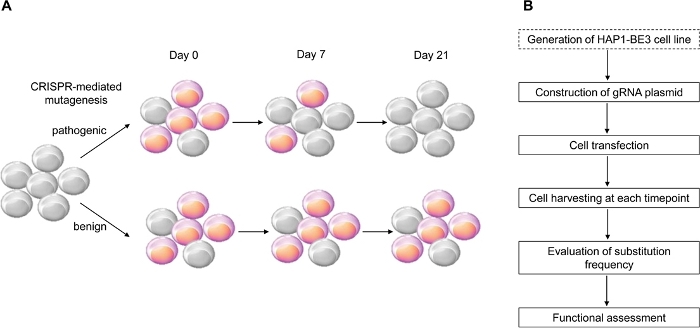
Figura 1: Una visión general del flujo de trabajo para la evaluación funcional. (A) Esquema que muestra la evaluación funcional de BRCA1. Debido a que el LOF de BRCA1 afecta la viabilidad celular, cuando la mutación BRCA1 es patógena, las células mueren a medida que aumenta el número de pasajes. B) Etapas de la evaluación funcional de BRCA1. El cuadro punteado es opcional. Puede ser reemplazado por la co-transfección de la expresión de GRNA y BE3 expresando ADN plásmidos. Haga clic aquí para ver una versión más grande de esta figura.
Protocol
Representative Results
Discussion
Este protocolo describe un método simple para evaluaciones funcionales de variantes BRCA1 utilizando el editor base de citosina meditado por CRISPR. El protocolo describe los métodos para el diseño de gRNAs en el locus objetivo y la construcción de los DNAs plásmidos a partir de los cuales se expresan. Los editores base de citosina inducen la conversión de nucleótidos en una ventana activa (en el caso de BE3, nucleótidos 4–8 en el extremo PAM-distal de las secuencias de destino de gRNA). El investigado…
Declarações
The authors have nothing to disclose.
Acknowledgements
Este trabajo fue apoyado por la Fundación Nacional de Investigación de Corea (subvenciones 2017M3A9B4062419, 2019R1F1A1057637, y 2018R1A5A2020732 a Y.K.).
Materials
| BamHI | NEB | R3136 | Restriction enzyme |
| Blasticidin | Thermo Fisher Scientific | A1113903 | Drug for selecting transduced cells |
| BsaI | NEB | R0535 | Restriction enzyme |
| DNeasy Blood & Tissue Kit | Qiagen | 69504 | Genomic DNA prep. kit |
| Dulbecco’s modified Eagle’s medium | Gibco | 11965092 | Medium for HEK293T/17 cells |
| Fetal bovine serum | Gibco | 16000036 | Supplemetal for cell culture |
| FuGENE HD Transfection Reagent | Promega | E2311 | Transfection reagent |
| Gibson Assembly Master Mix | NEB | E2611L | Gibson assembly kit |
| Iscove’s modified Dulbecco’s medium | Gibco | 12440046 | Medium for HAP1 cells |
| lentiCas9-Blast | Addgene | 52962 | Plasmids DNA for lentiBE3 cloning |
| Lipofectamine 2000 | Thermo Fisher Scientific | 11668027 | Transfection reagent |
| Opti-MEM | Gibco | 31985070 | Transfection materials |
| pCMV-BE3 | Addgene | 73021 | Plasmids DNA for lentiBE3 cloning |
| Penicillin-Streptomycin | Gibco | 15140 | Supplemetal for cell culture |
| Phusion High-Fidelity DNA Polymerase | NEB | M0530SQ | High-fidelity polymerase |
| pMD2.G | Addgene | 12259 | Plasmids DNA for virus prep. |
| pRG2 | Addgene | 104174 | gRNA cloning vector |
| psPAX2 | Addgene | 12260 | Plasmids DNA for virus prep. |
| QIAprep Spin Miniprep kit | Qiagen | 27106 | Plasmid DNA prep. Kit |
| QIAquick Gel extraction Kit | Qiagen | 28704 | Gel extraction kit |
| QIAquick PCR Purification Kit | Qiagen | 28104 | PCR product prep. kit |
| Quick Ligation Kit | NEB | M2200 | Ligase for gRNA cloning |
| T7 Endonuclease I | NEB | M0302 | Materials for T7E1 assay |
| XbaI | NEB | R0145 | Restriction enzyme |
Referências
- Roy, R., Chun, J., Powell, S. N. BRCA1 and BRCA2: different roles in a common pathway of genome protection. Nature Reviews Cancer. 12 (1), 68-78 (2011).
- Kuchenbaecker, K. B., et al. Risks of Breast, Ovarian, and Contralateral Breast Cancer for BRCA1 and BRCA2 Mutation Carriers. Journal of the American Medical Association. 317 (23), 2402-2416 (2017).
- Millot, G. A., et al. A guide for functional analysis of BRCA1 variants of uncertain significance. Human Mutation. 33 (11), 1526-1537 (2012).
- Santos, C., et al. Pathogenicity evaluation of BRCA1 and BRCA2 unclassified variants identified in Portuguese breast/ovarian cancer families. Journal of Molecular Diagnostics. 16 (3), 324-334 (2014).
- Starita, L. M., et al. A Multiplex Homology-Directed DNA Repair Assay Reveals the Impact of More Than 1,000 BRCA1 Missense Substitution Variants on Protein Function. American Journal of Human Genetics. 103 (4), 498-508 (2018).
- Anantha, R. W., et al. Functional and mutational landscapes of BRCA1 for homology-directed repair and therapy resistance. Elife. 6, (2017).
- Gibson, T. J., Seiler, M., Veitia, R. A. The transience of transient overexpression. Nature Methods. 10 (8), 715-721 (2013).
- Quann, K., Jing, Y., Rigoutsos, I. Post-transcriptional regulation of BRCA1 through its coding sequence by the miR-15/107 group of miRNAs. Frontiers in Genetics. 6, 242 (2015).
- Saunus, J. M., et al. Posttranscriptional regulation of the breast cancer susceptibility gene BRCA1 by the RNA binding protein HuR. Pesquisa do Câncer. 68 (22), 9469-9478 (2008).
- Knott1, G. J., Doudna, J. A. CRISPR-Cas guides the future of genetic engineering. Science. 361, 866-869 (2018).
- Sander, J. D., Joung, J. K. CRISPR-Cas systems for editing, regulating and targeting genomes. Nature Biotechnology. 32 (4), 347-355 (2014).
- Hess, G. T., et al. Directed evolution using dCas9-targeted somatic hypermutation in mammalian cells. Nature Methods. 13 (12), 1036-1042 (2016).
- Kim, K., et al. Highly efficient RNA-guided base editing in mouse embryos. Nature Biotechnology. 35 (5), 435-437 (2017).
- Komor, A. C., Kim, Y. B., Packer, M. S., Zuris, J. A., Liu, D. R. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature. 533 (7603), 420-424 (2016).
- Park, D. S., et al. Targeted Base Editing via RNA-Guided Cytidine Deaminases in Xenopus laevis Embryos. Molecules and Cells. 40 (11), 823-827 (2017).
- Kweon, J., et al. A CRISPR-based base-editing screen for the functional assessment of BRCA1 variants. Oncogene. 39 (1), 30-35 (2020).
- Gibson, D. G. Enzymatic assembly of overlapping DNA fragments. Methods in Enzymology. 498, 349-361 (2011).
- Nageshwaran, S., et al. CRISPR Guide RNA Cloning for Mammalian Systems. Journal of Visualized Experiments. (140), (2018).
- Findlay, G. M., et al. Accurate classification of BRCA1 variants with saturation genome editing. Nature. 562 (7726), 217-222 (2018).
- Kweon, J., Kim, D. E., Jang, A. H., Kim, Y. CRISPR/Cas-based customization of pooled CRISPR libraries. PLoS One. 13 (6), 0199473 (2018).
- Kim, Y., et al. A library of TAL effector nucleases spanning the human genome. Nature Biotechnology. 31 (3), 251-258 (2013).
- Sayers, E. W., et al. GenBank. Nucleic Acids Research. 47 (1), 94-99 (2019).
- Hwang, G. H., et al. Web-based design and analysis tools for CRISPR base editing. BMC Bioinformatics. 19 (1), 542 (2018).
- Kim, D., Kim, D. E., Lee, G., Cho, S. I., Kim, J. S. Genome-wide target specificity of CRISPR RNA-guided adenine base editors. Nature Biotechnology. 37 (4), 430-435 (2019).
- Clement, K., et al. CRISPResso2 provides accurate and rapid genome editing sequence analysis. Nature Biotechnology. 37 (3), 224-226 (2019).
- Hu, J. H., et al. Evolved Cas9 variants with broad PAM compatibility and high DNA specificity. Nature. 556 (7699), 57-63 (2018).
- Nishimasu, H., et al. Engineered CRISPR-Cas9 nuclease with expanded targeting space. Science. 361 (6408), 1259-1262 (2018).
- Walton, R. T., Christie, K. A., Whittaker, M. N., Kleinstiver, B. P. Unconstrained genome targeting with near-PAMless engineered CRISPR-Cas9 variants. Science. , (2020).
- Kim, D., et al. Genome-wide target specificities of CRISPR RNA-guided programmable deaminases. Nature Biotechnology. 35 (5), 475-480 (2017).
- Zuo, E., et al. Cytosine base editor generates substantial off-target single-nucleotide variants in mouse embryos. Science. 364 (6437), 289-292 (2019).
- Jin, S., et al. Cytosine, but not adenine, base editors induce genome-wide off-target mutations in rice. Science. 364 (6437), 292-295 (2019).
- Grunewald, J., et al. Transcriptome-wide off-target RNA editing induced by CRISPR-guided DNA base editors. Nature. 569 (7756), 433-437 (2019).
- Doman, J. L., Raguram, A., Newby, G. A., Liu, D. R. Evaluation and minimization of Cas9-independent off-target DNA editing by cytosine base editors. Nature Biotechnology. 38 (5), 620-628 (2020).
- Gaudelli, N. M., et al. Programmable base editing of A*T to G*C in genomic DNA without DNA cleavage. Nature. 551 (7681), 464-471 (2017).
- Anzalone, A. V., et al. Search-and-replace genome editing without double-strand breaks or donor DNA. Nature. 576 (7785), 149-157 (2019).

