CRISPR Aracılı Temel Editörler kullanılarak BRCA1 varyantlarının fonksiyonel değerlendirmesi
Summary
BRCA1 mutasyonu olan kişilerin kansere yakalanma riski daha yüksektir, bu da BRCA1 varyantlarının işlevinin doğru bir şekilde değerlendirilmesini garanti eder. Burada, brca1 varyantlarının canlı hücrelerde hedeflenen C:G ila T:A dönüşümünü sağlayan CRISPR aracılı sitozin baz editörleri kullanılarak işlevsel olarak değerlendirilmesi için bir protokol açıkladık.
Abstract
Son çalışmalar, floresan muhabir tahlilleri, embriyonik kök hücre canlılık tahlilleri ve terapötik ilaç bazlı duyarlılık tahlilleri gibi çeşitli fonksiyonel değerlendirme yöntemlerini kullanarak BRCA1 gen mutasyonları ile ilişkili riskleri araştırmıştır. Birçok BRCA1 varyantını açıklığa kavuşturmuş olmalarına rağmen, eksojen olarak ifade edilen BRCA1 varyantlarının kullanımını içeren bu tahliller aşırı ifade sorunlarıyla ilişkilidir ve transkripsiyon sonrası düzenlemeye uygulanamaz. Bu sınırlamaları çözmek için, daha önce CRISPR aracılı sitozin baz editörü aracılığıyla BRCA1 varyantlarının fonksiyonel analizi için canlı hücrelerde hedefli nükleotid ikamesine neden olan bir yöntem bildirdik. Bu yöntemi kullanarak, işlevleri belirsiz kalan varyantlar belirledik, c.-97C>T, c.154C>T, c.3847C>T, c.5056C>T ve c.4986+5G>A dahil olmak üzere, CRISPR aracılı temel editörlerin BRCA1’debelirsiz öneme sahip varyantları yeniden sınıflandırmak için yararlı araçlar olduğunu doğruladı. Burada, CRISPR tabanlı sitozin baz editörü kullanarak BRCA1 varyantlarının işlevsel analizi için bir protokol açıklıyoruz. Bu protokol, hedef sitelerin seçimi, BRCA1 varyantlarının işlevsel analizi ve değerlendirilmesi için yönergeler sağlar.
Introduction
Meme kanseri tip 1 duyarlılık geni (BRCA1) yaygın olarak bilinen bir tümör baskılayıcı gendir. BRCA1 geni DNA hasarının onarımı ile ilgili olduğundan, bu gendeki mutasyonlar bireyde daha fazla kanser gelişme riskine yol açacaktır1. Meme, yumurtalık, prostat ve pankreas kanserleri BRCA1 geninin kalıtsal fonksiyon kaybı (LOF) mutasyonları ile bağlantılıdır2. BRCA1 varyantlarının fonksiyonel olarak değerlendirilmesi ve tanımlanması, çeşitli hastalıkların önlenmesine ve teşhisine yardımcı olabilir. BRCA1 varyantlarının işlevini ele almak için, embriyonik kök hücre canlılık tahlilleri, floresan muhabir tahlilleri ve terapötik ilaç bazlı duyarlılık tahlilleri 3 ,4,5,6gibi BRCA1 varyantlarının patojenitesini araştırmak için çeşitli yöntemler geliştirilmiş ve yaygın olarak kullanılmaktadır. Bu yöntemler birçok BRCA1 varyantının işlevini değerlendirmiş olsa da, eksojen olarak ifade edilen BRCA1 varyantlarını içeren yöntemler, aşağı akış regülasyonunu, gen dozajını ve protein katlamayı etkileyebilecek aşırı ekspresyon açısından sınırlamalar oluşturur7. Ayrıca, bu tahliller mRNA birleştirme, transkript stabilitesi ve çevrilmemiş bölge8,9’unetkisi gibi posttranscriptional düzenlemeye yararlanılamaz.
CRISPR-Cas9 sistemi canlı hücrelerde ve organizmalarda hedefli genom düzenlemesini sağlar10. Tek kılavuzlu bir RNA aracılığıyla Cas9, iki DNA onarım yolunu etkinleştirmek için belirli genomik locide kromozomal DNA’da çift iplikçik kırılmalarına (DSB’ ler) neden olabilir: hataya eğilimli nonhomologous end-joining (NHEJ) yolu ve hatasız homolojiye yönelik onarım (HDR) yolu11. HDR hassas bir onarım mekanizmasıdır; bununla birlikte, HDR için Cas9 çekirdeği tarafından indüklenen DSB’ler genellikle istenmeyen ekleme ve silme (indel) mutasyonuna neden olur. Ek olarak, DNA hasarını onarmak için homolog donör DNA şablonlarına ihtiyaç duyar ve nispeten düşük verimliliğe sahiptir. Son zamanlarda, Cas9 nickase (nCas9), homolog DNA şablonlarına ve DNA çift iplik kırılmalarına gerek kalmadan C:G’den T:A’ya hedeflemek için sitidin deaminaz alan adlarıyla kaynaşmıştır12,13,14,15. Sitozin baz editörunu kullanarak, BRCA1 varyantlarının fonksiyonel analizi için yeni bir yöntem geliştirdik16.
Bu çalışmada, BRCA1 varyantlarının fonksiyonel değerlendirmesini uygulamak için verimli C:G ila T:A nokta mutasyonlarına neden olan CRISPR aracılı sitozin baz editörü BE314’ükullandık ve birkaç BRCA1 varyantının işlevlerini başarıyla tanımladık (Şekil 1).
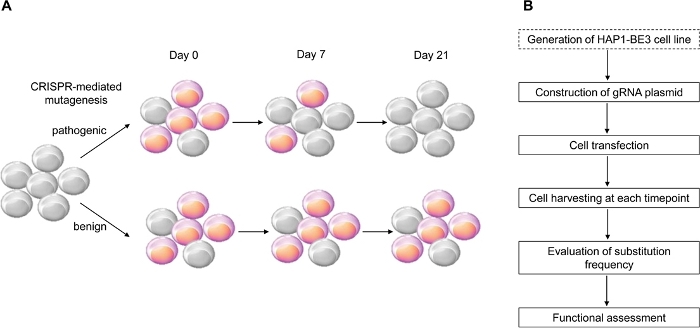
Şekil 1: İşlevsel değerlendirme için iş akışına genel bakış. (A) BRCA1’inişlevsel değerlendirmesini gösteren şematik . BRCA1 LOF’si hücre canlılığını etkilediğinden, BRCA1 mutasyonu patojenik olduğunda, geçiş sayısı arttıkça hücreler ölür. (B) BRCA1fonksiyonel değerlendirme aşamaları . Noktalı kutu isteğe bağlıdır. GRNA ekspresyonunun ko-transfeksiyon ve plazmid DNA’ları ifade eden BE3 ile değiştirilebilir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Protocol
Representative Results
Discussion
Bu protokol, CRISPR meditasyonlu sitozin baz editörü kullanılarak BRCA1 varyantlarının işlevsel değerlendirmeleri için basit bir yöntemi açıklar. Protokol, hedef lokusta gRNA’ların tasarımı ve ifade edildikleri plazmid DNA’ların yapımı için yöntemleri açıklar. Sitozin temel editörleri aktif bir pencerede nükleotid dönüşümüne neden olur (BE3 durumunda, gRNA hedef dizilerinin PAM-distal ucunda nükleotidler 4-8). Araştırmacı hedef dizileri dikkatlice seçmelidir, çünkü aktif pence…
Declarações
The authors have nothing to disclose.
Acknowledgements
Bu çalışma Kore Ulusal Araştırma Vakfı tarafından desteklendi (2017M3A9B4062419, 2019R1F1A1057637 ve 2018R1A5A2020732’yi Y.K.’ye verir).
Materials
| BamHI | NEB | R3136 | Restriction enzyme |
| Blasticidin | Thermo Fisher Scientific | A1113903 | Drug for selecting transduced cells |
| BsaI | NEB | R0535 | Restriction enzyme |
| DNeasy Blood & Tissue Kit | Qiagen | 69504 | Genomic DNA prep. kit |
| Dulbecco’s modified Eagle’s medium | Gibco | 11965092 | Medium for HEK293T/17 cells |
| Fetal bovine serum | Gibco | 16000036 | Supplemetal for cell culture |
| FuGENE HD Transfection Reagent | Promega | E2311 | Transfection reagent |
| Gibson Assembly Master Mix | NEB | E2611L | Gibson assembly kit |
| Iscove’s modified Dulbecco’s medium | Gibco | 12440046 | Medium for HAP1 cells |
| lentiCas9-Blast | Addgene | 52962 | Plasmids DNA for lentiBE3 cloning |
| Lipofectamine 2000 | Thermo Fisher Scientific | 11668027 | Transfection reagent |
| Opti-MEM | Gibco | 31985070 | Transfection materials |
| pCMV-BE3 | Addgene | 73021 | Plasmids DNA for lentiBE3 cloning |
| Penicillin-Streptomycin | Gibco | 15140 | Supplemetal for cell culture |
| Phusion High-Fidelity DNA Polymerase | NEB | M0530SQ | High-fidelity polymerase |
| pMD2.G | Addgene | 12259 | Plasmids DNA for virus prep. |
| pRG2 | Addgene | 104174 | gRNA cloning vector |
| psPAX2 | Addgene | 12260 | Plasmids DNA for virus prep. |
| QIAprep Spin Miniprep kit | Qiagen | 27106 | Plasmid DNA prep. Kit |
| QIAquick Gel extraction Kit | Qiagen | 28704 | Gel extraction kit |
| QIAquick PCR Purification Kit | Qiagen | 28104 | PCR product prep. kit |
| Quick Ligation Kit | NEB | M2200 | Ligase for gRNA cloning |
| T7 Endonuclease I | NEB | M0302 | Materials for T7E1 assay |
| XbaI | NEB | R0145 | Restriction enzyme |
Referências
- Roy, R., Chun, J., Powell, S. N. BRCA1 and BRCA2: different roles in a common pathway of genome protection. Nature Reviews Cancer. 12 (1), 68-78 (2011).
- Kuchenbaecker, K. B., et al. Risks of Breast, Ovarian, and Contralateral Breast Cancer for BRCA1 and BRCA2 Mutation Carriers. Journal of the American Medical Association. 317 (23), 2402-2416 (2017).
- Millot, G. A., et al. A guide for functional analysis of BRCA1 variants of uncertain significance. Human Mutation. 33 (11), 1526-1537 (2012).
- Santos, C., et al. Pathogenicity evaluation of BRCA1 and BRCA2 unclassified variants identified in Portuguese breast/ovarian cancer families. Journal of Molecular Diagnostics. 16 (3), 324-334 (2014).
- Starita, L. M., et al. A Multiplex Homology-Directed DNA Repair Assay Reveals the Impact of More Than 1,000 BRCA1 Missense Substitution Variants on Protein Function. American Journal of Human Genetics. 103 (4), 498-508 (2018).
- Anantha, R. W., et al. Functional and mutational landscapes of BRCA1 for homology-directed repair and therapy resistance. Elife. 6, (2017).
- Gibson, T. J., Seiler, M., Veitia, R. A. The transience of transient overexpression. Nature Methods. 10 (8), 715-721 (2013).
- Quann, K., Jing, Y., Rigoutsos, I. Post-transcriptional regulation of BRCA1 through its coding sequence by the miR-15/107 group of miRNAs. Frontiers in Genetics. 6, 242 (2015).
- Saunus, J. M., et al. Posttranscriptional regulation of the breast cancer susceptibility gene BRCA1 by the RNA binding protein HuR. Pesquisa do Câncer. 68 (22), 9469-9478 (2008).
- Knott1, G. J., Doudna, J. A. CRISPR-Cas guides the future of genetic engineering. Science. 361, 866-869 (2018).
- Sander, J. D., Joung, J. K. CRISPR-Cas systems for editing, regulating and targeting genomes. Nature Biotechnology. 32 (4), 347-355 (2014).
- Hess, G. T., et al. Directed evolution using dCas9-targeted somatic hypermutation in mammalian cells. Nature Methods. 13 (12), 1036-1042 (2016).
- Kim, K., et al. Highly efficient RNA-guided base editing in mouse embryos. Nature Biotechnology. 35 (5), 435-437 (2017).
- Komor, A. C., Kim, Y. B., Packer, M. S., Zuris, J. A., Liu, D. R. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature. 533 (7603), 420-424 (2016).
- Park, D. S., et al. Targeted Base Editing via RNA-Guided Cytidine Deaminases in Xenopus laevis Embryos. Molecules and Cells. 40 (11), 823-827 (2017).
- Kweon, J., et al. A CRISPR-based base-editing screen for the functional assessment of BRCA1 variants. Oncogene. 39 (1), 30-35 (2020).
- Gibson, D. G. Enzymatic assembly of overlapping DNA fragments. Methods in Enzymology. 498, 349-361 (2011).
- Nageshwaran, S., et al. CRISPR Guide RNA Cloning for Mammalian Systems. Journal of Visualized Experiments. (140), (2018).
- Findlay, G. M., et al. Accurate classification of BRCA1 variants with saturation genome editing. Nature. 562 (7726), 217-222 (2018).
- Kweon, J., Kim, D. E., Jang, A. H., Kim, Y. CRISPR/Cas-based customization of pooled CRISPR libraries. PLoS One. 13 (6), 0199473 (2018).
- Kim, Y., et al. A library of TAL effector nucleases spanning the human genome. Nature Biotechnology. 31 (3), 251-258 (2013).
- Sayers, E. W., et al. GenBank. Nucleic Acids Research. 47 (1), 94-99 (2019).
- Hwang, G. H., et al. Web-based design and analysis tools for CRISPR base editing. BMC Bioinformatics. 19 (1), 542 (2018).
- Kim, D., Kim, D. E., Lee, G., Cho, S. I., Kim, J. S. Genome-wide target specificity of CRISPR RNA-guided adenine base editors. Nature Biotechnology. 37 (4), 430-435 (2019).
- Clement, K., et al. CRISPResso2 provides accurate and rapid genome editing sequence analysis. Nature Biotechnology. 37 (3), 224-226 (2019).
- Hu, J. H., et al. Evolved Cas9 variants with broad PAM compatibility and high DNA specificity. Nature. 556 (7699), 57-63 (2018).
- Nishimasu, H., et al. Engineered CRISPR-Cas9 nuclease with expanded targeting space. Science. 361 (6408), 1259-1262 (2018).
- Walton, R. T., Christie, K. A., Whittaker, M. N., Kleinstiver, B. P. Unconstrained genome targeting with near-PAMless engineered CRISPR-Cas9 variants. Science. , (2020).
- Kim, D., et al. Genome-wide target specificities of CRISPR RNA-guided programmable deaminases. Nature Biotechnology. 35 (5), 475-480 (2017).
- Zuo, E., et al. Cytosine base editor generates substantial off-target single-nucleotide variants in mouse embryos. Science. 364 (6437), 289-292 (2019).
- Jin, S., et al. Cytosine, but not adenine, base editors induce genome-wide off-target mutations in rice. Science. 364 (6437), 292-295 (2019).
- Grunewald, J., et al. Transcriptome-wide off-target RNA editing induced by CRISPR-guided DNA base editors. Nature. 569 (7756), 433-437 (2019).
- Doman, J. L., Raguram, A., Newby, G. A., Liu, D. R. Evaluation and minimization of Cas9-independent off-target DNA editing by cytosine base editors. Nature Biotechnology. 38 (5), 620-628 (2020).
- Gaudelli, N. M., et al. Programmable base editing of A*T to G*C in genomic DNA without DNA cleavage. Nature. 551 (7681), 464-471 (2017).
- Anzalone, A. V., et al. Search-and-replace genome editing without double-strand breaks or donor DNA. Nature. 576 (7785), 149-157 (2019).

