Live-Cell Imaging of Single-Cell Arrays (LISCA) - eine vielseitige Technik zur Quantifizierung der zellulären Kinetik
Summary
Wir stellen eine Methode zur Erfassung von Fluoreszenz-Reporter-Zeitkursen aus einzelnen Zellen mit mikrostrukturierten Arrays vor. Das Protokoll beschreibt die Vorbereitung von Einzelzellarrays, den Aufbau und Betrieb der Live-Cell-Scanning-Zeitraffermikroskopie und ein Open-Source-Bildanalyse-Tool zur automatisierten Vorauswahl, visuellen Kontrolle und Verfolgung von zellintegrierten Fluoreszenzzeitverläufen pro Adhäsionsstelle.
Abstract
Live-cell Imaging of Single-Cell Arrays (LISCA) ist eine vielseitige Methode, um Zeitverläufe von Fluoreszenzsignalen einzelner Zellen bei hohem Durchsatz zu sammeln. Im Allgemeinen wird der Erwerb von Einzelzellzeitverläufen aus kultivierten Zellen durch die Zellmotilität und die Vielfalt der Zellformen behindert. Adhäsive Mikroarrays standardisieren Einzelzellbedingungen und erleichtern die Bildanalyse. LISCA kombiniert Einzelzell-Microarrays mit Rasterzeitraffermikroskopie und automatisierter Bildverarbeitung. Hier beschreiben wir die experimentellen Schritte der Teilnahme an einzelligen Fluoreszenzzeitkursen im LISCA-Format. Wir transfizieren Zellen, die an einem mikrostrukturierten Array haften, indem wir mRNA verwenden, das für ein verbessertes grün fluoreszierendes Protein (eGFP) kodiert, und überwachen die eGFP-Expressionskinetik von Hunderten von Zellen parallel mittels Rasterzeitraffermikroskopie. Die Bilddatenstapel werden automatisch von einer neu entwickelten Software verarbeitet, die die Fluoreszenzintensität über ausgewählte Zellkonturen integriert, um Einzelzell-Fluoreszenzzeitverläufe zu erzeugen. Wir zeigen, dass eGFP-Expressionszeitkurse nach der mRNA-Transfektion durch ein einfaches kinetisches Translationsmodell gut beschrieben werden, das Expressions- und Abbauraten von mRNA aufdeckt. Weitere Anwendungen von LISCA für Ereigniszeitkorrelationen multipler Marker im Kontext der Signalapoptose werden diskutiert.
Introduction
In den letzten Jahren hat sich die Bedeutung von Einzelzellexperimenten gezeigt. Daten aus einzelnen Zellen ermöglichen die Untersuchung der Zell-zu-Zell-Variabilität, die Auflösung intrazellulärer Parameterkorrelationen und die Detektion zellulärer Kinetik, die in Ensemblemessungen verborgen bleiben1,2,3. Um die zelluläre Kinetik von Tausenden von Einzelzellen parallel zu untersuchen, bedarf es neuer Ansätze, die es ermöglichen, die Zellen unter standardisierten Bedingungen über einen Zeitraum von mehreren Stunden bis zu mehreren Tagen zu überwachen, gefolgt von einer quantitativen Datenanalyse 4. Hier stellen wir Live-cell Imaging of Single-Cell Arrays (LISCA) vor, das den Einsatz mikrostrukturierter Arrays mit Zeitraffermikroskopie und automatisierter Bildanalyse kombiniert.
Mehrere Methoden zur Erzeugung mikrostrukturierter Einzelzellarrays wurden etabliert und in literatur5,6veröffentlicht. Hier beschreiben wir kurz das mikroskalige plasmainitiierte Proteinmuster (μPIPP). Ein detailliertes Protokoll der Einzelzellarray-Herstellung mit μPIPP finden Sie auch in Referenz7. Die Verwendung von Einzelzellarrays ermöglicht die Ausrichtung von Tausenden von Zellen auf standardisierten Adhäsionsstellen, die definierte Mikroumgebungen für jede Zelle darstellen, und reduziert so eine Quelle experimenteller Variabilität (Abbildung 1A). Einzelzellarrays werden verwendet, um die Zeitverläufe von fluoreszierenden Markern zu überwachen, die eine Vielzahl von zellulären Prozessen anzeigen sollen. Die Langzeitmikroskopie im Rasterzeitraffermodus ermöglicht die Überwachung eines großen Bereichs der Einzelzellarrays und damit die Abtastung von Einzelzelldaten in hohem Durchsatz über eine Beobachtungszeit von mehreren Stunden oder sogar Tagen. Dadurch werden Zeitlinienstapel von Bildern aus jeder Position des Arrays generiert (Abbildung 1B). Um die große Menge an Bilddaten zu reduzieren und die gewünschten Einzelzellfluoreszenzzeitverläufe effizient zu extrahieren, ist eine automatisierte Bildverarbeitung erforderlich, die sich die Positionierung der Zellen zunutze macht (Bild 1C).
Die Herausforderung von LISCA besteht darin, die experimentellen Protokolle und Berechnungswerkzeuge anzupassen, um einen Hochdurchsatz-Assay zu bilden, der quantitative und reproduzierbare Daten der zellulären Kinetik generiert. In diesem Artikel beschreiben wir Schritt für Schritt die einzelnen Methoden und wie sie in einem LISCA-Assay kombiniert werden. Als Beispiel diskutieren wir den zeitlichen Verlauf der Expression von enhanced green fluorescent protein (eGFP) nach künstlicher mRNA-Abgabe. Die eGFP-Expression nach mRNA-Abgabe wird durch Reaktionsratengleichungen beschrieben, die die Translation und den Abbau von mRNA modellieren. Die Anpassung der Modellfunktion für den zeitlichen Verlauf der eGFP-Konzentration an die LISCA-Ablesung der Fluoreszenzintensität für jede einzelne Zelle im Zeitverlauf liefert beste Schätzungen von Modellparametern wie der mRNA-Abbaurate. Als repräsentatives Ergebnis diskutieren wir die mRNA-Abgabeeffizienz von zwei verschiedenen lipidbasierten Transfektionsmitteln und wie sich ihre Parameterverteilungen unterscheiden.
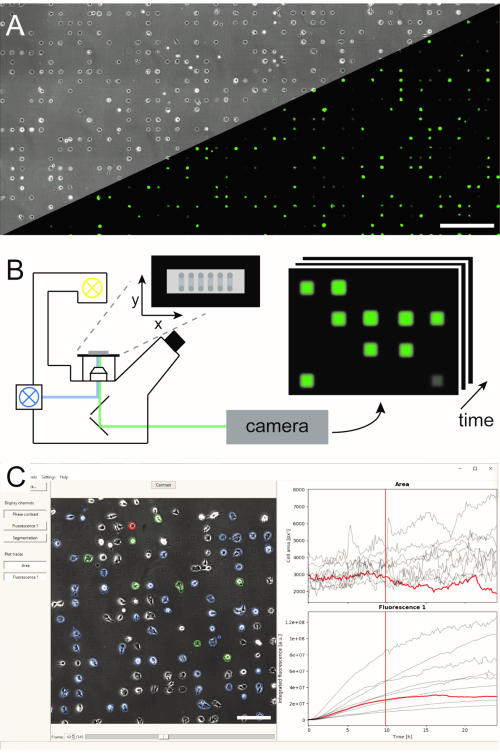
Abbildung 1: Darstellung des LISCA-Workflows, der (A) mikrogemusterte Einzelzellarrays (B), Rasterzeitraffermikroskopie und (C) automatisierte Bildanalyse von aufgezeichneten Bildserien kombiniert. Die Einzelzellarrays bestehen aus einem zweidimensionalen Muster von zellklebenden Quadraten mit einem zellabweisenden Zwischenraum, der zu einer Anordnung der Zellen auf dem Mikromuster führt, wie im Phasenkontrastbild sowie im Fluoreszenzbild von eGFP-exprimierenden Zellen (A) zu sehen ist. Der gesamte mikrostrukturierte Bereich wird im Scan-Zeitraffermodus abgebildet, wobei wiederholt Bilder an einer Sequenz von Positionen (B) aufnahmen. Aufgezeichnete Bildserien werden verarbeitet, um die Fluoreszenzintensität pro Zelle über die Zeit auszulesen (C). Maßstabsstäbe: 500 μm (A), 200 μm (C). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Protocol
Representative Results
Discussion
Hier beschrieben wir LISCA als eine vielseitige Technik, um die zelluläre Kinetik intrazellulärer fluoreszierender Markierungen auf Einzelzellebene zu verfolgen. Um ein erfolgreiches LISCA-Experiment durchführen zu können, muss jeder der beschriebenen Schritte des Protokollabschnitts einzeln festgelegt und dann alle Schritte kombiniert werden. Jeder der drei Hauptaspekte von LISCA zeichnet sich durch entscheidende Schritte aus.
Einzelzell-Microarray-Herstellung
Die Qualität de…
Declarações
The authors have nothing to disclose.
Acknowledgements
Diese Arbeit wurde durch Zuschüsse der Deutschen Forschungsgemeinschaft (DFG) zum Sonderforschungsbereich (SFB) 1032 unterstützt. Die Unterstützung durch das Bundesministerium für Bildung, Forschung und Technologie (BMBF) im Rahmen des Kooperationsprojekts 05K2018-2017-06716 Medisoft sowie eine Förderung durch die Bayerische Forschungsstiftung werden dankbar gewürdigt. Anita Reiser wurde durch ein DFG-Fellowship der Graduate School of Quantitative Biosciences Munich (QBM) gefördert.
Materials
| Adtech Polymer Engineering PTFE Microtubing | Fisher Scientific | 10178071 | |
| baking oven | Binder | 9010-0190 | |
| CFI Plan Fluor DL 10x | Nikon | MRH20100 | |
| Desiccator | Roth | NX07.1 | |
| Eclipse Ti-E | Nikon | ||
| eGFP mRNA | Trilink | L-7601 | |
| Female Luer to Tube Connector | MEDNET | FTL210-6005 | |
| Fetal bovine serum | Thermo Fisher | 10270106 | |
| Fibronectin | Yo Proteins | 663 | |
| Filter set eGFP | AHF | F46-002 | |
| Fisherbrand Translucent Platinum-Cured Silicone Tubing | Fisher Scientific | 11768088 | |
| HEPES (1 M) | Thermo Fisher | 15630080 | |
| Incubation Box | Okolab | OKO-H201 | |
| incubator | Binder | 9040-0012 | |
| L-15 without phenol red | Thermo Fisher | 21083027 | |
| Lipofectamine 2000 | Thermo Fisher | 11668027 | |
| Male Luer | in-house fabricated consisting of teflon | ||
| Male Luer to Tube Connector | MEDNET | MTLS210-6005 | alternative to in-house fabricated male luers |
| NaCl (5 M) | Thermo Fisher | AM9760G | |
| Needleless Valve to Male Luer Connector | MEDNET | NVFMLLPC | |
| NIS Elements | Nikon | Imaging software Version 5.02.00 | |
| NOA81 | Thorlabs | NOA81 | Fast Curing Optical Adhesive for tube system assembly |
| Opti-MEM | Thermo Fisher | 31985062 | |
| PCO edge 4.2 M-USB-HQ-PCO | pco | ||
| Phosphate buffered saline (PBS) | in-house prepared | ||
| Plasma Cleaner | Diener Femto | Pico-BRS | |
| PLL(20 kDa)-g[3.5]-PEG(2 kDa) | SuSoS AG | ||
| silicon wafer mit mircorstructures | in-house fabricated | ||
| Sola Light Engine | Lumencor | ||
| sticky slide VI 0.4 | ibidi | 80608 | |
| Sylgard 184 Silicone Elastomer Kit | Dow Corning | 1673921 | |
| Tango 2 | Märzhäuser | 00-24-626-0000 | |
| Ultrapure water | in-house prepared | ||
| uncoated coverslips | ibidi | 10813 | |
| Injekt-F Solo, 1 mL | Omilab | 9166017V | with replacement sporn |
Referências
- Altschuler, S. J., Wu, L. F. Cellular heterogeneity: do differences make a difference. Cell. 141 (4), 559-563 (2010).
- Locke, J. C., Elowitz, M. B. Using movies to analyse gene circuit dynamics in single cells. Nature Reviews Microbiology. 7 (5), 383-392 (2009).
- Spencer, S. L., Gaudet, S., Albeck, J. G., Burke, J. M., Sorger, P. K. Non-genetic origins of cell-to-cell variability in TRAIL-induced apoptosis. Nature. 459 (7245), 428-432 (2009).
- El-Ali, J., Sorger, P. K., Jensen, K. F. Cells on chips. Nature. 442 (7101), 403-411 (2006).
- Segerer, F. J., et al. Versatile method to generate multiple types of micropatterns. Biointerphases. 11 (1), 011005 (2016).
- Piel, M., Théry, M. . Micropatterning in cell biology, part A/B/C. , (2014).
- Reiser, A., Zorn, M. L., Murschhauser, A., Rädler, J. O., Ertl, P., Rothbauer, M. . Cell-Based Microarrays: Methods and Protocols. , 41-54 (2018).
- Picone, R., Baum, B., McKendry, R. . Methods in cell biology. 119, 73-90 (2014).
- Reiser, A. . Single-cell time courses of mRNA transport and translation kinetics. , (2019).
- . Softmatter LMU-Raedler Group Available from: https://github.com/SoftmatterLMU-RaedlerGroup/pyama (2020)
- . MIAAB, 29262 Available from: https://github.com/SoftmatterLMU-RaedlerGroup/pyama (2020)
- Reiser, A., et al. Correlation of mRNA delivery timing and protein expression in lipid-based transfection. Integrative Biology. 11 (9), 362-371 (2019).
- Reiser, A., Woschée, D., Mehrotra, N., Krzysztoń, R. S., Strey, H. H., Rädler, J. O. Supplementing data and code for: “Correlation of mRNA delivery timing and protein expression in lipid-based transfection”. Zenodo. , (2019).
- Ferizi, M., et al. Stability analysis of chemically modified mRNA using micropattern-based single-cell arrays. Lab on a Chip. 15 (17), 3561-3571 (2015).
- Fröhlich, F., et al. Multi-experiment nonlinear mixed effect modeling of single-cell translation kinetics after transfection. NPJ systems biology and applications. 4 (1), 1-12 (2018).
- Krzysztoń, R., et al. Single-cell kinetics of siRNA-mediated mRNA degradation. Nanomedicine: Nanotechnology, Biology and Medicine. 21, 102077 (2019).
- Röttgermann, P. J., Alberola, A. P., Rädler, J. O. Cellular self-organization on micro-structured surfaces. Soft Matter. 10 (14), 2397-2404 (2014).
- Röttgermann, P. J., Dawson, K. A., Rädler, J. O. Time-resolved study of nanoparticle induced apoptosis using microfabricated single cell arrays. Microarrays. 5 (2), 8 (2016).
- Murschhauser, A., et al. A high-throughput microscopy method for single-cell analysis of event-time correlations in nanoparticle-induced cell death. Communications Biology. 2 (1), 1-11 (2019).
- Chatzopoulou, E. I., et al. Chip-based platform for dynamic analysis of NK cell cytolysis mediated by a triplebody. Analyst. 141 (7), 2284-2295 (2016).
- Sztilkovics, M., et al. Single-cell adhesion force kinetics of cell populations from combined label-free optical biosensor and robotic fluidic force microscopy. Scientific Reports. 10 (1), 1-13 (2020).

