单细胞阵列的活细胞成像 (LISCA) - 量化细胞动力学的多功能技术
Summary
我们提出了一种利用微模式阵列从单个细胞获取荧光记者时间课程的方法。该协议描述了单细胞阵列的准备、活细胞扫描延时显微镜的设置和操作,以及用于自动预选、可视控制和跟踪每个粘附站点的细胞集成荧光时间课程的开源图像分析工具。
Abstract
单细胞阵列的活细胞成像 (LISCA) 是一种多功能方法,可收集来自高吞吐量单个细胞的荧光信号的时间过程。一般来说,从培养细胞获得单细胞时间课程受到细胞移动性和细胞形状多样性的阻碍。粘合微阵列标准化单细胞条件,便于图像分析。LISCA 将单细胞微阵列与扫描延时显微镜和自动图像处理相结合。在这里,我们描述了以 LISCA 格式参加单细胞荧光时间课程的实验步骤。我们通过 mRNA 编码对增强型绿色荧光蛋白 (eGFP) 进行微模式阵列的传输,并通过扫描延时显微镜同步监测数百个细胞的 eGFP 表达动力学。图像数据堆栈由新开发的软件自动处理,该软件集成了选定细胞轮廓上的荧光强度,以生成单细胞荧光时间过程。我们演示了 mRNA 转染后的 eGFP 表达时间过程,通过一个简单的动能转换模型很好地描述了 mRNA 的表达和退化率。讨论了 LISCA 在信号凋亡背景下多个标记事件时间相关性进一步应用。
Introduction
近年来,单细胞实验的重要性日益显现。来自单个细胞的数据允许研究细胞对细胞的变异性,细胞内参数相关性的分辨率和细胞动力学的检测,这些都隐藏在合奏测量1、2、3中。为了同时研究数千个单个细胞的细胞动力学,需要采用新的方法,在长达数小时、持续数天的标准化条件下对细胞进行监测,然后进行定量数据分析4。在这里,我们介绍单细胞阵列的活细胞成像 (LISCA),它结合了微结构阵列的使用与延时显微镜和自动图像分析。
在文献5、6中,已经建立并出版了几种产生微结构单细胞阵列的方法。在这里,我们简要地描述了微尺度等离子启动蛋白模式 (μPIPP)。参考文献7中还发现了使用μPIPP制造的单细胞阵列制造的详细协议。单细胞阵列的使用使数千个细胞对齐标准化粘附点,为每个细胞呈现定义的微环境,从而减少实验变异性来源(图1A)。单细胞阵列用于监控荧光标记的时间过程,这些标记旨在指示各种蜂窝过程。扫描延时模式的长期显微镜允许监测大面积的单细胞阵列,从而在几个小时甚至几天的观测时间内以高通量采样单细胞数据。这会生成来自阵列每个位置的时线图像堆栈(图1B)。为了减少大量的图像数据,并有效地提取所需的单细胞荧光时间课程,需要利用细胞定位(图1C)进行自动图像处理。
LISCA 面临的挑战是调整实验协议和计算工具,形成高通量检测,生成蜂窝动力学的定量和可重复数据。在本文中,我们逐步描述了各个方法以及它们如何在 LISCA 分析中组合。例如,我们讨论了人工mRNA传递后增强绿色荧光蛋白(eGFP)表达的时间过程。mRNA交付后的eGFP表达通过mRNA的转化和降解反应速率方程来描述。将 eGFP 浓度的时间过程的模型功能与 LISCA 读出每个细胞的荧光强度(随着时间的推移)相适应,可对 mRNA 退化率等模型参数进行最佳估计。作为一个具有代表性的结果,我们讨论了两种不同脂基转染剂的 mRNA 输送效率及其参数分布的差异。
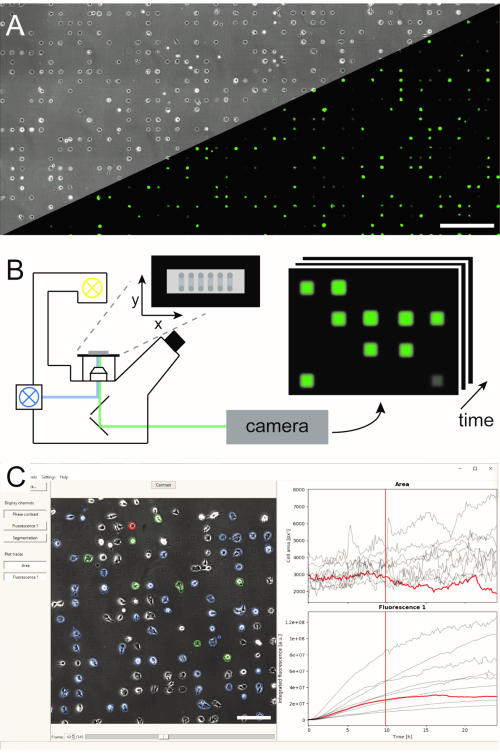
图1:LISCA 工作流程的表示,结合 (A) 微模式单细胞阵列 (B) 扫描延时显微镜和 (C) 记录图像系列的自动图像分析。单细胞阵列由细胞粘合方块的二维模式组成,具有细胞排斥的间空,可在微观图案上排列细胞,如相对比图像以及 eGFP 表达细胞(A)的荧光图像。整个微结构区域以扫描延时模式进行成像,在一系列位置(B) 中反复拍摄图像。记录的图像系列经过处理,以读出每个细胞的荧光强度(C)。比例杆:500微米(A),200微米(C)。请单击此处查看此图的较大版本。
Protocol
Representative Results
Discussion
在这里,我们描述 LISCA 是一种多功能技术,在单细胞水平上遵循细胞内荧光标签的细胞动力学。为了执行成功的 LISCA 实验,协议部分的每个描述步骤必须单独确定,然后必须合并所有步骤。LISCA 的三个主要方面都具有关键步骤。
单细胞微阵拉制造
微传讯的质量至关重要,因为微传管上的细胞对齐不仅对所有进一步的实验步骤都很重要,而且对数据质量也有影响。?…
Declarações
The authors have nothing to disclose.
Acknowledgements
这项工作得到了德国科学基金会(DFG)向合作研究中心(SFB)1032的赠款的支持。德国联邦教育、研究和技术部 (BMBF) 根据合作项目 05K2018-2017-06716 美帝索夫的支持以及巴耶里舍·福松施蒂夫通的赠款,都表示感谢。安妮塔·赖瑟通过慕尼黑定量生物科学研究生院(QBM)得到了DFG奖学金的支持。
Materials
| Adtech Polymer Engineering PTFE Microtubing | Fisher Scientific | 10178071 | |
| baking oven | Binder | 9010-0190 | |
| CFI Plan Fluor DL 10x | Nikon | MRH20100 | |
| Desiccator | Roth | NX07.1 | |
| Eclipse Ti-E | Nikon | ||
| eGFP mRNA | Trilink | L-7601 | |
| Female Luer to Tube Connector | MEDNET | FTL210-6005 | |
| Fetal bovine serum | Thermo Fisher | 10270106 | |
| Fibronectin | Yo Proteins | 663 | |
| Filter set eGFP | AHF | F46-002 | |
| Fisherbrand Translucent Platinum-Cured Silicone Tubing | Fisher Scientific | 11768088 | |
| HEPES (1 M) | Thermo Fisher | 15630080 | |
| Incubation Box | Okolab | OKO-H201 | |
| incubator | Binder | 9040-0012 | |
| L-15 without phenol red | Thermo Fisher | 21083027 | |
| Lipofectamine 2000 | Thermo Fisher | 11668027 | |
| Male Luer | in-house fabricated consisting of teflon | ||
| Male Luer to Tube Connector | MEDNET | MTLS210-6005 | alternative to in-house fabricated male luers |
| NaCl (5 M) | Thermo Fisher | AM9760G | |
| Needleless Valve to Male Luer Connector | MEDNET | NVFMLLPC | |
| NIS Elements | Nikon | Imaging software Version 5.02.00 | |
| NOA81 | Thorlabs | NOA81 | Fast Curing Optical Adhesive for tube system assembly |
| Opti-MEM | Thermo Fisher | 31985062 | |
| PCO edge 4.2 M-USB-HQ-PCO | pco | ||
| Phosphate buffered saline (PBS) | in-house prepared | ||
| Plasma Cleaner | Diener Femto | Pico-BRS | |
| PLL(20 kDa)-g[3.5]-PEG(2 kDa) | SuSoS AG | ||
| silicon wafer mit mircorstructures | in-house fabricated | ||
| Sola Light Engine | Lumencor | ||
| sticky slide VI 0.4 | ibidi | 80608 | |
| Sylgard 184 Silicone Elastomer Kit | Dow Corning | 1673921 | |
| Tango 2 | Märzhäuser | 00-24-626-0000 | |
| Ultrapure water | in-house prepared | ||
| uncoated coverslips | ibidi | 10813 | |
| Injekt-F Solo, 1 mL | Omilab | 9166017V | with replacement sporn |
Referências
- Altschuler, S. J., Wu, L. F. Cellular heterogeneity: do differences make a difference. Cell. 141 (4), 559-563 (2010).
- Locke, J. C., Elowitz, M. B. Using movies to analyse gene circuit dynamics in single cells. Nature Reviews Microbiology. 7 (5), 383-392 (2009).
- Spencer, S. L., Gaudet, S., Albeck, J. G., Burke, J. M., Sorger, P. K. Non-genetic origins of cell-to-cell variability in TRAIL-induced apoptosis. Nature. 459 (7245), 428-432 (2009).
- El-Ali, J., Sorger, P. K., Jensen, K. F. Cells on chips. Nature. 442 (7101), 403-411 (2006).
- Segerer, F. J., et al. Versatile method to generate multiple types of micropatterns. Biointerphases. 11 (1), 011005 (2016).
- Piel, M., Théry, M. . Micropatterning in cell biology, part A/B/C. , (2014).
- Reiser, A., Zorn, M. L., Murschhauser, A., Rädler, J. O., Ertl, P., Rothbauer, M. . Cell-Based Microarrays: Methods and Protocols. , 41-54 (2018).
- Picone, R., Baum, B., McKendry, R. . Methods in cell biology. 119, 73-90 (2014).
- Reiser, A. . Single-cell time courses of mRNA transport and translation kinetics. , (2019).
- . Softmatter LMU-Raedler Group Available from: https://github.com/SoftmatterLMU-RaedlerGroup/pyama (2020)
- . MIAAB, 29262 Available from: https://github.com/SoftmatterLMU-RaedlerGroup/pyama (2020)
- Reiser, A., et al. Correlation of mRNA delivery timing and protein expression in lipid-based transfection. Integrative Biology. 11 (9), 362-371 (2019).
- Reiser, A., Woschée, D., Mehrotra, N., Krzysztoń, R. S., Strey, H. H., Rädler, J. O. Supplementing data and code for: “Correlation of mRNA delivery timing and protein expression in lipid-based transfection”. Zenodo. , (2019).
- Ferizi, M., et al. Stability analysis of chemically modified mRNA using micropattern-based single-cell arrays. Lab on a Chip. 15 (17), 3561-3571 (2015).
- Fröhlich, F., et al. Multi-experiment nonlinear mixed effect modeling of single-cell translation kinetics after transfection. NPJ systems biology and applications. 4 (1), 1-12 (2018).
- Krzysztoń, R., et al. Single-cell kinetics of siRNA-mediated mRNA degradation. Nanomedicine: Nanotechnology, Biology and Medicine. 21, 102077 (2019).
- Röttgermann, P. J., Alberola, A. P., Rädler, J. O. Cellular self-organization on micro-structured surfaces. Soft Matter. 10 (14), 2397-2404 (2014).
- Röttgermann, P. J., Dawson, K. A., Rädler, J. O. Time-resolved study of nanoparticle induced apoptosis using microfabricated single cell arrays. Microarrays. 5 (2), 8 (2016).
- Murschhauser, A., et al. A high-throughput microscopy method for single-cell analysis of event-time correlations in nanoparticle-induced cell death. Communications Biology. 2 (1), 1-11 (2019).
- Chatzopoulou, E. I., et al. Chip-based platform for dynamic analysis of NK cell cytolysis mediated by a triplebody. Analyst. 141 (7), 2284-2295 (2016).
- Sztilkovics, M., et al. Single-cell adhesion force kinetics of cell populations from combined label-free optical biosensor and robotic fluidic force microscopy. Scientific Reports. 10 (1), 1-13 (2020).

