Live-cell Imaging af single-cell arrays (LISCA) - en alsidig teknik til at kvantificere Cellular Kinetics
Summary
Vi præsenterer en metode til erhvervelse af fluorescens reporter tid kurser fra enkelt celler ved hjælp af mikropatterned arrays. Protokollen beskriver forberedelsen af enkeltcellede arrays, opsætningen og driften af live-celle scanning time-lapse mikroskopi og en open source billedanalyse værktøj til automatiseret forvalg, visuel kontrol og sporing af celle-integreret fluorescens tid kurser pr vedhæftning site.
Abstract
Live-cell Imaging of Single-Cell Arrays (LISCA) er en alsidig metode til at indsamle tidsforløb af fluorescenssignaler fra individuelle celler i høj overførselshastighed. Generelt hæmmes erhvervelsen af enkeltcelletidskurser fra kultiverede celler af cellemotilitet og mangfoldighed af celleformer. Klæbende mikro-arrays standardisere enkelt-celle betingelser og lette billedanalyse. LISCA kombinerer enkeltcellemikrofi med scanning af time-lapse-mikroskopi og automatiseret billedbehandling. Her beskriver vi de eksperimentelle trin til at tage enkeltcellede fluorescenstidskurser i LISCA-format. Vi transfect celler klæber til en micropatterned array ved hjælp af mRNA kodning for forbedret grønt fluorescerende protein (eGFP) og overvåge eGFP udtryk kinetik af hundredvis af celler parallelt via scanning time-lapse mikroskopi. Billeddatastakkene behandles automatisk af nyudviklet software, der integrerer fluorescensintensiteten over udvalgte cellekonturer for at generere kurser i fluorescens i en enkelt celle. Vi demonstrerer, at eGFP udtryk tidskurser efter mRNA transfection er godt beskrevet af en simpel kinetisk oversættelse model, der afslører udtryk og nedbrydning satser for mRNA. Yderligere anvendelser af LISCA for begivenhedstid korrelationer af flere markører i forbindelse med signalering apoptose diskuteres.
Introduction
I de senere år er betydningen af enkeltcelleeksperimenter blevet tydelig. Data fra enkelte celler gør det muligt at undersøge celle-til-cellevariation, opløsningen af intracellulære parameterkorrelationer og detektion af cellulære kinetik, der forbliver skjult i ensemblemålinger1,2,3. For at undersøge cellulære kinetik af tusinder af enkeltceller parallelt er der behov for nye tilgange, der gør det muligt at overvåge cellerne under standardiserede forhold over en periode på flere timer op til flere dage efterfulgt af en kvantitativ dataanalyse 4. Her præsenterer vi Live-cell Imaging of Single-Cell Arrays (LISCA), som kombinerer brugen af mikrostrukturerede arrays med time-lapse mikroskopi og automatiseret billedanalyse.
Flere metoder til generering af mikrostrukturerede enkeltcelle arrays er blevet etableret og offentliggjort i litteratur5,6. Her beskriver vi kort Microscale Plasma-Initieret Protein Patterning (μPIPP). En detaljeret protokol for enkeltcellematrixfremstillingen ved hjælp af μPIPP findes også i reference7. Brugen af enkeltcellede arrays muliggør justering af tusindvis af celler på standardiserede vedhæftningspletter, der præsenterer definerede mikromiljøer for hver celle og reducerer dermed en kilde til eksperimentel variation (Figur 1A). Enkeltcellede arrays bruges til at overvåge tidsforløbene for fluorescerende markører, der har til formål at angive en række cellulære processer. Langsigtet mikroskopi i scanningstidsforskydningstilstand gør det muligt at overvåge et stort område af enkeltcellematrixerne og dermed udtage stikprøver af enkeltcelledata i høj overførselshastighed over en observationstid på flere timer eller endda dage. Dette genererer tidslinjestakke af billeder fra hver position af matrixen (Figur 1B). For at reducere den store mængde billeddata og udtrække de ønskede enkeltcellede fluorescenstidskurser på en effektiv måde kræves automatiseret billedbehandling, der drager fordel af placeringen af celler (Figur 1C).
Udfordringen ved LISCA er at tilpasse de eksperimentelle protokoller og beregningsværktøjer til at danne en høj gennemstrømningsanalyse, der genererer kvantitative og reproducerbare data fra cellulære kinetik. I denne artikel giver vi en trinvis beskrivelse af de enkelte metoder, og hvordan de kombineres i en LISCA-analyse. Som et eksempel diskuterer vi tidsforløbet for udvidet grønt fluorescerende protein (eGFP) udtryk efter kunstig mRNA-levering. eGFP-udtryk efter mRNA-levering beskrives ved reaktionshastighedsligninger, der modellerer oversættelse og nedbrydning af mRNA. Tilpasning af modelfunktionen for eGFP-koncentrationens tidsforløb til LISCA-udlæsningen af fluorescensintensiteten for hver enkelt celle over tid giver de bedste skøn over modelparametre som mRNA-nedbrydningshastigheden. Som et repræsentativt resultat diskuterer vi mRNA-leveringseffektiviteten af to forskellige lipidbaserede transfektmidler, og hvordan deres parameterfordelinger adskiller sig.
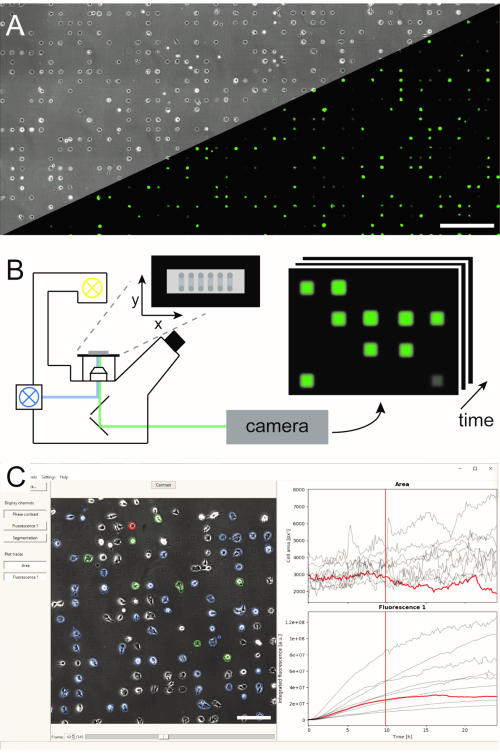
Figur 1: Repræsentation af LISCA-arbejdsgangen, der kombinerer (A) mikromønstrede enkeltcellematrixer (B) scanning af time-lapse-mikroskopi og (C) automatiseret billedanalyse af indspillede billedserier. Enkeltcellematrixerne består af et todimensionelt mønster af celleklæbende firkanter med et celleafvisende interrum, der fører til et arrangement af cellerne på mikropatternen, som det kan ses i fasekontrastbilledet samt fluorescensbilledet af eGFP, der udtrykker celler (A). Hele det mikrostrukturerede område afbildes i en scanningstidsforskydningstilstand, hvor der gentagne gange optages billeder i en sekvens af positioner (B). Optagede billedserier behandles for at aflæse fluorescensintensiteten pr. celle over tid (C). Skalastænger: 500 μm (A), 200 μm (C). Klik her for at se en større version af dette tal.
Protocol
Representative Results
Discussion
Her beskrev vi LISCA som en alsidig teknik til at følge cellulære kinetik af intracellulære fluorescerende etiketter på enkeltcelleniveau. For at kunne udføre et vellykket LISCA-eksperiment skal hvert af de beskrevne trin i protokolafsnittet fastlægges individuelt, og derefter skal alle trin kombineres. Hvert af de tre hovedaspekter af LISCA har afgørende skridt.
Fremstilling af mikroarray med en enkelt celle
Kvaliteten af mikroarray er afgørende, da den cellulære tilpasni…
Declarações
The authors have nothing to disclose.
Acknowledgements
Dette arbejde blev støttet af tilskud fra Den Tyske Videnskabsfond (DFG) til Collaborative Research Center (SFB) 1032. Støtte fra det tyske forbundsministerium for uddannelse, forskning og teknologi (BMBF) under samarbejdsprojektet 05K2018-2017-06716 Medisoft samt et tilskud fra Bayerische Forschungsstiftung er taknemmeligt anerkendt. Anita Reiser blev støttet af et DFG Fellowship gennem Graduate School of Quantitative Biosciences München (QBM).
Materials
| Adtech Polymer Engineering PTFE Microtubing | Fisher Scientific | 10178071 | |
| baking oven | Binder | 9010-0190 | |
| CFI Plan Fluor DL 10x | Nikon | MRH20100 | |
| Desiccator | Roth | NX07.1 | |
| Eclipse Ti-E | Nikon | ||
| eGFP mRNA | Trilink | L-7601 | |
| Female Luer to Tube Connector | MEDNET | FTL210-6005 | |
| Fetal bovine serum | Thermo Fisher | 10270106 | |
| Fibronectin | Yo Proteins | 663 | |
| Filter set eGFP | AHF | F46-002 | |
| Fisherbrand Translucent Platinum-Cured Silicone Tubing | Fisher Scientific | 11768088 | |
| HEPES (1 M) | Thermo Fisher | 15630080 | |
| Incubation Box | Okolab | OKO-H201 | |
| incubator | Binder | 9040-0012 | |
| L-15 without phenol red | Thermo Fisher | 21083027 | |
| Lipofectamine 2000 | Thermo Fisher | 11668027 | |
| Male Luer | in-house fabricated consisting of teflon | ||
| Male Luer to Tube Connector | MEDNET | MTLS210-6005 | alternative to in-house fabricated male luers |
| NaCl (5 M) | Thermo Fisher | AM9760G | |
| Needleless Valve to Male Luer Connector | MEDNET | NVFMLLPC | |
| NIS Elements | Nikon | Imaging software Version 5.02.00 | |
| NOA81 | Thorlabs | NOA81 | Fast Curing Optical Adhesive for tube system assembly |
| Opti-MEM | Thermo Fisher | 31985062 | |
| PCO edge 4.2 M-USB-HQ-PCO | pco | ||
| Phosphate buffered saline (PBS) | in-house prepared | ||
| Plasma Cleaner | Diener Femto | Pico-BRS | |
| PLL(20 kDa)-g[3.5]-PEG(2 kDa) | SuSoS AG | ||
| silicon wafer mit mircorstructures | in-house fabricated | ||
| Sola Light Engine | Lumencor | ||
| sticky slide VI 0.4 | ibidi | 80608 | |
| Sylgard 184 Silicone Elastomer Kit | Dow Corning | 1673921 | |
| Tango 2 | Märzhäuser | 00-24-626-0000 | |
| Ultrapure water | in-house prepared | ||
| uncoated coverslips | ibidi | 10813 | |
| Injekt-F Solo, 1 mL | Omilab | 9166017V | with replacement sporn |
Referências
- Altschuler, S. J., Wu, L. F. Cellular heterogeneity: do differences make a difference. Cell. 141 (4), 559-563 (2010).
- Locke, J. C., Elowitz, M. B. Using movies to analyse gene circuit dynamics in single cells. Nature Reviews Microbiology. 7 (5), 383-392 (2009).
- Spencer, S. L., Gaudet, S., Albeck, J. G., Burke, J. M., Sorger, P. K. Non-genetic origins of cell-to-cell variability in TRAIL-induced apoptosis. Nature. 459 (7245), 428-432 (2009).
- El-Ali, J., Sorger, P. K., Jensen, K. F. Cells on chips. Nature. 442 (7101), 403-411 (2006).
- Segerer, F. J., et al. Versatile method to generate multiple types of micropatterns. Biointerphases. 11 (1), 011005 (2016).
- Piel, M., Théry, M. . Micropatterning in cell biology, part A/B/C. , (2014).
- Reiser, A., Zorn, M. L., Murschhauser, A., Rädler, J. O., Ertl, P., Rothbauer, M. . Cell-Based Microarrays: Methods and Protocols. , 41-54 (2018).
- Picone, R., Baum, B., McKendry, R. . Methods in cell biology. 119, 73-90 (2014).
- Reiser, A. . Single-cell time courses of mRNA transport and translation kinetics. , (2019).
- . Softmatter LMU-Raedler Group Available from: https://github.com/SoftmatterLMU-RaedlerGroup/pyama (2020)
- . MIAAB, 29262 Available from: https://github.com/SoftmatterLMU-RaedlerGroup/pyama (2020)
- Reiser, A., et al. Correlation of mRNA delivery timing and protein expression in lipid-based transfection. Integrative Biology. 11 (9), 362-371 (2019).
- Reiser, A., Woschée, D., Mehrotra, N., Krzysztoń, R. S., Strey, H. H., Rädler, J. O. Supplementing data and code for: “Correlation of mRNA delivery timing and protein expression in lipid-based transfection”. Zenodo. , (2019).
- Ferizi, M., et al. Stability analysis of chemically modified mRNA using micropattern-based single-cell arrays. Lab on a Chip. 15 (17), 3561-3571 (2015).
- Fröhlich, F., et al. Multi-experiment nonlinear mixed effect modeling of single-cell translation kinetics after transfection. NPJ systems biology and applications. 4 (1), 1-12 (2018).
- Krzysztoń, R., et al. Single-cell kinetics of siRNA-mediated mRNA degradation. Nanomedicine: Nanotechnology, Biology and Medicine. 21, 102077 (2019).
- Röttgermann, P. J., Alberola, A. P., Rädler, J. O. Cellular self-organization on micro-structured surfaces. Soft Matter. 10 (14), 2397-2404 (2014).
- Röttgermann, P. J., Dawson, K. A., Rädler, J. O. Time-resolved study of nanoparticle induced apoptosis using microfabricated single cell arrays. Microarrays. 5 (2), 8 (2016).
- Murschhauser, A., et al. A high-throughput microscopy method for single-cell analysis of event-time correlations in nanoparticle-induced cell death. Communications Biology. 2 (1), 1-11 (2019).
- Chatzopoulou, E. I., et al. Chip-based platform for dynamic analysis of NK cell cytolysis mediated by a triplebody. Analyst. 141 (7), 2284-2295 (2016).
- Sztilkovics, M., et al. Single-cell adhesion force kinetics of cell populations from combined label-free optical biosensor and robotic fluidic force microscopy. Scientific Reports. 10 (1), 1-13 (2020).

