Live-cell Imaging of Single-Cell Arrays (LISCA) - een veelzijdige techniek om cellulaire kinetiek te kwantificeren
Summary
We presenteren een methode voor het verkrijgen van fluorescentie reporter tijdcursussen van enkele cellen met behulp van micropatterned arrays. Het protocol beschrijft de voorbereiding van eencellige arrays, de installatie en werking van live-cell scanning time-lapse microscopie en een open-source beeldanalysetool voor geautomatiseerde voorselectie, visuele controle en tracking van celgeïntegreerde fluorescentietijdcursussen per adhesieplaats.
Abstract
Live-cell Imaging of Single-Cell Arrays (LISCA) is een veelzijdige methode om tijdscursussen van fluorescentiesignalen van individuele cellen in hoge doorvoer te verzamelen. Over het algemeen wordt de verwerving van eencellige tijdcursussen van gekweekte cellen belemmerd door celmotiliteit en diversiteit van celvormen. Zelfklevende microarrays standaardiseren eencellige omstandigheden en vergemakkelijken beeldanalyse. LISCA combineert eencellige microarrays met scanning time-lapse microscopie en geautomatiseerde beeldverwerking. Hier beschrijven we de experimentele stappen van het volgen van eencellige fluorescentietijdcursussen in een LISCA-formaat. We transfecteren cellen die zich hechten aan een micropatterned array met behulp van mRNA-codering voor verbeterde groene fluorescerende eiwitten (eGFP) en monitoren de eGFP-expressiekinetiek van honderden cellen parallel via scanning time-lapse microscopie. De beeldgegevensstacks worden automatisch verwerkt door nieuw ontwikkelde software die fluorescentie-intensiteit over geselecteerde celcontouren integreert om fluorescentietijdcursussen met één cel te genereren. We tonen aan dat eGFP-expressietijdcursussen na mRNA-transfectie goed worden beschreven door een eenvoudig kinetisch vertaalmodel dat expressie- en afbraaksnelheden van mRNA onthult. Verdere toepassingen van LISCA voor correlaties van gebeurtenistijd van meerdere markers in de context van signalering van apoptose worden besproken.
Introduction
In de afgelopen jaren is het belang van eencellige experimenten duidelijk geworden. Gegevens van afzonderlijke cellen maken het mogelijk cel-celvariabiliteit, de resolutie van intracellulaire parametercorrelaties en de detectie van cellulaire kinetiek die verborgen blijven in ensemblemetingen1,2,3. Om de cellulaire kinetiek van duizenden afzonderlijke cellen parallel te onderzoeken, zijn nieuwe benaderingen nodig die het mogelijk maken de cellen onder gestandaardiseerde omstandigheden te monitoren over een periode van enkele uren tot enkele dagen, gevolgd door een kwantitatieve gegevensanalyse 4. Hier presenteren we Live-cell Imaging of Single-Cell Arrays (LISCA), die het gebruik van microgestructureerde arrays combineert met time-lapse microscopie en geautomatiseerde beeldanalyse.
In literatuur5,6zijn verschillende methoden voor het genereren van microgestructureerde eencellige arrays vastgesteld en gepubliceerd . Hier beschrijven we kort microschaal plasma-geïnitieerde eiwitpatronen (μPIPP). Een gedetailleerd protocol van de vervaardiging van eencellige arrays met behulp van μPIPP is ook te vinden in referentie7. Het gebruik van eencellige arrays maakt het mogelijk om duizenden cellen uit te lijnen op gestandaardiseerde hechtingspunten met gedefinieerde micromilieus voor elke cel en vermindert zo een bron van experimentele variabiliteit (figuur 1A). Eencellige arrays worden gebruikt om de tijdsvakken van fluorescerende markers te volgen die bedoeld zijn om een verscheidenheid aan cellulaire processen aan te geven. Lange termijn microscopie in de scantijdvervalmodus maakt het mogelijk om een groot deel van de eencellige arrays te bewaken en dus gegevens met één cel in hoge doorvoer te bemonsteren gedurende een observatietijd van enkele uren of zelfs dagen. Dit genereert tijdregelstapels van afbeeldingen vanaf elke positie van de matrix (figuur 1B). Om de grote hoeveelheid beeldgegevens te verminderen en de gewenste eencellige fluorescentietijdcursussen op een efficiënte manier te extraheren, is geautomatiseerde beeldverwerking vereist die profiteert van de positionering van cellen (Figuur 1C).
De uitdaging van LISCA is om de experimentele protocollen en computationele tools aan te passen om een high-throughput test te vormen die kwantitatieve en reproduceerbare gegevens van cellulaire kinetiek genereert. In dit artikel geven we een stapsgewijze beschrijving van de individuele methoden en hoe ze worden gecombineerd in een LISCA-test. Als voorbeeld bespreken we het tijdsverloop van verbeterde groene fluorescerende eiwit (eGFP) expressie na kunstmatige mRNA-levering. eGFP-expressie na mRNA-levering wordt beschreven door reactiesnelheidsvergelijkingen die de vertaling en degradatie van mRNA modelleren. Het aanpassen van de modelfunctie voor het tijdsverloop van de eGFP-concentratie aan de LISCA-uitlezing van de fluorescentie-intensiteit voor elke afzonderlijke cel in de loop van de tijd levert de beste schattingen op van modelparameters zoals de mRNA-afbraaksnelheid. Als representatief resultaat bespreken we de mRNA-leveringsefficiëntie van twee verschillende lipidegebaseerde transfectiemiddelen en hoe hun parameterverdelingen verschillen.
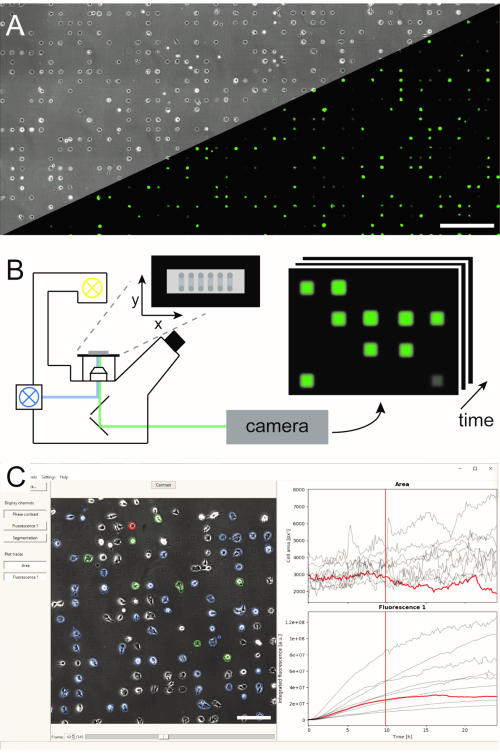
Figuur 1: Weergave van de LISCA-workflow die (A) micro-patroon eencellige arrays (B) scanning time-lapse microscopie en (C) geautomatiseerde beeldanalyse van opgenomen beeldreeksen combineert. De eencellige arrays bestaan uit een tweedimensionaal patroon van cellijme vierkanten met een celafstotende interruimte die leidt tot een rangschikking van de cellen op het micropattern, zoals te zien is in het fasecontrastbeeld en het fluorescentiebeeld van eGFP-uitdrukkende cellen (A). Het gehele microgestructureerde gebied wordt afgebeeld in een scantijdspannemodus die herhaaldelijk afbeeldingen maakt op een reeks posities (B). Opgenomen beeldreeksen worden verwerkt om de fluorescentie-intensiteit per cel in de loop van de tijd uit te lezen (C). Schaalbalken: 500 μm(A),200 μm(C). Klik hier om een grotere versie van deze afbeelding te bekijken.
Protocol
Representative Results
Discussion
Hier beschreven we LISCA als een veelzijdige techniek om cellulaire kinetiek van intracellulaire fluorescerende labels op ééncellig niveau te volgen. Om een succesvol LISCA-experiment uit te voeren, moeten alle beschreven stappen van de protocolsectie afzonderlijk worden vastgesteld en vervolgens moeten alle stappen worden gecombineerd. Elk van de drie belangrijkste aspecten van LISCA bevat cruciale stappen.
Vervaardiging van eencellige microarray
De kwaliteit van de microarray i…
Declarações
The authors have nothing to disclose.
Acknowledgements
Dit werk werd ondersteund door subsidies van de German Science Foundation (DFG) aan Collaborative Research Center (SFB) 1032. Steun van het Duitse federale ministerie van Onderwijs, Onderzoek en Technologie (BMBF) in het kader van het samenwerkingsproject 05K2018-2017-06716 Medisoft en een subsidie van de Bayerische Forschungsstiftung worden dankbaar erkend. Anita Reiser werd ondersteund door een DFG Fellowship via de Graduate School of Quantitative Biosciences Munich (QBM).
Materials
| Adtech Polymer Engineering PTFE Microtubing | Fisher Scientific | 10178071 | |
| baking oven | Binder | 9010-0190 | |
| CFI Plan Fluor DL 10x | Nikon | MRH20100 | |
| Desiccator | Roth | NX07.1 | |
| Eclipse Ti-E | Nikon | ||
| eGFP mRNA | Trilink | L-7601 | |
| Female Luer to Tube Connector | MEDNET | FTL210-6005 | |
| Fetal bovine serum | Thermo Fisher | 10270106 | |
| Fibronectin | Yo Proteins | 663 | |
| Filter set eGFP | AHF | F46-002 | |
| Fisherbrand Translucent Platinum-Cured Silicone Tubing | Fisher Scientific | 11768088 | |
| HEPES (1 M) | Thermo Fisher | 15630080 | |
| Incubation Box | Okolab | OKO-H201 | |
| incubator | Binder | 9040-0012 | |
| L-15 without phenol red | Thermo Fisher | 21083027 | |
| Lipofectamine 2000 | Thermo Fisher | 11668027 | |
| Male Luer | in-house fabricated consisting of teflon | ||
| Male Luer to Tube Connector | MEDNET | MTLS210-6005 | alternative to in-house fabricated male luers |
| NaCl (5 M) | Thermo Fisher | AM9760G | |
| Needleless Valve to Male Luer Connector | MEDNET | NVFMLLPC | |
| NIS Elements | Nikon | Imaging software Version 5.02.00 | |
| NOA81 | Thorlabs | NOA81 | Fast Curing Optical Adhesive for tube system assembly |
| Opti-MEM | Thermo Fisher | 31985062 | |
| PCO edge 4.2 M-USB-HQ-PCO | pco | ||
| Phosphate buffered saline (PBS) | in-house prepared | ||
| Plasma Cleaner | Diener Femto | Pico-BRS | |
| PLL(20 kDa)-g[3.5]-PEG(2 kDa) | SuSoS AG | ||
| silicon wafer mit mircorstructures | in-house fabricated | ||
| Sola Light Engine | Lumencor | ||
| sticky slide VI 0.4 | ibidi | 80608 | |
| Sylgard 184 Silicone Elastomer Kit | Dow Corning | 1673921 | |
| Tango 2 | Märzhäuser | 00-24-626-0000 | |
| Ultrapure water | in-house prepared | ||
| uncoated coverslips | ibidi | 10813 | |
| Injekt-F Solo, 1 mL | Omilab | 9166017V | with replacement sporn |
Referências
- Altschuler, S. J., Wu, L. F. Cellular heterogeneity: do differences make a difference. Cell. 141 (4), 559-563 (2010).
- Locke, J. C., Elowitz, M. B. Using movies to analyse gene circuit dynamics in single cells. Nature Reviews Microbiology. 7 (5), 383-392 (2009).
- Spencer, S. L., Gaudet, S., Albeck, J. G., Burke, J. M., Sorger, P. K. Non-genetic origins of cell-to-cell variability in TRAIL-induced apoptosis. Nature. 459 (7245), 428-432 (2009).
- El-Ali, J., Sorger, P. K., Jensen, K. F. Cells on chips. Nature. 442 (7101), 403-411 (2006).
- Segerer, F. J., et al. Versatile method to generate multiple types of micropatterns. Biointerphases. 11 (1), 011005 (2016).
- Piel, M., Théry, M. . Micropatterning in cell biology, part A/B/C. , (2014).
- Reiser, A., Zorn, M. L., Murschhauser, A., Rädler, J. O., Ertl, P., Rothbauer, M. . Cell-Based Microarrays: Methods and Protocols. , 41-54 (2018).
- Picone, R., Baum, B., McKendry, R. . Methods in cell biology. 119, 73-90 (2014).
- Reiser, A. . Single-cell time courses of mRNA transport and translation kinetics. , (2019).
- . Softmatter LMU-Raedler Group Available from: https://github.com/SoftmatterLMU-RaedlerGroup/pyama (2020)
- . MIAAB, 29262 Available from: https://github.com/SoftmatterLMU-RaedlerGroup/pyama (2020)
- Reiser, A., et al. Correlation of mRNA delivery timing and protein expression in lipid-based transfection. Integrative Biology. 11 (9), 362-371 (2019).
- Reiser, A., Woschée, D., Mehrotra, N., Krzysztoń, R. S., Strey, H. H., Rädler, J. O. Supplementing data and code for: “Correlation of mRNA delivery timing and protein expression in lipid-based transfection”. Zenodo. , (2019).
- Ferizi, M., et al. Stability analysis of chemically modified mRNA using micropattern-based single-cell arrays. Lab on a Chip. 15 (17), 3561-3571 (2015).
- Fröhlich, F., et al. Multi-experiment nonlinear mixed effect modeling of single-cell translation kinetics after transfection. NPJ systems biology and applications. 4 (1), 1-12 (2018).
- Krzysztoń, R., et al. Single-cell kinetics of siRNA-mediated mRNA degradation. Nanomedicine: Nanotechnology, Biology and Medicine. 21, 102077 (2019).
- Röttgermann, P. J., Alberola, A. P., Rädler, J. O. Cellular self-organization on micro-structured surfaces. Soft Matter. 10 (14), 2397-2404 (2014).
- Röttgermann, P. J., Dawson, K. A., Rädler, J. O. Time-resolved study of nanoparticle induced apoptosis using microfabricated single cell arrays. Microarrays. 5 (2), 8 (2016).
- Murschhauser, A., et al. A high-throughput microscopy method for single-cell analysis of event-time correlations in nanoparticle-induced cell death. Communications Biology. 2 (1), 1-11 (2019).
- Chatzopoulou, E. I., et al. Chip-based platform for dynamic analysis of NK cell cytolysis mediated by a triplebody. Analyst. 141 (7), 2284-2295 (2016).
- Sztilkovics, M., et al. Single-cell adhesion force kinetics of cell populations from combined label-free optical biosensor and robotic fluidic force microscopy. Scientific Reports. 10 (1), 1-13 (2020).

