הדמיית תא חי של מערכים חד-תאיים (LISCA) - טכניקה רב-תכליתית לכימות קינטיקה תאית
Summary
אנו מציגים שיטה לרכישת קורסי זמן לכתב פלואורסצנטי מתאים בודדים באמצעות מערכים micropatterned. הפרוטוקול מתאר הכנת מערכים חד-תאיים, התקנה ותפעול של מיקרוסקופיית זמן-לשגות של סריקת תאים חיים וכלי לניתוח תמונות בקוד פתוח לצורך בחירה מראש אוטומטית, בקרה חזותית ומעקב אחר קורסי זמן פלואורסצנטיים משולבים בתאים לכל אתר הידבקות.
Abstract
דימות תא חי של מערכים חד-תאיים (LISCA) היא שיטה רב-תכליתית לאיסוף קורסי זמן של אותות פלואורסצנטיים מתאים בודדים בתפוקה גבוהה. באופן כללי, רכישת קורסי זמן חד-תאיים מתאים מתורבתים מעוכבת על ידי תנועתיות התא ומגוון צורות התאים. מיקרו-מערכים דביקים מתקננים תנאים של תא יחיד ומאפשרים ניתוח תמונה. LISCA משלבת מיקרו-סדרה חד-תאית עם מיקרוסקופיה של זמן לשגות ועיבוד תמונה אוטומטי. כאן, אנו מתארים את הצעדים הניסיוניים של לקיחת קורסי זמן פלואורסצנטיות חד-תאיים בפורמט LISCA. אנו חוצצים תאים הדבקים במערך micropatterned באמצעות קידוד mRNA עבור חלבון פלואורסצנטי ירוק משופר (eGFP) ומנטרים את הקינטיקה של ביטוי eGFP של מאות תאים במקביל באמצעות סריקת מיקרוסקופיה של זמן לשגות. ערימות נתוני התמונה מעובדות באופן אוטומטי על-ידי תוכנה חדשה שפותחה המשלבת עוצמת פלואורסצנטיות על-פני קווי מתאר נבחרים של תאים כדי ליצור קורסי זמן פלואורסצנטיים של תא יחיד. אנו מראים כי קורסי זמן ביטוי eGFP לאחר transfection mRNA מתוארים היטב על ידי מודל תרגום קינטי פשוט החושף ביטוי ושיעורי השפלה של mRNA. יישומים נוספים של LISCA עבור מתאמי זמן אירוע של סמנים מרובים בהקשר של אפופטוזיס איתות נדונים.
Introduction
בשנים האחרונות ניכרת החשיבות של ניסויים חד-תאיים. נתונים מתאים בודדים מאפשרים חקירה של שונות תא לתא, רזולוציה של מתאמי פרמטרים תאיים וזיהוי קינטיקה תאית שנותרו מוסתרים במדידות אנסמבל1,2,3. על מנת לחקור קינטיקה תאית של אלפי תאים בודדים במקביל, יש צורך בגישות חדשות המאפשרות ניטור התאים בתנאים מתוקננים לאורך תקופה של מספר שעות עד מספר ימים ולאחר מכן ניתוח נתונים כמותי 4. כאן, אנו מציגים הדמיה של תא חי של מערכים חד-תאיים (LISCA), המשלבת שימוש במערכים מיקרו-מובנים עם מיקרוסקופיה של זמן לשגות וניתוח תמונה אוטומטי.
מספר שיטות ליצירת מערכים חד-תאיים מיקרו-מובנים הוקמו ופורסמו בספרות5,6. כאן, אנו מתארים בקצרה דוגמאות חלבון מיקרומטריות ביוזמת פלזמה (μPIPP). פרוטוקול מפורט של ייצור מערך תא יחיד באמצעות μPIPP נמצא גם בהתייחסות7. השימוש במערכים חד-תאיים מאפשר יישור של אלפי תאים בנקודות הידבקות סטנדרטיות המציגות מיקרו-וירוסים מוגדרים לכל תא ובכך מפחית את מקור השונות הניסיונית (איור 1A). מערכים חד-תאיים משמשים לניטור משך הזמן של סמנים פלואורסצנטיים המיועדים להצביע על מגוון תהליכים תאיים. מיקרוסקופיה לטווח ארוך במצב סריקת זמן לשגות מאפשר ניטור שטח גדול של מערכי תא יחיד ומכאן דגימת נתונים חד-תאיים בתפוקה גבוהה על פני זמן תצפית של מספר שעות או אפילו ימים. פעולה זו יוצרת ערימות של תמונות לאורך זמן מכל מיקום של המערך (איור 1B). כדי להפחית את הכמות הגדולה של נתוני התמונה ולחלץ את קורסי זמן הפלואורסצנטיות הרצויים של תא יחיד בצורה יעילה, נדרש עיבוד תמונה אוטומטי המנצל את מיקום התאים (איור 1C).
האתגר של LISCA הוא להתאים את הפרוטוקולים הניסיוניים והכלים החישוביים כדי ליצור מבחני תפוקה גבוהה המייצרים נתונים כמותיים וניתנים לשחזור של קינטיקה תאית. במאמר זה אנו מספקים תיאור שלב אחר שלב של השיטות הבודדות וכיצד הן משולבות ב- LISCA assay. כדוגמה, אנו דנים במהלך הזמן של ביטוי חלבון פלואורסצנטי ירוק משופר (eGFP) לאחר אספקת mRNA מלאכותית. ביטוי eGFP לאחר מסירת mRNA מתואר על ידי תרגום מידול משוואות קצב תגובה והשפלה של mRNA. התאמת פונקציית המודל עבור משך הזמן של ריכוז eGFP לקריאת LISCA של עוצמת הפלואורסצנטיות עבור כל תא בודד לאורך זמן מניבה את ההערכות הטובות ביותר של פרמטרי מודל כגון שיעור השפלת mRNA. כתוצאה מייצגת אנו דנים ביעילות אספקת mRNA של שני סוכני transfection מבוססי שומנים שונים וכיצד התפלגות הפרמטרים שלהם שונה.
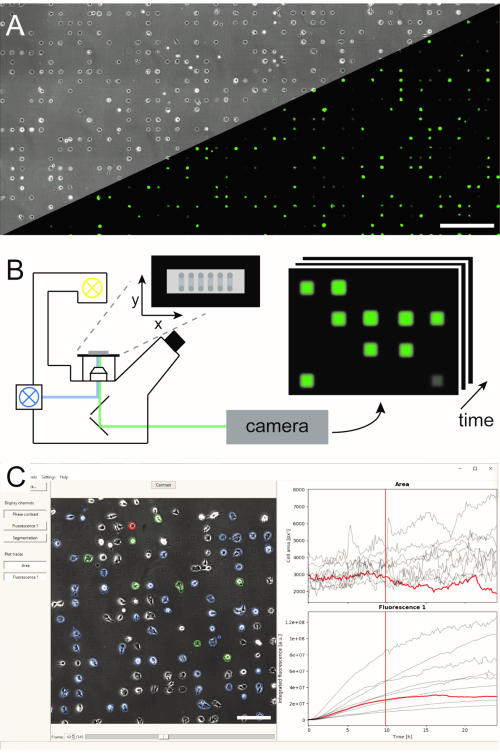
איור 1: ייצוג זרימת העבודה של LISCA המשלבת (A) מערכים חד-תאיים בעלי תבנית מיקרו (B) סורקים מיקרוסקופיה של זמן לשגות ו- (C) ניתוח תמונה אוטומטי של סדרות תמונות מוקלטות. מערכי התאים הבודדים מורכבים מתבנית דו-ממדית של ריבועי דבק תאים עם מרחב בין-מרחבי דוחה תאים המוביל לסידור התאים במיקרופטרן, כפי שניתן לראות בתמונת ניגודיות הפאזה, כמו גם בתמונת הפלואורסצנטיות של תאים המבטאים eGFP (A). האזור המיקרו-מובנה כולו נראה במצב סריקה של זמן לשגות שוב ושוב ומצלם תמונות ברצף של מיקומים (B). סדרות תמונות מוקלטות מעובדות כדי לקרוא את עוצמת הפלואורסצנטיות לכל תא לאורך זמן (C). סרגלי קנה מידה: 500 מיקרומטר (A), 200 מיקרומטר (C). לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
Protocol
Representative Results
Discussion
כאן תיארנו את LISCA כטכניקה רב-תכליתית לעקוב אחר קינטיקה תאית של תוויות פלואורסצנטיות תאיות ברמת תא יחיד. על מנת לבצע ניסוי מוצלח של LISCA, יש לקבוע כל אחד מהשלבים המתוארים בסעיף הפרוטוקול בנפרד ולאחר מכן לשלב את כל השלבים. כל אחד משלושת ההיבטים העיקריים של LISCA כולל שלבים מכריעים.
<p class="jove_content"…Declarações
The authors have nothing to disclose.
Acknowledgements
עבודה זו נתמכה על ידי מענקים של קרן המדע הגרמנית (DFG) למרכז מחקר שיתופי (SFB) 1032. תמיכה של משרד החינוך, המחקר והטכנולוגיה הפדרלי הגרמני (BMBF) במסגרת הפרויקט השיתופי 05K2018-2017-06716 Medisoft, כמו גם מענק מ Bayerische Forschungstiftung מוכרים בהכרת תודה. אניטה רייזר נתמכה על ידי מלגת DFG באמצעות בית הספר לתואר שני למדעי הביולוגיה הכמותיים במינכן (QBM).
Materials
| Adtech Polymer Engineering PTFE Microtubing | Fisher Scientific | 10178071 | |
| baking oven | Binder | 9010-0190 | |
| CFI Plan Fluor DL 10x | Nikon | MRH20100 | |
| Desiccator | Roth | NX07.1 | |
| Eclipse Ti-E | Nikon | ||
| eGFP mRNA | Trilink | L-7601 | |
| Female Luer to Tube Connector | MEDNET | FTL210-6005 | |
| Fetal bovine serum | Thermo Fisher | 10270106 | |
| Fibronectin | Yo Proteins | 663 | |
| Filter set eGFP | AHF | F46-002 | |
| Fisherbrand Translucent Platinum-Cured Silicone Tubing | Fisher Scientific | 11768088 | |
| HEPES (1 M) | Thermo Fisher | 15630080 | |
| Incubation Box | Okolab | OKO-H201 | |
| incubator | Binder | 9040-0012 | |
| L-15 without phenol red | Thermo Fisher | 21083027 | |
| Lipofectamine 2000 | Thermo Fisher | 11668027 | |
| Male Luer | in-house fabricated consisting of teflon | ||
| Male Luer to Tube Connector | MEDNET | MTLS210-6005 | alternative to in-house fabricated male luers |
| NaCl (5 M) | Thermo Fisher | AM9760G | |
| Needleless Valve to Male Luer Connector | MEDNET | NVFMLLPC | |
| NIS Elements | Nikon | Imaging software Version 5.02.00 | |
| NOA81 | Thorlabs | NOA81 | Fast Curing Optical Adhesive for tube system assembly |
| Opti-MEM | Thermo Fisher | 31985062 | |
| PCO edge 4.2 M-USB-HQ-PCO | pco | ||
| Phosphate buffered saline (PBS) | in-house prepared | ||
| Plasma Cleaner | Diener Femto | Pico-BRS | |
| PLL(20 kDa)-g[3.5]-PEG(2 kDa) | SuSoS AG | ||
| silicon wafer mit mircorstructures | in-house fabricated | ||
| Sola Light Engine | Lumencor | ||
| sticky slide VI 0.4 | ibidi | 80608 | |
| Sylgard 184 Silicone Elastomer Kit | Dow Corning | 1673921 | |
| Tango 2 | Märzhäuser | 00-24-626-0000 | |
| Ultrapure water | in-house prepared | ||
| uncoated coverslips | ibidi | 10813 | |
| Injekt-F Solo, 1 mL | Omilab | 9166017V | with replacement sporn |
Referências
- Altschuler, S. J., Wu, L. F. Cellular heterogeneity: do differences make a difference. Cell. 141 (4), 559-563 (2010).
- Locke, J. C., Elowitz, M. B. Using movies to analyse gene circuit dynamics in single cells. Nature Reviews Microbiology. 7 (5), 383-392 (2009).
- Spencer, S. L., Gaudet, S., Albeck, J. G., Burke, J. M., Sorger, P. K. Non-genetic origins of cell-to-cell variability in TRAIL-induced apoptosis. Nature. 459 (7245), 428-432 (2009).
- El-Ali, J., Sorger, P. K., Jensen, K. F. Cells on chips. Nature. 442 (7101), 403-411 (2006).
- Segerer, F. J., et al. Versatile method to generate multiple types of micropatterns. Biointerphases. 11 (1), 011005 (2016).
- Piel, M., Théry, M. . Micropatterning in cell biology, part A/B/C. , (2014).
- Reiser, A., Zorn, M. L., Murschhauser, A., Rädler, J. O., Ertl, P., Rothbauer, M. . Cell-Based Microarrays: Methods and Protocols. , 41-54 (2018).
- Picone, R., Baum, B., McKendry, R. . Methods in cell biology. 119, 73-90 (2014).
- Reiser, A. . Single-cell time courses of mRNA transport and translation kinetics. , (2019).
- . Softmatter LMU-Raedler Group Available from: https://github.com/SoftmatterLMU-RaedlerGroup/pyama (2020)
- . MIAAB, 29262 Available from: https://github.com/SoftmatterLMU-RaedlerGroup/pyama (2020)
- Reiser, A., et al. Correlation of mRNA delivery timing and protein expression in lipid-based transfection. Integrative Biology. 11 (9), 362-371 (2019).
- Reiser, A., Woschée, D., Mehrotra, N., Krzysztoń, R. S., Strey, H. H., Rädler, J. O. Supplementing data and code for: “Correlation of mRNA delivery timing and protein expression in lipid-based transfection”. Zenodo. , (2019).
- Ferizi, M., et al. Stability analysis of chemically modified mRNA using micropattern-based single-cell arrays. Lab on a Chip. 15 (17), 3561-3571 (2015).
- Fröhlich, F., et al. Multi-experiment nonlinear mixed effect modeling of single-cell translation kinetics after transfection. NPJ systems biology and applications. 4 (1), 1-12 (2018).
- Krzysztoń, R., et al. Single-cell kinetics of siRNA-mediated mRNA degradation. Nanomedicine: Nanotechnology, Biology and Medicine. 21, 102077 (2019).
- Röttgermann, P. J., Alberola, A. P., Rädler, J. O. Cellular self-organization on micro-structured surfaces. Soft Matter. 10 (14), 2397-2404 (2014).
- Röttgermann, P. J., Dawson, K. A., Rädler, J. O. Time-resolved study of nanoparticle induced apoptosis using microfabricated single cell arrays. Microarrays. 5 (2), 8 (2016).
- Murschhauser, A., et al. A high-throughput microscopy method for single-cell analysis of event-time correlations in nanoparticle-induced cell death. Communications Biology. 2 (1), 1-11 (2019).
- Chatzopoulou, E. I., et al. Chip-based platform for dynamic analysis of NK cell cytolysis mediated by a triplebody. Analyst. 141 (7), 2284-2295 (2016).
- Sztilkovics, M., et al. Single-cell adhesion force kinetics of cell populations from combined label-free optical biosensor and robotic fluidic force microscopy. Scientific Reports. 10 (1), 1-13 (2020).

