सिंगल-सेल सरेर (LISCA) की लाइव-सेल इमेजिंग - सेलुलर काइनेटिक्स की मात्रा निर्धारित करने के लिए एक बहुमुखी तकनीक
Summary
हम माइक्रोपैटर्न्ड सरणी का उपयोग करके एकल कोशिकाओं से फ्लोरेसेंस रिपोर्टर टाइम कोर्सेज के अधिग्रहण के लिए एक विधि प्रस्तुत करते हैं। प्रोटोकॉल एकल सेल सरणी की तैयारी, लाइव-सेल स्कैनिंग टाइम-लैप्स माइक्रोस्कोपी के सेटअप और संचालन और स्वचालित चयन के लिए एक ओपन-सोर्स इमेज एनालिसिस टूल, ऑडनेस साइट के अनुसार सेल-एकीकृत फ्लोरेसेंस टाइम कोर्सेज की ट्रैकिंग का वर्णन करता है।
Abstract
सिंगल-सेल ऐरे (एलएससीए) का लाइव-सेल इमेजिंग उच्च थ्रूपुट में व्यक्तिगत कोशिकाओं से फ्लोरेसेंस संकेतों के समय पाठ्यक्रम एकत्र करने के लिए एक बहुमुखी विधि है। सामान्य तौर पर, सुसंस्कृत कोशिकाओं से एकल कोशिका समय पाठ्यक्रमों का अधिग्रहण सेल गतिशीलता और कोशिका आकार की विविधता से बाधित होता है। चिपकने वाले माइक्रो-सरणी एकल-कोशिका स्थितियों को मानकीकृत करते हैं और छवि विश्लेषण की सुविधा प्रदान करते हैं। LISCA स्कैनिंग समय-चूक माइक्रोस्कोपी और स्वचालित छवि प्रसंस्करण के साथ एकल सेल microarrays को जोड़ती है । यहां, हम एलएससीए प्रारूप में एकल-कोशिका फ्लोरेसेंस टाइम कोर्स लेने के प्रयोगात्मक चरणों का वर्णन करते हैं। हम उन्नत हरे फ्लोरोसेंट प्रोटीन (ईजीएफपी) के लिए एमआरएनए एन्कोडिंग का उपयोग करके एक माइक्रोपैटर्न्ड सरणी के अनुयायी कोशिकाओं को स्थानांतरित करते हैं और स्कैनिंग टाइम-लैप्स माइक्रोस्कोपी के माध्यम से समानांतर में सैकड़ों कोशिकाओं के eGFP अभिव्यक्ति गतिज की निगरानी करते हैं। छवि डेटा स्टैक स्वचालित रूप से नए विकसित सॉफ्टवेयर द्वारा संसाधित किए जाते हैं जो एकल-सेल फ्लोरेसेंस टाइम कोर्स उत्पन्न करने के लिए चयनित सेल आकृति पर फ्लोरेसेंस तीव्रता को एकीकृत करते हैं। हम प्रदर्शित करते हैं कि एमआरएनए ट्रांसफैक्शन के बाद ईजीएफपी अभिव्यक्ति समय पाठ्यक्रमों को एक साधारण गतिज अनुवाद मॉडल द्वारा अच्छी तरह से वर्णित किया गया है जो एमआरएनए की अभिव्यक्ति और गिरावट दरों को प्रकट करता है। एपोप्टोसिस के संकेत के संदर्भ में कई मार्कर के इवेंट टाइम सहसंबंधों के लिए LISCA के आगे के अनुप्रयोगों पर चर्चा की जाती है।
Introduction
हाल के वर्षों में, एकल कोशिका प्रयोगों का महत्व स्पष्ट हो गया है । एकल कोशिकाओं के डेटा सेल-टू-सेल परिवर्तनशीलता की जांच, इंट्रासेलुलर पैरामीटर सहसंबंधों के संकल्प और सेलुलर काइनेटिक्स का पता लगाने की अनुमति देते हैं जो कलाकारों की टुकड़ी माप1,2,3में छिपे रहते हैं। समानांतर में हजारों एकल कोशिकाओं के सेलुलर काइनेटिक्स की जांच करने के लिए, नए दृष्टिकोणों की आवश्यकता होती है जो कई दिनों तक कई घंटों की समय अवधि में मानकीकृत परिस्थितियों में कोशिकाओं की निगरानी करने में सक्षम होते हैं और इसके बाद मात्रात्मक डेटा विश्लेषण 4होता है। यहां, हम सिंगल-सेल ऐरे (LISCA) की लाइव-सेल इमेजिंग पेश करते हैं, जो समय-चूक माइक्रोस्कोपी और स्वचालित छवि विश्लेषण के साथ माइक्रोस्ट्रक्चर्ड सरणी के उपयोग को जोड़ती है।
माइक्रोस्ट्रक्चर्ड एकल-कोशिका सरणी उत्पन्न करने के लिए कई तरीके स्थापित किए गए हैं और साहित्य5,6में प्रकाशित किए गए हैं। यहां, हम संक्षेप में माइक्रोस्केल प्लाज्मा-शुरू प्रोटीन पैटर्निंग (μPIPP) का वर्णन करते हैं। μPIPP का उपयोग करके एकल-सेल सरणी निर्माण का एक विस्तृत प्रोटोकॉल भी संदर्भ7में पाया गया है। एकल-कोशिका सरणी का उपयोग प्रत्येक कोशिका के लिए परिभाषित माइक्रोएनवायरमेंट्स को प्रस्तुत करने वाले मानकीकृत आसंजन स्थानों पर हजारों कोशिकाओं के संरेखण को सक्षम बनाता है और इस प्रकार प्रयोगात्मक परिवर्तनशीलता(चित्र 1 ए)के स्रोत को कम कर देता है। विभिन्न प्रकार की सेलुलर प्रक्रियाओं को इंगित करने के उद्देश्य से फ्लोरोसेंट मार्कर के समय पाठ्यक्रमों की निगरानी के लिए एकल-सेल सरणी का उपयोग किया जाता है। स्कैनिंग टाइम-लैप्स मोड में दीर्घकालिक माइक्रोस्कोपी एकल-सेल सरणी के एक बड़े क्षेत्र की निगरानी के लिए अनुमति देती है और इसलिए कई घंटों या दिनों के अवलोकन समय पर उच्च-थ्रूपुट में एकल-सेल डेटा का नमूना लेते हैं। यह सरणी(चित्रा 1B)की प्रत्येक स्थिति से छवियों के समय-रेखा ढेर उत्पन्न करता है। बड़ी मात्रा में छवि डेटा को कम करने और वांछित एकल-कोशिका फ्लोरेसेंस टाइम पाठ्यक्रमों को कुशल तरीके से निकालने के लिए, स्वचालित छवि प्रसंस्करण की आवश्यकता होती है जो कोशिकाओं की स्थिति(चित्रा 1 सी)का लाभ उठाती है।
LISCA की चुनौती एक उच्च थ्रूपुट परख बनाने के लिए प्रयोगात्मक प्रोटोकॉल और कम्प्यूटेशनल उपकरणों को अनुकूलित करना है जो सेलुलर काइनेटिक्स के मात्रात्मक और प्रजनन योग्य डेटा उत्पन्न करता है। इस लेख में हम व्यक्तिगत तरीकों का एक कदम-दर-कदम विवरण प्रदान करते हैं और उन्हें LISCA परख में कैसे जोड़ा जाता है। एक उदाहरण के रूप में, हम कृत्रिम एमआरएनए डिलीवरी के बाद उन्नत हरे फ्लोरोसेंट प्रोटीन (ईजीएफपी) अभिव्यक्ति के समय पाठ्यक्रम पर चर्चा करते हैं। एमआरएनए डिलीवरी के बाद ईजीएफपी अभिव्यक्ति को प्रतिक्रिया दर समीकरणों मॉडलिंग अनुवाद और एमआरएनए के क्षरण द्वारा वर्णित किया गया है। समय के साथ प्रत्येक व्यक्ति कोशिका के लिए फ्लोरेसेंस तीव्रता के LISCA readout के लिए LISFP एकाग्रता के समय पाठ्यक्रम के लिए मॉडल समारोह फिटिंग ऐसे mRNA गिरावट दर के रूप में मॉडल मापदंडों का सबसे अच्छा अनुमान पैदावार । एक प्रतिनिधि परिणाम के रूप में हम दो अलग-अलग लिपिड-आधारित ट्रांसफैक्शन एजेंटों की एमआरएनए वितरण दक्षता पर चर्चा करते हैं और उनके पैरामीटर वितरण कैसे भिन्न होते हैं।
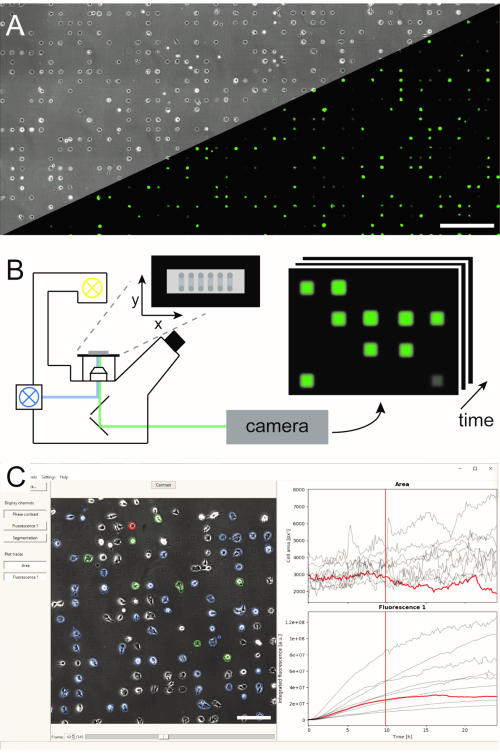
चित्रा 1:LISCA कार्यप्रवाह संयोजन का प्रतिनिधित्व (ए) सूक्ष्म पैटर्न वाले एकल-सेल सरणी (बी) स्कैनिंग टाइम-लैप्स माइक्रोस्कोपी और (सी) रिकॉर्ड की गई छवि श्रृंखला का स्वचालित छवि विश्लेषण। एकल-कोशिका सरणी में सेल-प्रतिरोधी इंटरस्पेस के साथ सेल-चिपकने वाले वर्गों का दो-आयामी पैटर्न होता है जिससे माइक्रोपैटर्न पर कोशिकाओं की व्यवस्था होती है, जैसा कि चरण-विपरीत छवि के साथ-साथ ईजीएफपी एक्सप्रेसिंग कोशिकाओं(ए)की फ्लोरेसेंस छवि में देखा जा सकता है। पूरे माइक्रोस्ट्रक्चर्ड क्षेत्र को स्कैनिंग टाइम-लैप्स मोड में चित्रित किया जाता है जो बार-बार स्थिति(बी)के अनुक्रम पर छवियों को ले जाता है। रिकॉर्ड की गई छवि श्रृंखला को समय के साथ प्रति सेल फ्लोरेसेंस तीव्रता (सी) को पढ़ने के लिए संसाधित कियाजाता है। स्केल बार: 500 माइक्रोन(ए),200 माइक्रोन(सी)। कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें ।
Protocol
Representative Results
Discussion
यहां हमने एल्ससीए को एकल-कोशिका स्तर पर इंट्रासेलुलर फ्लोरोसेंट लेबल के सेलुलर काइनेटिक्स का पालन करने के लिए एक बहुमुखी तकनीक के रूप में वर्णित किया। एक सफल LISCA प्रयोग करने के लिए, प्रोटोकॉल अनुभाग के …
Declarações
The authors have nothing to disclose.
Acknowledgements
इस काम को जर्मन साइंस फाउंडेशन (डीएफजी) से सहयोगी अनुसंधान केंद्र (एसएफबी) 1032 तक अनुदान द्वारा समर्थित किया गया था। सहकारी परियोजना 05K2018-2017-06716 मेडिसॉफ्ट के साथ-साथ बेयरिश फोर्स्टस्ट से अनुदान के साथ-साथ बेएरिसचे फोर्स्टिफांग से अनुदान के तहत जर्मन संघीय शिक्षा, अनुसंधान और प्रौद्योगिकी मंत्रालय (बीएमबीएफ) द्वारा समर्थन को कृतज्ञता से स्वीकार किया जाता है । अनीता रीसर को ग्रेजुएट स्कूल ऑफ क्वांटिटेटिव बायोसाइंसेज म्यूनिख (क्यूबीएम) के माध्यम से डीएफजी फेलोशिप द्वारा समर्थित किया गया था।
Materials
| Adtech Polymer Engineering PTFE Microtubing | Fisher Scientific | 10178071 | |
| baking oven | Binder | 9010-0190 | |
| CFI Plan Fluor DL 10x | Nikon | MRH20100 | |
| Desiccator | Roth | NX07.1 | |
| Eclipse Ti-E | Nikon | ||
| eGFP mRNA | Trilink | L-7601 | |
| Female Luer to Tube Connector | MEDNET | FTL210-6005 | |
| Fetal bovine serum | Thermo Fisher | 10270106 | |
| Fibronectin | Yo Proteins | 663 | |
| Filter set eGFP | AHF | F46-002 | |
| Fisherbrand Translucent Platinum-Cured Silicone Tubing | Fisher Scientific | 11768088 | |
| HEPES (1 M) | Thermo Fisher | 15630080 | |
| Incubation Box | Okolab | OKO-H201 | |
| incubator | Binder | 9040-0012 | |
| L-15 without phenol red | Thermo Fisher | 21083027 | |
| Lipofectamine 2000 | Thermo Fisher | 11668027 | |
| Male Luer | in-house fabricated consisting of teflon | ||
| Male Luer to Tube Connector | MEDNET | MTLS210-6005 | alternative to in-house fabricated male luers |
| NaCl (5 M) | Thermo Fisher | AM9760G | |
| Needleless Valve to Male Luer Connector | MEDNET | NVFMLLPC | |
| NIS Elements | Nikon | Imaging software Version 5.02.00 | |
| NOA81 | Thorlabs | NOA81 | Fast Curing Optical Adhesive for tube system assembly |
| Opti-MEM | Thermo Fisher | 31985062 | |
| PCO edge 4.2 M-USB-HQ-PCO | pco | ||
| Phosphate buffered saline (PBS) | in-house prepared | ||
| Plasma Cleaner | Diener Femto | Pico-BRS | |
| PLL(20 kDa)-g[3.5]-PEG(2 kDa) | SuSoS AG | ||
| silicon wafer mit mircorstructures | in-house fabricated | ||
| Sola Light Engine | Lumencor | ||
| sticky slide VI 0.4 | ibidi | 80608 | |
| Sylgard 184 Silicone Elastomer Kit | Dow Corning | 1673921 | |
| Tango 2 | Märzhäuser | 00-24-626-0000 | |
| Ultrapure water | in-house prepared | ||
| uncoated coverslips | ibidi | 10813 | |
| Injekt-F Solo, 1 mL | Omilab | 9166017V | with replacement sporn |
Referências
- Altschuler, S. J., Wu, L. F. Cellular heterogeneity: do differences make a difference. Cell. 141 (4), 559-563 (2010).
- Locke, J. C., Elowitz, M. B. Using movies to analyse gene circuit dynamics in single cells. Nature Reviews Microbiology. 7 (5), 383-392 (2009).
- Spencer, S. L., Gaudet, S., Albeck, J. G., Burke, J. M., Sorger, P. K. Non-genetic origins of cell-to-cell variability in TRAIL-induced apoptosis. Nature. 459 (7245), 428-432 (2009).
- El-Ali, J., Sorger, P. K., Jensen, K. F. Cells on chips. Nature. 442 (7101), 403-411 (2006).
- Segerer, F. J., et al. Versatile method to generate multiple types of micropatterns. Biointerphases. 11 (1), 011005 (2016).
- Piel, M., Théry, M. . Micropatterning in cell biology, part A/B/C. , (2014).
- Reiser, A., Zorn, M. L., Murschhauser, A., Rädler, J. O., Ertl, P., Rothbauer, M. . Cell-Based Microarrays: Methods and Protocols. , 41-54 (2018).
- Picone, R., Baum, B., McKendry, R. . Methods in cell biology. 119, 73-90 (2014).
- Reiser, A. . Single-cell time courses of mRNA transport and translation kinetics. , (2019).
- . Softmatter LMU-Raedler Group Available from: https://github.com/SoftmatterLMU-RaedlerGroup/pyama (2020)
- . MIAAB, 29262 Available from: https://github.com/SoftmatterLMU-RaedlerGroup/pyama (2020)
- Reiser, A., et al. Correlation of mRNA delivery timing and protein expression in lipid-based transfection. Integrative Biology. 11 (9), 362-371 (2019).
- Reiser, A., Woschée, D., Mehrotra, N., Krzysztoń, R. S., Strey, H. H., Rädler, J. O. Supplementing data and code for: “Correlation of mRNA delivery timing and protein expression in lipid-based transfection”. Zenodo. , (2019).
- Ferizi, M., et al. Stability analysis of chemically modified mRNA using micropattern-based single-cell arrays. Lab on a Chip. 15 (17), 3561-3571 (2015).
- Fröhlich, F., et al. Multi-experiment nonlinear mixed effect modeling of single-cell translation kinetics after transfection. NPJ systems biology and applications. 4 (1), 1-12 (2018).
- Krzysztoń, R., et al. Single-cell kinetics of siRNA-mediated mRNA degradation. Nanomedicine: Nanotechnology, Biology and Medicine. 21, 102077 (2019).
- Röttgermann, P. J., Alberola, A. P., Rädler, J. O. Cellular self-organization on micro-structured surfaces. Soft Matter. 10 (14), 2397-2404 (2014).
- Röttgermann, P. J., Dawson, K. A., Rädler, J. O. Time-resolved study of nanoparticle induced apoptosis using microfabricated single cell arrays. Microarrays. 5 (2), 8 (2016).
- Murschhauser, A., et al. A high-throughput microscopy method for single-cell analysis of event-time correlations in nanoparticle-induced cell death. Communications Biology. 2 (1), 1-11 (2019).
- Chatzopoulou, E. I., et al. Chip-based platform for dynamic analysis of NK cell cytolysis mediated by a triplebody. Analyst. 141 (7), 2284-2295 (2016).
- Sztilkovics, M., et al. Single-cell adhesion force kinetics of cell populations from combined label-free optical biosensor and robotic fluidic force microscopy. Scientific Reports. 10 (1), 1-13 (2020).

