単細胞アレイの生細胞イメージング(LISCA) - 細胞動態を定量化する多目的な技術
Summary
マイクロパターン配列を用いた単一細胞からの蛍光レポータータイムコースの取得方法を紹介する。このプロトコルは、単一細胞アレイの調製、ライブセルスキャンタイムラプス顕微鏡のセットアップと動作、および接着部位あたりの細胞統合蛍光時間コースの自動事前選択、視覚制御および追跡のためのオープンソース画像分析ツールを記述する。
Abstract
単一細胞アレイ(LISCA)のライブセルイメージングは、高スループットで個々の細胞からの蛍光信号の時間コースを収集するための汎用性の高い方法です。一般に、培養細胞からの単細胞時間コースの獲得は、細胞の運動性および細胞形状の多様性によって妨げられる。接着剤マイクロアレイは、単一細胞条件を標準化し、画像解析を容易にします。LISCAは、単一細胞マイクロアレイと、走査時間経過顕微鏡と自動画像処理を組み合わせたものです。ここでは、単細胞蛍光時間コースをLISCA形式で取る実験ステップについて説明する。また、強化された緑色蛍光タンパク質(eGFP)用のmRNAコードを用いて微小パターン配列に付着した細胞をトランスフェクトし、走査時間経過顕微鏡を介して数百個の細胞のeGFP発現動態を並行して監視します。画像データスタックは、選択した細胞の輪郭に蛍光強度を統合して単一細胞蛍光時間コースを生成する新開発のソフトウェアによって自動的に処理されます。mRNAトランスフェクション後のeGFP発現時間コースは、mRNAの発現および分解率を明らかにする単純な運動翻訳モデルによって十分に記述されていることを実証する。アポトーシスのシグナル伝達の文脈における複数マーカーのイベント時間相関に対するLISCAのさらなる応用が議論される。
Introduction
近年、単細胞実験の重要性が明らかになってきた。単一細胞からのデータは、細胞間変動、細胞内パラメータ相関の分解能、およびアンサンブル測定1、2、3に隠されたままの細胞動態の検出の調査を可能にする。数千個の単一細胞の細胞動態を並行して調べるには、数時間から数日間の期間にわたって標準化された条件下で細胞を監視し、定量的なデータ分析4を行う新しいアプローチが必要である。ここでは、マイクロ構造化アレイとタイムラプス顕微鏡と自動画像解析を組み合わせた単一細胞アレイ(LISCA)のライブセルイメージングを紹介します。
マイクロ構造化単一細胞アレイを生成するためのいくつかの方法が確立され、文献5,6に掲載されています。ここでは、マイクロスケールのプラズマ開始タンパク質パターニング(μPIPP)について簡単に説明します。μPIPPを用いた単一セルアレイ製作の詳細なプロトコルは、リファレンス7にも記載されています。単一細胞配列を用いると、各細胞に対して定義された微小環境を提示する標準化された接着スポットに数千個の細胞を整列させ、実験変動の原因を減少させる(図 1A)。単一細胞アレイは、さまざまな細胞プロセスを示すために目的とする蛍光マーカーの時間コースを監視するために使用されます。スキャンタイムラプスモードにおける長期顕微鏡検査により、単一細胞アレイの広い領域を監視し、数時間または数日の観察時間にわたってハイスループットで単一細胞データをサンプリングすることができます。これにより、配列の各位置から画像の時系列スタックが生成されます(図1B)。大量の画像データを削減し、望ましい単一細胞蛍光時間コースを効率的に抽出するためには、細胞の位置決めを利用した自動画像処理が必要である(図1C)。
LISCAの課題は、実験プロトコルと計算ツールを適応させて、細胞動態の定量的で再現可能なデータを生成するハイスループットアッセイを形成することです。本稿では、個々の方法と、それらをLISCAアッセイでどのように組み合わせるかについて、ステップバイステップで説明します。一例として、人工mRNA送達後の強化された緑色蛍光タンパク質(eGFP)発現の経時経過について考察する。mRNA送達後のeGFP発現は、mRNAの翻訳および分解をモデル化した反応速度式によって説明される。eGFP濃度の時間経過に対するモデル関数を、時間の経過とともに個々の細胞ごとに蛍光強度のLISCA読み出しに適合させることで、mRNA分解率などのモデルパラメータの最良の推定値が得られます。代表的な結果として、2種類の異なる脂質系トランスフェクション剤のmRNA送達効率と、そのパラメータ分布の違いについて考察する。
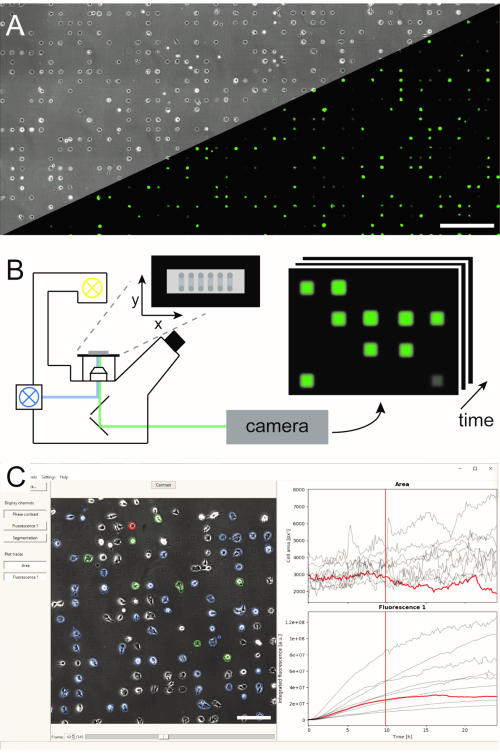
図1:(A)マイクロパターン単細胞アレイ(B)走査時間経過顕微鏡と(C)記録画像系列の自動画像解析を組み合わせたLISCAワークフローの表現単細胞アレイは、マイクロパターン上の細胞の配置につながる細胞忌避的な空間を有する細胞接着性の二次元パターンからなるが、相コントラスト画像ならびにeGFP発現細胞の蛍光像(A)に見られるように。マイクロ構造領域全体が、一連の位置(B)で繰り返し画像を撮影するスキャンタイムラプスモードで画像化されます。記録された画像シリーズは、時間の経過に関する細胞あたりの蛍光強度を読み出すために処理される(C)。スケールバー:500 μm(A)、200μm(C)この図の大きなバージョンを表示するには、ここをクリックしてください。
Protocol
Representative Results
Discussion
ここでは、単細胞レベルで細胞内蛍光標識の細胞動態に従う多目的技術としてLISCAを説明した。LISCA実験を成功させるためには、プロトコルセクションの各ステップを個別に確立し、すべてのステップを組み合わせる必要があります。LISCAの3つの主要な側面のそれぞれは、重要なステップを備えています。
単一細胞マイクロアレイ製造
マイクロアレイの細胞の位…
Declarações
The authors have nothing to disclose.
Acknowledgements
この研究は、ドイツ科学財団(DFG)から共同研究センター(SFB)1032への助成金によって支援されました。ドイツ連邦教育研究技術省(BMBF)による協力プロジェクト05K2018-2017-06716メディソフトの支援とバイエリッシェ・フォルシュングスシュティフトゥンからの助成金は感謝しています。アニタ・ライナーは、ミュンヘン(QBM)の定量バイオサイエンス大学院を通じてDFGフェローシップによって支援されました。
Materials
| Adtech Polymer Engineering PTFE Microtubing | Fisher Scientific | 10178071 | |
| baking oven | Binder | 9010-0190 | |
| CFI Plan Fluor DL 10x | Nikon | MRH20100 | |
| Desiccator | Roth | NX07.1 | |
| Eclipse Ti-E | Nikon | ||
| eGFP mRNA | Trilink | L-7601 | |
| Female Luer to Tube Connector | MEDNET | FTL210-6005 | |
| Fetal bovine serum | Thermo Fisher | 10270106 | |
| Fibronectin | Yo Proteins | 663 | |
| Filter set eGFP | AHF | F46-002 | |
| Fisherbrand Translucent Platinum-Cured Silicone Tubing | Fisher Scientific | 11768088 | |
| HEPES (1 M) | Thermo Fisher | 15630080 | |
| Incubation Box | Okolab | OKO-H201 | |
| incubator | Binder | 9040-0012 | |
| L-15 without phenol red | Thermo Fisher | 21083027 | |
| Lipofectamine 2000 | Thermo Fisher | 11668027 | |
| Male Luer | in-house fabricated consisting of teflon | ||
| Male Luer to Tube Connector | MEDNET | MTLS210-6005 | alternative to in-house fabricated male luers |
| NaCl (5 M) | Thermo Fisher | AM9760G | |
| Needleless Valve to Male Luer Connector | MEDNET | NVFMLLPC | |
| NIS Elements | Nikon | Imaging software Version 5.02.00 | |
| NOA81 | Thorlabs | NOA81 | Fast Curing Optical Adhesive for tube system assembly |
| Opti-MEM | Thermo Fisher | 31985062 | |
| PCO edge 4.2 M-USB-HQ-PCO | pco | ||
| Phosphate buffered saline (PBS) | in-house prepared | ||
| Plasma Cleaner | Diener Femto | Pico-BRS | |
| PLL(20 kDa)-g[3.5]-PEG(2 kDa) | SuSoS AG | ||
| silicon wafer mit mircorstructures | in-house fabricated | ||
| Sola Light Engine | Lumencor | ||
| sticky slide VI 0.4 | ibidi | 80608 | |
| Sylgard 184 Silicone Elastomer Kit | Dow Corning | 1673921 | |
| Tango 2 | Märzhäuser | 00-24-626-0000 | |
| Ultrapure water | in-house prepared | ||
| uncoated coverslips | ibidi | 10813 | |
| Injekt-F Solo, 1 mL | Omilab | 9166017V | with replacement sporn |
Referências
- Altschuler, S. J., Wu, L. F. Cellular heterogeneity: do differences make a difference. Cell. 141 (4), 559-563 (2010).
- Locke, J. C., Elowitz, M. B. Using movies to analyse gene circuit dynamics in single cells. Nature Reviews Microbiology. 7 (5), 383-392 (2009).
- Spencer, S. L., Gaudet, S., Albeck, J. G., Burke, J. M., Sorger, P. K. Non-genetic origins of cell-to-cell variability in TRAIL-induced apoptosis. Nature. 459 (7245), 428-432 (2009).
- El-Ali, J., Sorger, P. K., Jensen, K. F. Cells on chips. Nature. 442 (7101), 403-411 (2006).
- Segerer, F. J., et al. Versatile method to generate multiple types of micropatterns. Biointerphases. 11 (1), 011005 (2016).
- Piel, M., Théry, M. . Micropatterning in cell biology, part A/B/C. , (2014).
- Reiser, A., Zorn, M. L., Murschhauser, A., Rädler, J. O., Ertl, P., Rothbauer, M. . Cell-Based Microarrays: Methods and Protocols. , 41-54 (2018).
- Picone, R., Baum, B., McKendry, R. . Methods in cell biology. 119, 73-90 (2014).
- Reiser, A. . Single-cell time courses of mRNA transport and translation kinetics. , (2019).
- . Softmatter LMU-Raedler Group Available from: https://github.com/SoftmatterLMU-RaedlerGroup/pyama (2020)
- . MIAAB, 29262 Available from: https://github.com/SoftmatterLMU-RaedlerGroup/pyama (2020)
- Reiser, A., et al. Correlation of mRNA delivery timing and protein expression in lipid-based transfection. Integrative Biology. 11 (9), 362-371 (2019).
- Reiser, A., Woschée, D., Mehrotra, N., Krzysztoń, R. S., Strey, H. H., Rädler, J. O. Supplementing data and code for: “Correlation of mRNA delivery timing and protein expression in lipid-based transfection”. Zenodo. , (2019).
- Ferizi, M., et al. Stability analysis of chemically modified mRNA using micropattern-based single-cell arrays. Lab on a Chip. 15 (17), 3561-3571 (2015).
- Fröhlich, F., et al. Multi-experiment nonlinear mixed effect modeling of single-cell translation kinetics after transfection. NPJ systems biology and applications. 4 (1), 1-12 (2018).
- Krzysztoń, R., et al. Single-cell kinetics of siRNA-mediated mRNA degradation. Nanomedicine: Nanotechnology, Biology and Medicine. 21, 102077 (2019).
- Röttgermann, P. J., Alberola, A. P., Rädler, J. O. Cellular self-organization on micro-structured surfaces. Soft Matter. 10 (14), 2397-2404 (2014).
- Röttgermann, P. J., Dawson, K. A., Rädler, J. O. Time-resolved study of nanoparticle induced apoptosis using microfabricated single cell arrays. Microarrays. 5 (2), 8 (2016).
- Murschhauser, A., et al. A high-throughput microscopy method for single-cell analysis of event-time correlations in nanoparticle-induced cell death. Communications Biology. 2 (1), 1-11 (2019).
- Chatzopoulou, E. I., et al. Chip-based platform for dynamic analysis of NK cell cytolysis mediated by a triplebody. Analyst. 141 (7), 2284-2295 (2016).
- Sztilkovics, M., et al. Single-cell adhesion force kinetics of cell populations from combined label-free optical biosensor and robotic fluidic force microscopy. Scientific Reports. 10 (1), 1-13 (2020).

