Imagem de células vivas de matrizes unicelulares (LISCA) - uma técnica versátil para quantificar cinética celular
Summary
Apresentamos um método para a aquisição de cursos de tempo de repórter fluorescência de células únicas usando matrizes micropatteradas. O protocolo descreve a preparação de matrizes unicelulares, a configuração e o funcionamento de microscopia de lapso de tempo de varredura de células vivas e uma ferramenta de análise de imagem de código aberto para pré-seleção automatizada, controle visual e rastreamento de cursos de tempo de fluorescência integrados por células por local de adesão.
Abstract
A imagem de células vivas de Matrizes Unicelulares (LISCA) é um método versátil para coletar cursos de tempo de sinais de fluorescência de células individuais em alta produtividade. Em geral, a aquisição de cursos de tempo unicelulares de células cultivadas é dificultada pela motilidade celular e diversidade de formas celulares. Micro arrays adesivos padronizam as condições unicelulares e facilitam a análise de imagens. O LISCA combina microarrays unicelulares com microscopia de lapso de tempo de varredura e processamento automatizado de imagens. Aqui, descrevemos os passos experimentais de fazer cursos de tempo de fluorescência unicelulares em um formato LISCA. Transfectamos células aderentes a uma matriz micropatterada usando codificação mRNA para proteína fluorescente verde aprimorada (eGFP) e monitoramos a cinética de expressão eGFP de centenas de células em paralelo através da microscopia de lapso de tempo de varredura. As pilhas de dados de imagem são automaticamente processadas por um software recém-desenvolvido que integra a intensidade da fluorescência sobre contornos celulares selecionados para gerar cursos de tempo de fluorescência unicelulares. Demonstramos que os cursos de tempo de expressão do eGFP após a transfecção do mRNA são bem descritos por um simples modelo de tradução cinética que revela taxas de expressão e degradação do mRNA. Outras aplicações do LISCA para correlações de tempo de evento de múltiplos marcadores no contexto de sinalização da apoptose são discutidas.
Introduction
Nos últimos anos, a importância de experimentos unicelulares tornou-se aparente. Os dados de células únicas permitem a investigação da variabilidade celular-célula, a resolução de correlações de parâmetros intracelulares e a detecção de cinéticas celulares que permanecem escondidas nas medições do conjunto1,2,3. Para investigar cinética celular de milhares de células únicas em paralelo, são necessárias novas abordagens que permitam monitorar as células em condições padronizadas durante um período de várias horas até vários dias seguido de uma análise quantitativa de dados 4. Aqui, apresentamos imagens de células vivas de matrizes unicelulares (LISCA), que combina o uso de matrizes microestruturadas com microscopia de lapso de tempo e análise automatizada de imagens.
Vários métodos de geração de matrizes unicelulares microestruturadas foram estabelecidos e publicados na literatura5,6. Aqui, descrevemos brevemente a padronização de proteína iniciada por plasma de microescala (μPIPP). Um protocolo detalhado da fabricação de matriz unicelular usando μPIPP também é encontrado na referência7. O uso de matrizes unicelulares permite o alinhamento de milhares de células em pontos de adesão padronizados apresentando microambientes definidos para cada célula e, assim, reduz uma fonte de variabilidade experimental(Figura 1A). Matrizes unicelulares são usadas para monitorar os cursos de tempo de marcadores fluorescentes com propósito para indicar uma variedade de processos celulares. A microscopia de longo prazo no modo de verificação de lapso de tempo permite monitorar uma grande área das matrizes unicelulares e, portanto, amostrar dados unicelulares em alta taxa de distribuição durante um tempo de observação de várias horas ou até dias. Isso gera pilhas de imagens de linha de tempo de cada posição da matriz(Figura 1B). Para reduzir a grande quantidade de dados de imagem e extrair os cursos de tempo de fluorescência unicelular desejados de forma eficiente, é necessário um processamento automatizado de imagens que aproveita o posicionamento das células(Figura 1C).
O desafio do LISCA é adaptar os protocolos experimentais e ferramentas computacionais para formar um ensaio de alto rendimento que gere dados quantitativos e reprodutíveis de cinética celular. Neste artigo, fornecemos uma descrição passo a passo dos métodos individuais e como eles são combinados em um ensaio LISCA. Como exemplo, discutimos o curso temporal da expressão aprimorada de proteína fluorescente verde (eGFP) após a entrega artificial de mRNA. A expressão eGFP após a entrega do mRNA é descrita por equações de taxa de reação modelando tradução e degradação do mRNA. A montagem da função do modelo para o curso de tempo da concentração eGFP à leitura LISCA da intensidade de fluorescência para cada célula individual ao longo do tempo produz melhores estimativas de parâmetros de modelo, como a taxa de degradação do mRNA. Como resultado representativo, discutimos a eficiência de entrega de mRNA de dois diferentes agentes de transfecção à base de lipídios e como suas distribuições de parâmetros diferem.
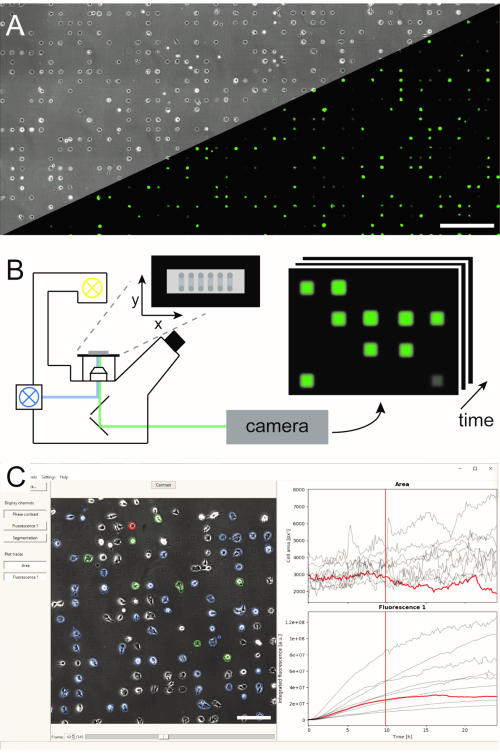
Figura 1: Representação do fluxo de trabalho LISCA que combina (A) matrizes unicelulares (B) micro-padronizadas de micro-padrão e análise automatizada de imagens (C) da série de imagens gravadas. As matrizes unicelulares consistem em um padrão bidimensional de quadrados adesivos celulares com um interespaço repelente celular levando a um arranjo das células no micropattern, como pode ser visto na imagem de contraste de fase, bem como na imagem de fluorescência das células expressas eGFP(A). Toda a área microestruturada é imagem em um modo de fluxo de tempo de varredura repetidamente tirando imagens em uma sequência de posições(B). Séries de imagens gravadas são processadas para ler a intensidade da fluorescência por célula ao longo do tempo(C). Barras de escala: 500 μm(A),200 μm(C). Clique aqui para ver uma versão maior desta figura.
Protocol
Representative Results
Discussion
Aqui descrevemos o LISCA como uma técnica versátil para seguir cinética celular de rótulos fluorescentes intracelulares no nível de célula única. Para realizar um experimento LISCA bem-sucedido, cada uma das etapas descritas da seção de protocolo deve ser estabelecida individualmente e, em seguida, todas as etapas devem ser combinadas. Cada um dos três principais aspectos do LISCA apresentam etapas cruciais.
Fabricação de microarraia unicelular
A qualidade da microarray…
Declarações
The authors have nothing to disclose.
Acknowledgements
Este trabalho foi apoiado por bolsas da Fundação Alemã de Ciência (DFG) ao Centro de Pesquisa Colaborativa (SFB) 1032. Apoio do Ministério Federal alemão da Educação, Pesquisa e Tecnologia (BMBF) no âmbito do projeto cooperativo 05K2018-2017-06716 Medisoft, bem como uma bolsa da Bayerische Forschungsstiftung são reconhecidas com gratidão. Anita Reiser foi apoiada por uma Bolsa DFG através da Escola de Pós-Graduação em Biociências Quantitativas de Munique (QBM).
Materials
| Adtech Polymer Engineering PTFE Microtubing | Fisher Scientific | 10178071 | |
| baking oven | Binder | 9010-0190 | |
| CFI Plan Fluor DL 10x | Nikon | MRH20100 | |
| Desiccator | Roth | NX07.1 | |
| Eclipse Ti-E | Nikon | ||
| eGFP mRNA | Trilink | L-7601 | |
| Female Luer to Tube Connector | MEDNET | FTL210-6005 | |
| Fetal bovine serum | Thermo Fisher | 10270106 | |
| Fibronectin | Yo Proteins | 663 | |
| Filter set eGFP | AHF | F46-002 | |
| Fisherbrand Translucent Platinum-Cured Silicone Tubing | Fisher Scientific | 11768088 | |
| HEPES (1 M) | Thermo Fisher | 15630080 | |
| Incubation Box | Okolab | OKO-H201 | |
| incubator | Binder | 9040-0012 | |
| L-15 without phenol red | Thermo Fisher | 21083027 | |
| Lipofectamine 2000 | Thermo Fisher | 11668027 | |
| Male Luer | in-house fabricated consisting of teflon | ||
| Male Luer to Tube Connector | MEDNET | MTLS210-6005 | alternative to in-house fabricated male luers |
| NaCl (5 M) | Thermo Fisher | AM9760G | |
| Needleless Valve to Male Luer Connector | MEDNET | NVFMLLPC | |
| NIS Elements | Nikon | Imaging software Version 5.02.00 | |
| NOA81 | Thorlabs | NOA81 | Fast Curing Optical Adhesive for tube system assembly |
| Opti-MEM | Thermo Fisher | 31985062 | |
| PCO edge 4.2 M-USB-HQ-PCO | pco | ||
| Phosphate buffered saline (PBS) | in-house prepared | ||
| Plasma Cleaner | Diener Femto | Pico-BRS | |
| PLL(20 kDa)-g[3.5]-PEG(2 kDa) | SuSoS AG | ||
| silicon wafer mit mircorstructures | in-house fabricated | ||
| Sola Light Engine | Lumencor | ||
| sticky slide VI 0.4 | ibidi | 80608 | |
| Sylgard 184 Silicone Elastomer Kit | Dow Corning | 1673921 | |
| Tango 2 | Märzhäuser | 00-24-626-0000 | |
| Ultrapure water | in-house prepared | ||
| uncoated coverslips | ibidi | 10813 | |
| Injekt-F Solo, 1 mL | Omilab | 9166017V | with replacement sporn |
Referências
- Altschuler, S. J., Wu, L. F. Cellular heterogeneity: do differences make a difference. Cell. 141 (4), 559-563 (2010).
- Locke, J. C., Elowitz, M. B. Using movies to analyse gene circuit dynamics in single cells. Nature Reviews Microbiology. 7 (5), 383-392 (2009).
- Spencer, S. L., Gaudet, S., Albeck, J. G., Burke, J. M., Sorger, P. K. Non-genetic origins of cell-to-cell variability in TRAIL-induced apoptosis. Nature. 459 (7245), 428-432 (2009).
- El-Ali, J., Sorger, P. K., Jensen, K. F. Cells on chips. Nature. 442 (7101), 403-411 (2006).
- Segerer, F. J., et al. Versatile method to generate multiple types of micropatterns. Biointerphases. 11 (1), 011005 (2016).
- Piel, M., Théry, M. . Micropatterning in cell biology, part A/B/C. , (2014).
- Reiser, A., Zorn, M. L., Murschhauser, A., Rädler, J. O., Ertl, P., Rothbauer, M. . Cell-Based Microarrays: Methods and Protocols. , 41-54 (2018).
- Picone, R., Baum, B., McKendry, R. . Methods in cell biology. 119, 73-90 (2014).
- Reiser, A. . Single-cell time courses of mRNA transport and translation kinetics. , (2019).
- . Softmatter LMU-Raedler Group Available from: https://github.com/SoftmatterLMU-RaedlerGroup/pyama (2020)
- . MIAAB, 29262 Available from: https://github.com/SoftmatterLMU-RaedlerGroup/pyama (2020)
- Reiser, A., et al. Correlation of mRNA delivery timing and protein expression in lipid-based transfection. Integrative Biology. 11 (9), 362-371 (2019).
- Reiser, A., Woschée, D., Mehrotra, N., Krzysztoń, R. S., Strey, H. H., Rädler, J. O. Supplementing data and code for: “Correlation of mRNA delivery timing and protein expression in lipid-based transfection”. Zenodo. , (2019).
- Ferizi, M., et al. Stability analysis of chemically modified mRNA using micropattern-based single-cell arrays. Lab on a Chip. 15 (17), 3561-3571 (2015).
- Fröhlich, F., et al. Multi-experiment nonlinear mixed effect modeling of single-cell translation kinetics after transfection. NPJ systems biology and applications. 4 (1), 1-12 (2018).
- Krzysztoń, R., et al. Single-cell kinetics of siRNA-mediated mRNA degradation. Nanomedicine: Nanotechnology, Biology and Medicine. 21, 102077 (2019).
- Röttgermann, P. J., Alberola, A. P., Rädler, J. O. Cellular self-organization on micro-structured surfaces. Soft Matter. 10 (14), 2397-2404 (2014).
- Röttgermann, P. J., Dawson, K. A., Rädler, J. O. Time-resolved study of nanoparticle induced apoptosis using microfabricated single cell arrays. Microarrays. 5 (2), 8 (2016).
- Murschhauser, A., et al. A high-throughput microscopy method for single-cell analysis of event-time correlations in nanoparticle-induced cell death. Communications Biology. 2 (1), 1-11 (2019).
- Chatzopoulou, E. I., et al. Chip-based platform for dynamic analysis of NK cell cytolysis mediated by a triplebody. Analyst. 141 (7), 2284-2295 (2016).
- Sztilkovics, M., et al. Single-cell adhesion force kinetics of cell populations from combined label-free optical biosensor and robotic fluidic force microscopy. Scientific Reports. 10 (1), 1-13 (2020).

