Imágenes de células vivas de matrices unicelulares (LISCA) - una técnica versátil para cuantificar la cinética celular
Summary
Presentamos un método para la adquisición de cursos de tiempo reportero de fluorescencia de células individuales utilizando matrices micropatterned. El protocolo describe la preparación de matrices unicelulares, la configuración y el funcionamiento de la microscopía de lapso de tiempo de barrido de células vivas y una herramienta de análisis de imágenes de código abierto para la preselección automatizada, el control visual y el seguimiento de los cursos de tiempo de fluorescencia integrados en células por sitio de adhesión.
Abstract
Imágenes de células vivas de matrices unicelulares (LISCA) es un método versátil para recopilar cursos de tiempo de señales de fluorescencia de células individuales en alto rendimiento. En general, la adquisición de cursos de tiempo unicelulares de células cultivadas se ve obstaculizada por la motilidad celular y la diversidad de formas celulares. Las micro-matrices adhesivas estandarizan las condiciones unicelulares y facilitan el análisis de imágenes. LISCA combina microarrays unicelulares con microscopía de lapso de tiempo de barrido y procesamiento automatizado de imágenes. Aquí, se describen los pasos experimentales de tomar cursos de tiempo de fluorescencia unicelular en un formato LISCA. Transfectamos las células adherentes a una matriz micropatterned utilizando la codificación de ARNm para la proteína fluorescente verde mejorada (eGFP) y supervisar la cinética de expresión de eGFP de cientos de células en paralelo a través de microscopía de lapso de tiempo de barrido. Las pilas de datos de imagen son procesadas automáticamente por un software recientemente desarrollado que integra la intensidad de fluorescencia sobre los contornos celulares seleccionados para generar cursos de tiempo de fluorescencia unicelulares. Demostramos que los cursos de tiempo de expresión de eGFP después de la transfección de ARNm están bien descritos por un modelo de traducción cinética simple que revela la expresión y las tasas de degradación de ARNm. Otros usos de LISCA para las correlaciones del tiempo del acontecimiento de marcadores múltiples en el contexto del apoptosis de la señalización se discuten.
Introduction
En los últimos años, la importancia de los experimentos unicelulares se ha hecho evidente. Los datos de células individuales permiten la investigación de la variabilidad célula a célula, la resolución de correlaciones de parámetros intracelulares y la detección de cinéticas celulares que permanecen ocultas en las mediciones deconjuntos 1,2,3. Con el fin de investigar la cinética celular de miles de células individuales en paralelo, se necesitan nuevos enfoques que permitan el monitoreo de las células en condiciones estandarizadas durante un período de tiempo de varias horas hasta varios días seguido de un análisis cuantitativo de datos 4. Aquí, presentamos imágenes de células vivas de matrices unicelulares (LISCA), que combina el uso de matrices microestructuradas con microscopía de lapso de tiempo y análisis de imágenes automatizado.
Se han establecido y publicado en la literatura5,6. Aquí, describimos brevemente microescala plasma-iniciado patrón de proteínas (μPIPP). Un protocolo detallado de la fabricación de matrices unicelulares utilizando μPIPP también se encuentra en la referencia7. El uso de matrices unicelulares permite la alineación de miles de células en puntos de adhesión estandarizados que presentan microambientes definidos para cada célula y, por lo tanto, reduce una fuente de variabilidad experimental (Figura 1A). Las matrices unicelulares se utilizan para monitorear los cursos de tiempo de los marcadores fluorescentes con el propósito de indicar una variedad de procesos celulares. La microscopía a largo plazo en el modo de lapso de tiempo de barrido permite monitorear una gran área de las matrices unicelulares y, por lo tanto, tomar muestras de datos unicelulares en alto rendimiento durante un tiempo de observación de varias horas o incluso días. Esto genera pilas de línea de tiempo de imágenes de cada posición de la matriz (Figura 1B). Con el fin de reducir la gran cantidad de datos de imagen y extraer los cursos de tiempo de fluorescencia unicelular deseados de una manera eficiente, se requiere un procesamiento automatizado de imágenes que aproveche el posicionamiento de las células (Figura 1C).
El reto de LISCA es adaptar los protocolos experimentales y las herramientas computacionales para formar un ensayo de alto rendimiento que genere datos cuantitativos y reproducibles de cinética celular. En este artículo proporcionamos una descripción paso a paso de los métodos individuales y cómo se combinan en un ensayo LISCA. Como ejemplo, discutimos el curso del tiempo de la expresión mejorada de la proteína fluorescente verde (eGFP) después de la entrega artificial del mRNA. La expresión del eGFP que sigue entrega del mRNA es descrita por las ecuaciones de la tarifa de reacción que modelan la traducción y la degradación del mRNA. Ajustar la función del modelo para el curso temporal de la concentración de eGFP a la lectura LISCA de la intensidad de fluorescencia para cada célula individual a lo largo del tiempo produce las mejores estimaciones de los parámetros del modelo, como la tasa de degradación del ARNm. Como resultado representativo discutimos la eficacia de la entrega del mRNA de dos diversos agentes lípido-basados de la transfección y cómo sus distribuciones del parámetro diferencian.
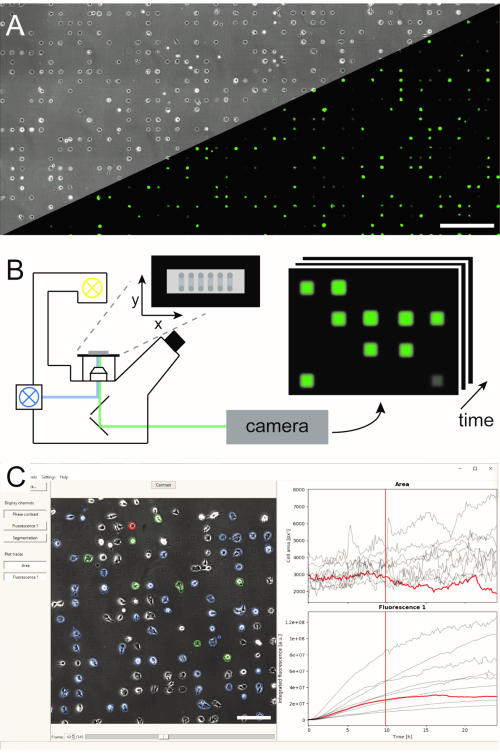
Figura 1:Representación del flujo de trabajo lisca combinando (A) micro-patrones de matrices unicelulares (B) microscopía de lapso de tiempo de barrido y (C) análisis automatizado de imágenes de series de imágenes grabadas. Las matrices unicelulares consisten en un patrón bidimensional de cuadrados adhesivos celulares con un interespacio repelente celular que conduce a una disposición de las células en el micropatrón, como se puede ver en la imagen de contraste de fase, así como la imagen de fluorescencia de las células que expresan eGFP(A). Toda el área microestructurada se toma una imagen en un modo de lapso de tiempo de escaneo tomando repetidamente imágenes en una secuencia de posiciones (B). Las series de imágenes grabadas se procesan para leer la intensidad de fluorescencia por célula a lo largo del tiempo(C). Barras de escala: 500 μm (A), 200 μm (C). Haga clic aquí para ver una versión más amplia de esta figura.
Protocol
Representative Results
Discussion
Aquí describimos LISCA como una técnica versátil para seguir la cinética celular de las etiquetas fluorescentes intracelulares a nivel unicelular. Para realizar un experimento LISCA exitoso, cada uno de los pasos descritos de la sección de protocolo debe establecerse individualmente y, a continuación, se deben combinar todos los pasos. Cada uno de los tres aspectos principales de LISCA cuenta con pasos cruciales.
Fabricación de microarrays unicelulares
La calidad del microar…
Declarações
The authors have nothing to disclose.
Acknowledgements
Este trabajo fue apoyado por subvenciones de la Fundación Alemana de la Ciencia (DFG) al Centro de Investigación Colaborativa (SFB) 1032. El apoyo del Ministerio Federal alemán de Educación, Investigación y Tecnología (BMBF) en el marco del proyecto cooperativo 05K2018-2017-06716 Medisoft, así como una subvención de la Bayerische Forschungsstiftung son agradecidos. Anita Reiser recibió el apoyo de una beca DFG a través de la Graduate School of Quantitative Biosciences Munich (QBM).
Materials
| Adtech Polymer Engineering PTFE Microtubing | Fisher Scientific | 10178071 | |
| baking oven | Binder | 9010-0190 | |
| CFI Plan Fluor DL 10x | Nikon | MRH20100 | |
| Desiccator | Roth | NX07.1 | |
| Eclipse Ti-E | Nikon | ||
| eGFP mRNA | Trilink | L-7601 | |
| Female Luer to Tube Connector | MEDNET | FTL210-6005 | |
| Fetal bovine serum | Thermo Fisher | 10270106 | |
| Fibronectin | Yo Proteins | 663 | |
| Filter set eGFP | AHF | F46-002 | |
| Fisherbrand Translucent Platinum-Cured Silicone Tubing | Fisher Scientific | 11768088 | |
| HEPES (1 M) | Thermo Fisher | 15630080 | |
| Incubation Box | Okolab | OKO-H201 | |
| incubator | Binder | 9040-0012 | |
| L-15 without phenol red | Thermo Fisher | 21083027 | |
| Lipofectamine 2000 | Thermo Fisher | 11668027 | |
| Male Luer | in-house fabricated consisting of teflon | ||
| Male Luer to Tube Connector | MEDNET | MTLS210-6005 | alternative to in-house fabricated male luers |
| NaCl (5 M) | Thermo Fisher | AM9760G | |
| Needleless Valve to Male Luer Connector | MEDNET | NVFMLLPC | |
| NIS Elements | Nikon | Imaging software Version 5.02.00 | |
| NOA81 | Thorlabs | NOA81 | Fast Curing Optical Adhesive for tube system assembly |
| Opti-MEM | Thermo Fisher | 31985062 | |
| PCO edge 4.2 M-USB-HQ-PCO | pco | ||
| Phosphate buffered saline (PBS) | in-house prepared | ||
| Plasma Cleaner | Diener Femto | Pico-BRS | |
| PLL(20 kDa)-g[3.5]-PEG(2 kDa) | SuSoS AG | ||
| silicon wafer mit mircorstructures | in-house fabricated | ||
| Sola Light Engine | Lumencor | ||
| sticky slide VI 0.4 | ibidi | 80608 | |
| Sylgard 184 Silicone Elastomer Kit | Dow Corning | 1673921 | |
| Tango 2 | Märzhäuser | 00-24-626-0000 | |
| Ultrapure water | in-house prepared | ||
| uncoated coverslips | ibidi | 10813 | |
| Injekt-F Solo, 1 mL | Omilab | 9166017V | with replacement sporn |
Referências
- Altschuler, S. J., Wu, L. F. Cellular heterogeneity: do differences make a difference. Cell. 141 (4), 559-563 (2010).
- Locke, J. C., Elowitz, M. B. Using movies to analyse gene circuit dynamics in single cells. Nature Reviews Microbiology. 7 (5), 383-392 (2009).
- Spencer, S. L., Gaudet, S., Albeck, J. G., Burke, J. M., Sorger, P. K. Non-genetic origins of cell-to-cell variability in TRAIL-induced apoptosis. Nature. 459 (7245), 428-432 (2009).
- El-Ali, J., Sorger, P. K., Jensen, K. F. Cells on chips. Nature. 442 (7101), 403-411 (2006).
- Segerer, F. J., et al. Versatile method to generate multiple types of micropatterns. Biointerphases. 11 (1), 011005 (2016).
- Piel, M., Théry, M. . Micropatterning in cell biology, part A/B/C. , (2014).
- Reiser, A., Zorn, M. L., Murschhauser, A., Rädler, J. O., Ertl, P., Rothbauer, M. . Cell-Based Microarrays: Methods and Protocols. , 41-54 (2018).
- Picone, R., Baum, B., McKendry, R. . Methods in cell biology. 119, 73-90 (2014).
- Reiser, A. . Single-cell time courses of mRNA transport and translation kinetics. , (2019).
- . Softmatter LMU-Raedler Group Available from: https://github.com/SoftmatterLMU-RaedlerGroup/pyama (2020)
- . MIAAB, 29262 Available from: https://github.com/SoftmatterLMU-RaedlerGroup/pyama (2020)
- Reiser, A., et al. Correlation of mRNA delivery timing and protein expression in lipid-based transfection. Integrative Biology. 11 (9), 362-371 (2019).
- Reiser, A., Woschée, D., Mehrotra, N., Krzysztoń, R. S., Strey, H. H., Rädler, J. O. Supplementing data and code for: “Correlation of mRNA delivery timing and protein expression in lipid-based transfection”. Zenodo. , (2019).
- Ferizi, M., et al. Stability analysis of chemically modified mRNA using micropattern-based single-cell arrays. Lab on a Chip. 15 (17), 3561-3571 (2015).
- Fröhlich, F., et al. Multi-experiment nonlinear mixed effect modeling of single-cell translation kinetics after transfection. NPJ systems biology and applications. 4 (1), 1-12 (2018).
- Krzysztoń, R., et al. Single-cell kinetics of siRNA-mediated mRNA degradation. Nanomedicine: Nanotechnology, Biology and Medicine. 21, 102077 (2019).
- Röttgermann, P. J., Alberola, A. P., Rädler, J. O. Cellular self-organization on micro-structured surfaces. Soft Matter. 10 (14), 2397-2404 (2014).
- Röttgermann, P. J., Dawson, K. A., Rädler, J. O. Time-resolved study of nanoparticle induced apoptosis using microfabricated single cell arrays. Microarrays. 5 (2), 8 (2016).
- Murschhauser, A., et al. A high-throughput microscopy method for single-cell analysis of event-time correlations in nanoparticle-induced cell death. Communications Biology. 2 (1), 1-11 (2019).
- Chatzopoulou, E. I., et al. Chip-based platform for dynamic analysis of NK cell cytolysis mediated by a triplebody. Analyst. 141 (7), 2284-2295 (2016).
- Sztilkovics, M., et al. Single-cell adhesion force kinetics of cell populations from combined label-free optical biosensor and robotic fluidic force microscopy. Scientific Reports. 10 (1), 1-13 (2020).

