Live-cell imaging av encelliga matriser (LISCA) - en mångsidig teknik för att kvantifiera cellulära kinetik
Summary
Vi presenterar en metod för förvärv av fluorescens reporter tidskurser från enstaka celler med hjälp av mikropatterned arrays. Protokollet beskriver förberedelsen av encelliga matriser, installation och drift av live-cell scanning time-lapse mikroskopi och ett open source bild analys verktyg för automatiserad förval, visuell kontroll och spårning av cellintegrerade fluorescens tids kurser per vidhäftnings plats.
Abstract
Live-cell Imaging of Single-Cell Arrays (LISCA) är en mångsidig metod för att samla tidskurser av fluorescenssignaler från enskilda celler i högt genomströmning. I allmänhet hämmas förvärvet av encelliga tidskurser från odlade celler av cellmotilitet och mångfald av cellformer. Självhäftande mikromatriser standardiserar encelliga förhållanden och underlättar bildanalys. LISCA kombinerar encelliga mikroarrayer med skanning av tidsfördröjningsmikroskopi och automatiserad bildbehandling. Här beskriver vi de experimentella stegen för att ta encelliga fluorescenstidskurser i LISCA-format. Vi transfect celler vidhäftande till en mikropatterned array med mRNA kodning för förbättrad grön fluorescerande protein (eGFP) och övervaka eGFP uttryck kinetik av hundratals celler parallellt via skanning tid-förfall mikroskopi. Bilddatastaplarna bearbetas automatiskt av nyutvecklad programvara som integrerar fluorescensintensitet över valda cellkonturer för att generera tidskurser för fluorescens i en cell. Vi visar att eGFP uttryck tid kurser efter mRNA transfection beskrivs väl av en enkel kinetisk översättning modell som avslöjar uttryck och nedbrytning priser av mRNA. Ytterligare tillämpningar av LISCA för händelse tid korrelationer av flera markörer i samband med signalering apoptos diskuteras.
Introduction
Under de senaste åren har vikten av encellsexperiment blivit uppenbar. Data från enstaka celler gör det möjligt att undersökning av cell-till-cell variabilitet, upplösningen av intracellulära parameterkorrelationer och detektion av cellulär kinetik som förblir dolda iensemblemätningar 1,2,3. För att undersöka cellulär kinetik för tusentals enstaka celler parallellt behövs nya metoder som gör det möjligt att övervaka cellerna under standardiserade förhållanden under en tidsperiod på flera timmar upp till flera dagar följt av en kvantitativ dataanalys 4. Här presenterar vi Live-cell Imaging of Single-Cell Arrays (LISCA), som kombinerar användningen av mikrostrukturerade matriser med timelapse-mikroskopi och automatiserad bildanalys.
Flera metoder för att generera mikrostrukturerade encelliga matriser har etablerats och publicerats i litteratur5,6. Här beskriver vi kortfattat Mikroskala Plasma-initierad proteinmönster (μPIPP). Ett detaljerat protokoll för encellsmatristillverkningen med μPIPP finns också i referens7. Användningen av encelliga matriser möjliggör justering av tusentals celler på standardiserade vidhäftningsfläckar som presenterar definierade mikromiljö för varje cell och minskar därmed en källa till experimentell variabilitet (figur 1A). Encelliga matriser används för att övervaka tidsförrättningar för fluorescerande markörer som är avsedda att indikera en mängd olika cellulära processer. Långsiktig mikroskopi i skanningstidsfördröjningsläge gör det möjligt att övervaka ett stort område av encellsmatriserna och därmed ta prov på encellsdata i hög genomströmning under en observationstid på flera timmar eller till och med dagar. Detta genererar tidsradsstaplar med bilder från varje position i matrisen (bild 1B). För att minska den stora mängden bilddata och extrahera önskade fluorescenstidskurser med en cell på ett effektivt sätt krävs automatiserad bildbehandling som utnyttjar cellernas placering (figur 1C).
Utmaningen med LISCA är att anpassa de experimentella protokollen och beräkningsverktygen för att bilda en höggenomströmningsanalys som genererar kvantitativa och reproducerbara data från cellulär kinetik. I den här artikeln ger vi en steg-för-steg-beskrivning av de enskilda metoderna och hur de kombineras i en LISCA-analys. Som ett exempel diskuterar vi tidsföringen för förbättrat grönt fluorescerande protein (eGFP) uttryck efter konstgjord mRNA leverans. eGFP uttryck efter mRNA leverans beskrivs av reaktionshastighet ekvationer modellering översättning och nedbrytning av mRNA. Att anpassa modellfunktionen för tidsförvröjningen av eGFP-koncentrationen till LISCA-avläsningen av fluorescensintensiteten för varje enskild cell över tiden ger bästa uppskattningar av modellparametrar som mRNA-nedbrytningshastigheten. Som ett representativt resultat diskuterar vi mRNA leverans effektivitet av två olika lipid-baserade transfection agenter och hur deras parameter distributioner skiljer sig åt.
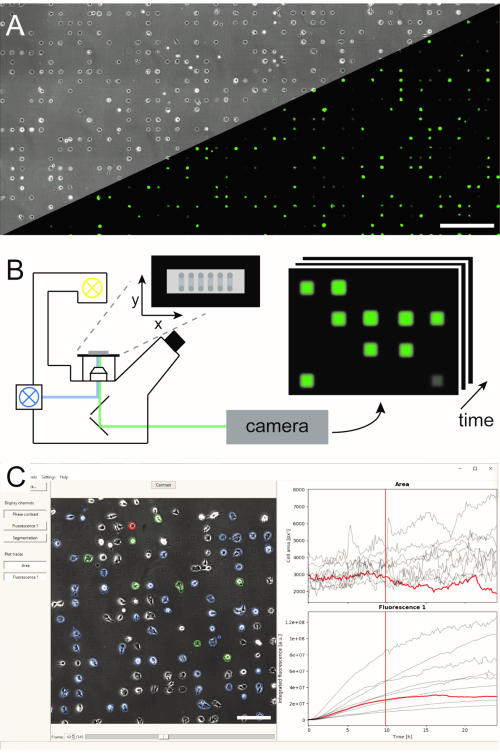
Bild 1:Representation av LISCA-arbetsflödet som kombinerar (A) mikromönstrade encelliga matriser (B) som skannar tidsfördröjningsmikroskopi och (C) automatiserad bildanalys av inspelade bildserier. Encelliga matriser består av ett tvådimensionellt mönster av celllimrutor med ett cellavvisande interrymd som leder till ett arrangemang av cellerna på mikromönster, vilket kan ses i faskontrastbilden samt fluorescensbilden av eGFP-uttrycksceller (A). Hela det mikrostrukturerade området avbildas i ett skanningstidsfördröjningsläge och tar upprepade gånger bilder i en sekvens av positioner (B). Inspelade bildserier bearbetas för att läsa upp fluorescensintensiteten per cell över tid (C). Skalstänger: 500 μm (A), 200 μm (C). Klicka här om du vill visa en större version av den här figuren.
Protocol
Representative Results
Discussion
Här beskrev vi LISCA som en mångsidig teknik för att följa cellulär kinetik av intracellulära fluorescerande etiketter på encellsnivå. För att utföra ett framgångsrikt LISCA-experiment måste vart och ett av de beskrivna stegen i protokollavsnittet upprättas individuellt och sedan måste alla steg kombineras. Var och en av de tre viktigaste aspekterna av LISCA har avgörande steg.
Encellig mikroarraytillverkning
Kvaliteten på mikroarrayen är avgörande eftersom cellul…
Declarações
The authors have nothing to disclose.
Acknowledgements
Detta arbete stöddes av bidrag från German Science Foundation (DFG) till Collaborative Research Center (SFB) 1032. Stöd från det tyska federala ministeriet för utbildning, forskning och teknik (BMBF) inom ramen för samarbetsprojektet 05K2018-2017-06716 Medisoft samt ett bidrag från Bayerische Forschungsstiftung är tacksamt erkänt. Anita Reiser fick stöd av ett DFG-stipendium genom Graduate School of Quantitative Biosciences Munich (QBM).
Materials
| Adtech Polymer Engineering PTFE Microtubing | Fisher Scientific | 10178071 | |
| baking oven | Binder | 9010-0190 | |
| CFI Plan Fluor DL 10x | Nikon | MRH20100 | |
| Desiccator | Roth | NX07.1 | |
| Eclipse Ti-E | Nikon | ||
| eGFP mRNA | Trilink | L-7601 | |
| Female Luer to Tube Connector | MEDNET | FTL210-6005 | |
| Fetal bovine serum | Thermo Fisher | 10270106 | |
| Fibronectin | Yo Proteins | 663 | |
| Filter set eGFP | AHF | F46-002 | |
| Fisherbrand Translucent Platinum-Cured Silicone Tubing | Fisher Scientific | 11768088 | |
| HEPES (1 M) | Thermo Fisher | 15630080 | |
| Incubation Box | Okolab | OKO-H201 | |
| incubator | Binder | 9040-0012 | |
| L-15 without phenol red | Thermo Fisher | 21083027 | |
| Lipofectamine 2000 | Thermo Fisher | 11668027 | |
| Male Luer | in-house fabricated consisting of teflon | ||
| Male Luer to Tube Connector | MEDNET | MTLS210-6005 | alternative to in-house fabricated male luers |
| NaCl (5 M) | Thermo Fisher | AM9760G | |
| Needleless Valve to Male Luer Connector | MEDNET | NVFMLLPC | |
| NIS Elements | Nikon | Imaging software Version 5.02.00 | |
| NOA81 | Thorlabs | NOA81 | Fast Curing Optical Adhesive for tube system assembly |
| Opti-MEM | Thermo Fisher | 31985062 | |
| PCO edge 4.2 M-USB-HQ-PCO | pco | ||
| Phosphate buffered saline (PBS) | in-house prepared | ||
| Plasma Cleaner | Diener Femto | Pico-BRS | |
| PLL(20 kDa)-g[3.5]-PEG(2 kDa) | SuSoS AG | ||
| silicon wafer mit mircorstructures | in-house fabricated | ||
| Sola Light Engine | Lumencor | ||
| sticky slide VI 0.4 | ibidi | 80608 | |
| Sylgard 184 Silicone Elastomer Kit | Dow Corning | 1673921 | |
| Tango 2 | Märzhäuser | 00-24-626-0000 | |
| Ultrapure water | in-house prepared | ||
| uncoated coverslips | ibidi | 10813 | |
| Injekt-F Solo, 1 mL | Omilab | 9166017V | with replacement sporn |
Referências
- Altschuler, S. J., Wu, L. F. Cellular heterogeneity: do differences make a difference. Cell. 141 (4), 559-563 (2010).
- Locke, J. C., Elowitz, M. B. Using movies to analyse gene circuit dynamics in single cells. Nature Reviews Microbiology. 7 (5), 383-392 (2009).
- Spencer, S. L., Gaudet, S., Albeck, J. G., Burke, J. M., Sorger, P. K. Non-genetic origins of cell-to-cell variability in TRAIL-induced apoptosis. Nature. 459 (7245), 428-432 (2009).
- El-Ali, J., Sorger, P. K., Jensen, K. F. Cells on chips. Nature. 442 (7101), 403-411 (2006).
- Segerer, F. J., et al. Versatile method to generate multiple types of micropatterns. Biointerphases. 11 (1), 011005 (2016).
- Piel, M., Théry, M. . Micropatterning in cell biology, part A/B/C. , (2014).
- Reiser, A., Zorn, M. L., Murschhauser, A., Rädler, J. O., Ertl, P., Rothbauer, M. . Cell-Based Microarrays: Methods and Protocols. , 41-54 (2018).
- Picone, R., Baum, B., McKendry, R. . Methods in cell biology. 119, 73-90 (2014).
- Reiser, A. . Single-cell time courses of mRNA transport and translation kinetics. , (2019).
- . Softmatter LMU-Raedler Group Available from: https://github.com/SoftmatterLMU-RaedlerGroup/pyama (2020)
- . MIAAB, 29262 Available from: https://github.com/SoftmatterLMU-RaedlerGroup/pyama (2020)
- Reiser, A., et al. Correlation of mRNA delivery timing and protein expression in lipid-based transfection. Integrative Biology. 11 (9), 362-371 (2019).
- Reiser, A., Woschée, D., Mehrotra, N., Krzysztoń, R. S., Strey, H. H., Rädler, J. O. Supplementing data and code for: “Correlation of mRNA delivery timing and protein expression in lipid-based transfection”. Zenodo. , (2019).
- Ferizi, M., et al. Stability analysis of chemically modified mRNA using micropattern-based single-cell arrays. Lab on a Chip. 15 (17), 3561-3571 (2015).
- Fröhlich, F., et al. Multi-experiment nonlinear mixed effect modeling of single-cell translation kinetics after transfection. NPJ systems biology and applications. 4 (1), 1-12 (2018).
- Krzysztoń, R., et al. Single-cell kinetics of siRNA-mediated mRNA degradation. Nanomedicine: Nanotechnology, Biology and Medicine. 21, 102077 (2019).
- Röttgermann, P. J., Alberola, A. P., Rädler, J. O. Cellular self-organization on micro-structured surfaces. Soft Matter. 10 (14), 2397-2404 (2014).
- Röttgermann, P. J., Dawson, K. A., Rädler, J. O. Time-resolved study of nanoparticle induced apoptosis using microfabricated single cell arrays. Microarrays. 5 (2), 8 (2016).
- Murschhauser, A., et al. A high-throughput microscopy method for single-cell analysis of event-time correlations in nanoparticle-induced cell death. Communications Biology. 2 (1), 1-11 (2019).
- Chatzopoulou, E. I., et al. Chip-based platform for dynamic analysis of NK cell cytolysis mediated by a triplebody. Analyst. 141 (7), 2284-2295 (2016).
- Sztilkovics, M., et al. Single-cell adhesion force kinetics of cell populations from combined label-free optical biosensor and robotic fluidic force microscopy. Scientific Reports. 10 (1), 1-13 (2020).

