Tek Hücreli Dizilerin Canlı Hücre Görüntülemesi (LISCA) - Hücresel Kinetiği Ölçmek için Çok Yönlü Bir Teknik
Summary
Mikropatterned diziler kullanılarak tek hücrelerden floresan muhabir zaman kurslarının alınması için bir yöntem sunuyoruz. Protokolde, tek hücreli dizilerin hazırlanması, canlı hücre tarama zaman atlamalı mikroskopinin kurulumu ve çalışması ve otomatik önseçim, görsel kontrol ve yapıştırma alanı başına hücre entegre floresan zaman kurslarının izlenmesi için açık kaynaklı bir görüntü analiz aracı açıklanmaktadır.
Abstract
Tek Hücreli Dizilerin Canlı Hücre Görüntülemesi (LISCA), yüksek verimdeki tek tek hücrelerden floresan sinyallerinin zaman akışlarını toplamak için çok yönlü bir yöntemdir. Genel olarak, kültürlü hücrelerden tek hücreli zaman kurslarının kazanılması hücre hareketliliği ve hücre şekillerinin çeşitliliği ile engellenir. Yapışkan mikro diziler tek hücreli koşulları standartlaştırır ve görüntü analizini kolaylaştırır. LISCA, tek hücreli mikroarrayları tarama zaman atlamalı mikroskopi ve otomatik görüntü işleme ile birleştirir. Burada, tek hücreli floresan zamanı derslerini LISCA formatında almanın deneysel adımlarını açıklıyoruz. Gelişmiş yeşil floresan protein (eGFP) için mRNA kodlaması kullanarak mikropatterned bir diziye yapışan hücreleri transfect ediyoruz ve tarama zaman atlamalı mikroskopi ile paralel olarak yüzlerce hücrenin eGFP ekspresyon kinetiğini izliyoruz. Görüntü veri yığınları, tek hücreli floresan süresi kursları oluşturmak için floresan yoğunluğunu seçilen hücre konturları üzerinde entegre eden yeni geliştirilen yazılım tarafından otomatik olarak işlenir. mRNA transfection sonrası eGFP ifade süresi derslerinin, mRNA’nın ifade ve bozulma oranlarını ortaya koyan basit bir kinetik çeviri modeli ile iyi tanımlandığını gösteriyoruz. Lisca’nın sinyal kesme apoptoz bağlamında birden fazla belirtecin olay süresi korelasyonları için daha fazla uygulaması tartışılmıştır.
Introduction
Son yıllarda tek hücreli deneylerin önemi belirginleşmiştir. Tek hücrelerden elde edilen veriler, hücreden hücreye değişkenliğin araştırılmasına, hücre içi parametre korelasyonlarının çözülmesine ve topluluk ölçümlerinde gizli kalan hücresel kinetiklerin tespitine izin verir1,2,3. Binlerce tek hücrenin hücresel kinetiğini paralel olarak araştırmak için, hücrelerin birkaç güne kadar birkaç saatlik bir zaman diliminde standart koşullar altında izlenmesini sağlayan yeni yaklaşımlara ihtiyaç vardır ve ardından nicel veri analizi 4. Burada, mikro yapılandırılmış dizilerin kullanımını zaman atlamalı mikroskopi ve otomatik görüntü analizi ile birleştiren Tek Hücreli Dizilerin Canlı Hücre Görüntülemesini (LISCA) sunuyoruz.
Mikro yapılandırılmış tek hücreli diziler üretmek için çeşitli yöntemler oluşturulmuş ve literatür5,6‘da yayınlanmıştır. Burada, Mikro Ölçekli Plazma Tarafından Başlatılan Protein Desenlemesini (μPIPP) kısaca açıklıyoruz. μPIPP kullanarak tek hücreli dizi imalatının ayrıntılı bir protokolü de referans7’debulunur. Tek hücreli dizilerin kullanımı, her hücre için tanımlanmış mikroçevirtiler sunan standartlaştırılmış yapışma noktalarına binlerce hücrenin hizalanmasına olanak tanır ve böylece deneysel değişkenlik kaynağını azaltır (Şekil 1A). Tek hücreli diziler, çeşitli hücresel süreçleri belirtmek için amaçlanan floresan belirteçlerin zaman akışlarını izlemek için kullanılır. Tarama zaman atlamalı modunda uzun süreli mikroskopi, tek hücreli dizilerin geniş bir alanını izlemenize ve böylece birkaç saat hatta gün boyunca yüksek aktarım hızına sahip tek hücreli verileri örneklemenize olanak tanır. Bu, dizinin her konumundan zaman çizgisi görüntü yığınları oluşturur (Şekil 1B). Büyük miktarda görüntü verisini azaltmak ve istenen tek hücreli floresan süresi derslerini verimli bir şekilde çıkarmak için, hücrelerin konumlandırılmasından yararlanan otomatik görüntü işleme gereklidir (Şekil 1C).
LISCA’nın zorluğu, deneysel protokolleri ve hesaplama araçlarını hücresel kinetiğin nicel ve tekrarlanabilir verilerini üreten yüksek verimli bir test oluşturacak şekilde uyarlamaktır. Bu yazıda, bireysel yöntemlerin ve bunların bir LISCA testinde nasıl birleştirildiklerinin adım adım bir açıklamasını sunuyoruz. Örnek olarak, yapay mRNA teslimatından sonra geliştirilmiş yeşil floresan protein (eGFP) ekspresyonunun zaman seyrini tartışıyoruz. mRNA teslimini izleyen eGFP ifadesi, reaksiyon hızı denklemleri modelleme çevirisi ve mRNA’nın bozulması ile açıklanmaktadır. eGFP konsantrasyonunun zaman akışı için model işlevinin zaman içinde her bir hücre için floresan yoğunluğunun LISCA okumasına takılması, mRNA bozulma hızı gibi model parametrelerinin en iyi tahminlerini verir. Temsili bir sonuç olarak, iki farklı lipid bazlı transfeksiyon ajanının mRNA teslimat verimliliğini ve parametre dağılımlarının nasıl farklılık gösterir olduğunu tartışıyoruz.
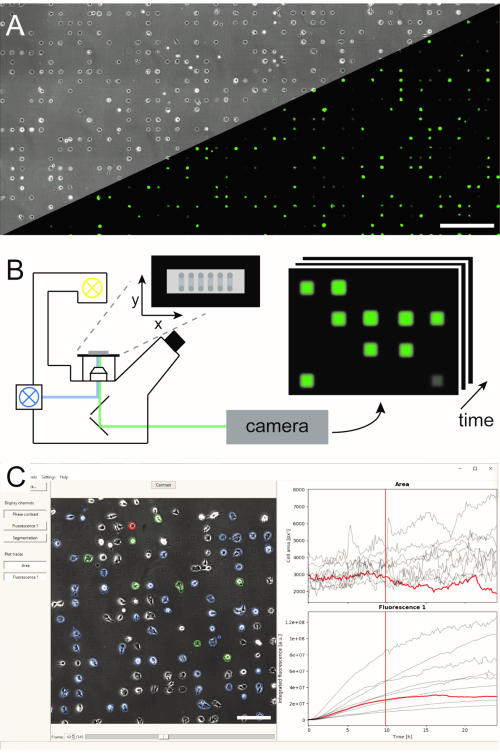
Şekil 1: (A) mikro desenli tek hücreli dizileri (B) tarama zaman atlamalı mikroskopi ve (C) kaydedilen görüntü serilerinin otomatik görüntü analizini birleştiren LISCA iş akışının gösterimi. Tek hücreli diziler, faz kontrastı görüntüsünün yanı sıra eGFP ifade hücrelerinin floresan görüntüsünde(A)görülebileceği gibi, mikropattern üzerindeki hücrelerin düzenlenmesine yol açan hücre itici ara alana sahip iki boyutlu bir hücre yapışkan kare deseni oluşur. Tüm mikro yapılandırılmış alan, bir dizi konumda(B)tekrar tekrar görüntü alan bir tarama zaman atlamalı modunda görüntülenir. Kaydedilen görüntü serileri, zaman içinde hücre başına floresan yoğunluğunu(C)okumak için işlenir. Ölçek çubukları: 500 μm (A), 200 μm (C). Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Protocol
Representative Results
Discussion
Burada LISCA’yı hücre içi floresan etiketlerin hücresel kinetiğini tek hücreli düzeyde takip etmek için çok yönlü bir teknik olarak tanımladık. Başarılı bir LISCA denemesi gerçekleştirmek için, protokol bölümünün açıklanan adımlarının her biri ayrı ayrı oluşturulmalı ve ardından tüm adımlar birleştirilmelidir. LISCA’nın üç ana yönünün her biri çok önemli adımlara sahiptir.
Tek hücreli mikroarray imalatı
Mikroarrayın kalitesi çok öne…
Declarações
The authors have nothing to disclose.
Acknowledgements
Bu çalışma, Alman Bilim Vakfı’nın (DFG) İşbirlikçi Araştırma Merkezi (SFB) 1032’ye verdiği hibelerle desteklendi. Almanya Federal Eğitim, Araştırma ve Teknoloji Bakanlığı’nın (BMBF) 05K2018-2017-06716 Medisoft kooperatif projesi kapsamındaki desteğinin yanı sıra Bayerische Forschungsstiftung’dan bir hibe de minnetle kabul edilmektedir. Anita Reiser, Nicel Biyobilim münih Enstitüsü (QBM) aracılığıyla bir DFG Bursu tarafından desteklendi.
Materials
| Adtech Polymer Engineering PTFE Microtubing | Fisher Scientific | 10178071 | |
| baking oven | Binder | 9010-0190 | |
| CFI Plan Fluor DL 10x | Nikon | MRH20100 | |
| Desiccator | Roth | NX07.1 | |
| Eclipse Ti-E | Nikon | ||
| eGFP mRNA | Trilink | L-7601 | |
| Female Luer to Tube Connector | MEDNET | FTL210-6005 | |
| Fetal bovine serum | Thermo Fisher | 10270106 | |
| Fibronectin | Yo Proteins | 663 | |
| Filter set eGFP | AHF | F46-002 | |
| Fisherbrand Translucent Platinum-Cured Silicone Tubing | Fisher Scientific | 11768088 | |
| HEPES (1 M) | Thermo Fisher | 15630080 | |
| Incubation Box | Okolab | OKO-H201 | |
| incubator | Binder | 9040-0012 | |
| L-15 without phenol red | Thermo Fisher | 21083027 | |
| Lipofectamine 2000 | Thermo Fisher | 11668027 | |
| Male Luer | in-house fabricated consisting of teflon | ||
| Male Luer to Tube Connector | MEDNET | MTLS210-6005 | alternative to in-house fabricated male luers |
| NaCl (5 M) | Thermo Fisher | AM9760G | |
| Needleless Valve to Male Luer Connector | MEDNET | NVFMLLPC | |
| NIS Elements | Nikon | Imaging software Version 5.02.00 | |
| NOA81 | Thorlabs | NOA81 | Fast Curing Optical Adhesive for tube system assembly |
| Opti-MEM | Thermo Fisher | 31985062 | |
| PCO edge 4.2 M-USB-HQ-PCO | pco | ||
| Phosphate buffered saline (PBS) | in-house prepared | ||
| Plasma Cleaner | Diener Femto | Pico-BRS | |
| PLL(20 kDa)-g[3.5]-PEG(2 kDa) | SuSoS AG | ||
| silicon wafer mit mircorstructures | in-house fabricated | ||
| Sola Light Engine | Lumencor | ||
| sticky slide VI 0.4 | ibidi | 80608 | |
| Sylgard 184 Silicone Elastomer Kit | Dow Corning | 1673921 | |
| Tango 2 | Märzhäuser | 00-24-626-0000 | |
| Ultrapure water | in-house prepared | ||
| uncoated coverslips | ibidi | 10813 | |
| Injekt-F Solo, 1 mL | Omilab | 9166017V | with replacement sporn |
Referências
- Altschuler, S. J., Wu, L. F. Cellular heterogeneity: do differences make a difference. Cell. 141 (4), 559-563 (2010).
- Locke, J. C., Elowitz, M. B. Using movies to analyse gene circuit dynamics in single cells. Nature Reviews Microbiology. 7 (5), 383-392 (2009).
- Spencer, S. L., Gaudet, S., Albeck, J. G., Burke, J. M., Sorger, P. K. Non-genetic origins of cell-to-cell variability in TRAIL-induced apoptosis. Nature. 459 (7245), 428-432 (2009).
- El-Ali, J., Sorger, P. K., Jensen, K. F. Cells on chips. Nature. 442 (7101), 403-411 (2006).
- Segerer, F. J., et al. Versatile method to generate multiple types of micropatterns. Biointerphases. 11 (1), 011005 (2016).
- Piel, M., Théry, M. . Micropatterning in cell biology, part A/B/C. , (2014).
- Reiser, A., Zorn, M. L., Murschhauser, A., Rädler, J. O., Ertl, P., Rothbauer, M. . Cell-Based Microarrays: Methods and Protocols. , 41-54 (2018).
- Picone, R., Baum, B., McKendry, R. . Methods in cell biology. 119, 73-90 (2014).
- Reiser, A. . Single-cell time courses of mRNA transport and translation kinetics. , (2019).
- . Softmatter LMU-Raedler Group Available from: https://github.com/SoftmatterLMU-RaedlerGroup/pyama (2020)
- . MIAAB, 29262 Available from: https://github.com/SoftmatterLMU-RaedlerGroup/pyama (2020)
- Reiser, A., et al. Correlation of mRNA delivery timing and protein expression in lipid-based transfection. Integrative Biology. 11 (9), 362-371 (2019).
- Reiser, A., Woschée, D., Mehrotra, N., Krzysztoń, R. S., Strey, H. H., Rädler, J. O. Supplementing data and code for: “Correlation of mRNA delivery timing and protein expression in lipid-based transfection”. Zenodo. , (2019).
- Ferizi, M., et al. Stability analysis of chemically modified mRNA using micropattern-based single-cell arrays. Lab on a Chip. 15 (17), 3561-3571 (2015).
- Fröhlich, F., et al. Multi-experiment nonlinear mixed effect modeling of single-cell translation kinetics after transfection. NPJ systems biology and applications. 4 (1), 1-12 (2018).
- Krzysztoń, R., et al. Single-cell kinetics of siRNA-mediated mRNA degradation. Nanomedicine: Nanotechnology, Biology and Medicine. 21, 102077 (2019).
- Röttgermann, P. J., Alberola, A. P., Rädler, J. O. Cellular self-organization on micro-structured surfaces. Soft Matter. 10 (14), 2397-2404 (2014).
- Röttgermann, P. J., Dawson, K. A., Rädler, J. O. Time-resolved study of nanoparticle induced apoptosis using microfabricated single cell arrays. Microarrays. 5 (2), 8 (2016).
- Murschhauser, A., et al. A high-throughput microscopy method for single-cell analysis of event-time correlations in nanoparticle-induced cell death. Communications Biology. 2 (1), 1-11 (2019).
- Chatzopoulou, E. I., et al. Chip-based platform for dynamic analysis of NK cell cytolysis mediated by a triplebody. Analyst. 141 (7), 2284-2295 (2016).
- Sztilkovics, M., et al. Single-cell adhesion force kinetics of cell populations from combined label-free optical biosensor and robotic fluidic force microscopy. Scientific Reports. 10 (1), 1-13 (2020).

