Identificación de las proteínas de unión de pequeños ligandos con la acción capilar radial diferencial del ensayo de ligandos (DRaCALA)
Summary
El ensayo Differential Radial Capillary Action of Ligand Assay (DRaCALA) se puede utilizar para identificar pequeñas proteínas de unión a ligandos de un organismo mediante el uso de una biblioteca ORFeome.
Abstract
La última década ha visto un tremendo progreso en la comprensión de pequeñas moléculas de señalización en la fisiología bacteriana. En particular, las proteínas diana de varios mensajeros secundarios derivados de nucleótidos (NSM) se han identificado y estudiado sistemáticamente en organismos modelo. Estos logros se deben principalmente al desarrollo de varias técnicas nuevas, incluida la técnica de compuestos de captura y la acción capilar radial diferencial del ensayo de ligando (DRaCALA), que se utilizaron para identificar sistemáticamente las proteínas diana de estas pequeñas moléculas. Este artículo describe el uso de los NSM, penta- y tetrafosfatos de guanosina (p)ppGpp, como ejemplo y demostración en video de la técnica DRaCALA. Usando DRaCALA, se identificaron 9 de las 20 proteínas diana conocidas y 12 nuevas de (p)ppGpp en el organismo modelo, Escherichia coli K-12, lo que demuestra el poder de este ensayo. En principio, DRaCALA podría usarse para estudiar pequeños ligandos que pueden ser etiquetados por isótopos radiactivos o colorantes fluorescentes. Los pasos críticos, los pros y los contras de DRaCALA se discuten aquí para una mayor aplicación de esta técnica.
Introduction
Las bacterias utilizan varias moléculas de señalización pequeñas para adaptarse a entornos en constante cambio1,2. Por ejemplo, los autoinductores, las lactonas de N-acilhomoserina y sus oligopéptidos modificados, median la comunicación intercelular entre las bacterias para coordinar el comportamiento de la población, un fenómeno conocido como detección de quórum2. Otro grupo de moléculas de señalización pequeñas son los NSM, incluidos el ampliamente estudiado monofosfato de adenosina cíclico (cAMP), el di-AMP cíclico, el monofosfato de di-guanosina cíclico (di-GMP cíclico) y los pentafosfatos de guanosina y tetra (p)ppGpp1. Las bacterias producen estos NSM como respuesta a una variedad de diferentes condiciones de estrés. Una vez producidas, estas moléculas se unen a sus proteínas diana y regulan varias vías fisiológicas y metabólicas diferentes para hacer frente a las tensiones encontradas y mejorar la supervivencia bacteriana. Por lo tanto, la identificación de las proteínas diana es un requisito previo inevitable para descifrar las funciones moleculares de estas pequeñas moléculas.
La última década ha sido testigo de un auge del conocimiento de estas pequeñas moléculas de señalización, principalmente debido a varias innovaciones técnicas que dieron a conocer las proteínas objetivo de estas pequeñas moléculas. Estos incluyen la técnica del compuesto de captura3,4,5y la acción capilar radial diferencial del ensayo de ligando (DRaCALA)6 que se discutirá en este artículo.
Inventado por Vincent Lee y sus compañeros de trabajo en 20116,DRaCALA despliega la capacidad de una membrana de nitrocelulosa para secuestrar diferencialmente ligandos libres y unidos a proteínas. Las moléculas como las proteínas no pueden difundirse en una membrana de nitrocelulosa, mientras que los ligandos pequeños, como los NSM, pueden hacerlo. Al mezclar el NSM(por ejemplo,ppGpp) con la proteína a probar y detectarlos en la membrana, se pueden esperar dos escenarios (Figura 1): Si (p)ppGpp se une a la proteína, el radiomarcado (p)ppGpp será retenido en el centro del punto por la proteína y no se difundirá hacia afuera, dando un punto pequeño intenso (es decir, señal radiactiva fuerte) bajo un fosforrimager. Sin embargo, si (p)ppGpp no se une a la proteína, se difundirá libremente hacia afuera para producir una gran mancha con una señal radiactiva de fondo uniforme.
Además, DRaCALA puede detectar la interacción entre una molécula pequeña y una proteína no purificada en un lisato de células enteras si la proteína está presente en una cantidad suficiente. Esta simplicidad permite el uso de DRaCALA en la identificación rápida de objetivos de proteínas mediante el uso de una biblioteca de expresiones ORFeome. De hecho, las proteínas diana de cAMP7, cíclico di-AMP8, cíclico di-GMP9,10y (p)ppGpp11,12,13 se han identificado sistemáticamente mediante el uso de DRaCALA. Este artículo en vídeo utiliza (p)ppGpp como ejemplo para demostrar y describir los pasos y consideraciones críticos para realizar una proyección exitosa de DRaCALA. Cabe destacar que se recomienda leer una descripción más completa de DRaCALA14 en combinación con este artículo antes de realizar DRaCALA.
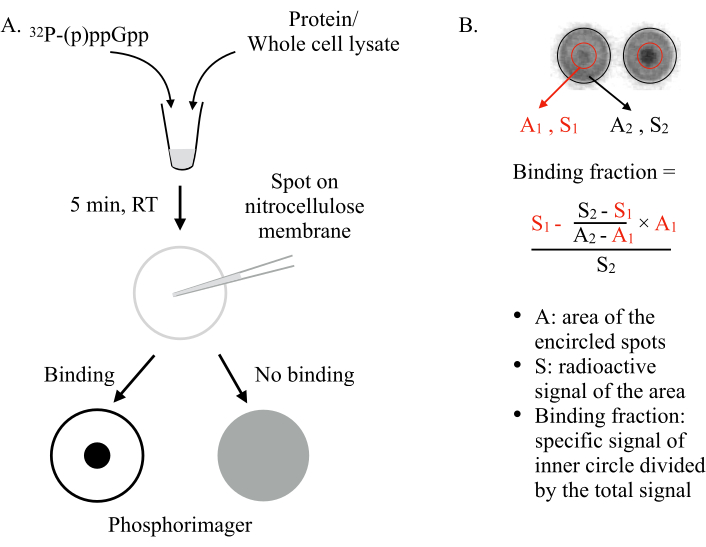
Figura 1: El principio de DRaCALA. (A) Esquema del ensayo DRaCALA. Consulte el texto para obtener más información. (B) Cuantificación y cálculo de la fracción de enlace. Consulte el texto para obtener más información. Brevemente, las manchas DRaCALA se analizarán dibujando dos círculos que circunscriben toda la mancha y el punto oscuro interno(es decir,el (p) ppGpp retenido debido a la unión de la proteína probada). La señal de unión específica es la señal radiactiva del círculo interior (S1) después de restar la señal de fondo no específica (calculada por A1 × ((S2-S1)/(A2-A1))). La fracción de unión es la señal de unión específica dividida por la señal radiactiva total (S2). Abreviaturas: DRaCALA = Acción Capilar Radial Diferencial del Ensayo de Ligandos; p)ppGpp = pentafosfatos de guanosina y tetrafosfatos; RT = temperatura ambiente. Haga clic aquí para ver una versión más grande de esta figura.
Protocol
Representative Results
Discussion
Uno de los pasos críticos para realizar el cribado de DRaCALA es obtener buenos lisados de células enteras. Primero, las proteínas probadas deben producirse en grandes cantidades y en formas solubles. En segundo lugar, la lisis de las células debe ser completa, y la viscosidad del lisato debe ser mínima. La inclusión de lisozima y el uso de tres ciclos de congelación-descongelación son a menudo suficientes para lisar las células por completo. Sin embargo, el ADN cromosómico liberado hace que el lisato sea visco…
Declarações
The authors have nothing to disclose.
Acknowledgements
El trabajo está respaldado por una subvención del proyecto NNF (NNF19OC0058331) a YEZ, y el programa de investigación e innovación Horizonte 2020 de la Unión Europea en virtud del acuerdo de subvención Marie Skłodowska-Curie (Nº 801199) a MLS.

Materials
| 32P-α-GTP | Perkinelmer | BLU006X250UC | |
| 96 x pin tool | V&P Scientific | VP 404 | 96 Bolt Replicator, on 9 mm centers, 4.2 mm Bolt Diameter, 24 mm long |
| 96-well V-bottom microtiter plate | Sterilin | MIC9004 | Sterilin Microplate V Well 611V96 |
| Agar | OXOID – Thermo Fisher | LP0011 | Agar no. 1 |
| ASKA collection strain | NBRP, SHIGEN, JAPAN | Ref: DNA Research, Volume 12, Issue 5, 2005, Pages 291–299. https://doi.org/10.1093/dnares/dsi012 | |
| Benzonase | SIGMA | E1014-25KU | genetically engineered endonuclease from Serratia marcescens |
| Bradford Protein Assay Dye | Bio-Rad | 5000006 | Reagent Concentrate |
| DMSO | SIGMA | D8418 | ≥99.9% |
| DNase 1 | SIGMA | DN25-1G | |
| gel filtration10x300 column | GE Healthcare | 28990944 | contains 20% ethanol as preservative |
| Glycerol | PanReac AppliChem | 122329.1214 | Glycerol 87% for analysis |
| Hypercassette | Amersham | RPN 11647 | 20 x 40 cm |
| Imidazole | SIGMA | 56750 | puriss. p.a., ≥ 99.5% (GC) |
| IP Storage Phosphor Screen | FUJIFILM | 28956474 | BAS-MS 2040 20x 40 cm |
| Isopropyl β-d-1-thiogalactopyranoside (IPTG) | SIGMA | I6758 | Isopropyl β-D-thiogalactoside |
| Lysogeny Broth (LB) | Invitrogen – Thermo Fisher | 12795027 | Miller's LB Broth Base |
| Lysozyme | SIGMA | L4949 | from chicken egg white; BioUltra, lyophilized powder, ≥98% |
| MgCl2 (Magnesium chloride) | SIGMA | 208337 | |
| MilliQ water | ultrapure water | ||
| multichannel pipette | Thermo Scientific | 4661110 | F1 – Clip Tip; 1-10 ul, 8 x channels |
| NaCl | VWR Chemicals | 27810 | AnalaR NORMAPUR, ACS, Reag. Ph. Eur. |
| Ni-NTA Agarose | Qiagen | 30230 | |
| Nitrocellulose Blotting Membrane | Amersham Protran | 10600003 | Premium 0.45 um 300 mm x 4 m |
| PBS | OXOID – Thermo Fisher | BR0014G | Phosphate buffered saline (Dulbecco A), Tablets |
| PEG3350 (Polyethylene glycol 3350) | SIGMA | 202444 | |
| phenylmethylsulfonyl fluoride (PMSF) | SIGMA | 93482 | Phenylmethanesulfonyl fluoride solution – 0.1 M in ethanol (T) |
| Phosphor-imager | GE Healthcare | 28955809 | Typhoon FLA-7000 Phosphor-imager |
| Pipette Tips, filtered | Thermo Scientific | 94410040 | ClipTip 12.5 μl nonsterile |
| Poly-Prep Chromatography column | Bio-Rad | 7311550 | polypropylene chromatography column |
| Protease inhibitor Mini | Pierce | A32955 | Tablets, EDTA-free |
| screw cap tube | Thermo Scientific | 3488 | Microcentifuge Tubes, 2.0 ml with screw cap, nonsterile |
| SLS 96-deep Well plates | Greiner | 780285 | MASTERBLOCK, 2 ML, PP, V-Bottom, Natural |
| spin column | Millipore | UFC500396 | Amicon Ultra -0.5 ml Centrifugal Filters |
| Thermomixer | Eppendorf | 5382000015 | Thermomixer C |
| TLC plate (PEI-cellulose F TLC plates) | Merck Millipore | 105579 | DC PEI-cellulose F (20 x 20 cm) |
| Tris | SIGMA | BP152 | Tris Base for Molecular Biology |
| Tween 20 | SIGMA | P1379 | viscous non-ionic detergent |
| β-mercaptoethanol | SIGMA | M3148 | 99% (GC/titration) |
Referências
- Kalia, D., et al. Nucleotide, c-di-GMP, c-di-AMP, cGMP, cAMP, (p)ppGpp signaling in bacteria and implications in pathogenesis. Chemical Society Reviews. 42 (1), 305-341 (2013).
- Camilli, A., Bassler, B. L. Bacterial small-molecule signaling pathways. Science. 311 (5764), 1113-1116 (2006).
- Luo, Y., et al. The cAMP capture compound mass spectrometry as a novel tool for targeting cAMP-binding proteins: from protein kinase A to potassium/sodium hyperpolarization-activated cyclic nucleotide-gated channels. Molecular & Cellular Proteomics. 8 (12), 2843-2856 (2009).
- Nesper, J., Reinders, A., Glatter, T., Schmidt, A., Jenal, U. A novel capture compound for the identification and analysis of cyclic di-GMP binding proteins. Journal of Proteomics. 75 (15), 4874-4878 (2012).
- Laventie, B. J., et al. Capture compound mass spectrometry–a powerful tool to identify novel c-di-GMP effector proteins. Journal of Visual Experiments. (97), e51404 (2015).
- Roelofs, K. G., Wang, J., Sintim, H. O., Lee, V. T. Differential radial capillary action of ligand assay for high-throughput detection of protein-metabolite interactions. Proceedings of the National Academy of Sciences of the United States of America. 108 (37), 15528-15533 (2011).
- Zhang, Y., et al. Evolutionary adaptation of the essential tRNA methyltransferase TrmD to the signaling molecule 3 ‘,5 ‘-cAMP in bacteria. Journal of Biological Chemistry. 292 (1), 313-327 (2017).
- Corrigan, R. M., et al. Systematic identification of conserved bacterial c-di-AMP receptor proteins. Proceedings of the National Academy of Sciences of the United States of America. 110 (22), 9084-9089 (2013).
- Roelofs, K. G., et al. Systematic identification of cyclic-di-GMP binding proteins in Vibrio cholerae reveals a novel class of cyclic-di-GMP-binding ATPases associated with type II secretion systems. PLoS Pathogen. 11 (10), 1005232 (2015).
- Fang, X., et al. GIL, a new c-di-GMP-binding protein domain involved in regulation of cellulose synthesis in enterobacteria. Molecular Microbiology. 93 (3), 439-452 (2014).
- Corrigan, R. M., Bellows, L. E., Wood, A., Grundling, A. ppGpp negatively impacts ribosome assembly affecting growth and antimicrobial tolerance in Gram-positive bacteria. Proceedings of the National Academy of Sciences of the United States of America. 113 (12), 1710-1719 (2016).
- Zhang, Y., Zbornikova, E., Rejman, D., Gerdes, K. Novel (p)ppGpp binding and metabolizing proteins of Escherichia coli. Mbio. 9 (2), 02188 (2018).
- Yang, J., et al. The nucleotide pGpp acts as a third alarmone in Bacillus, with functions distinct from those of (p) ppGpp. Nature Communications. 11 (1), 5388 (2020).
- Orr, M. W., Lee, V. T. Differential radial capillary action of ligand assay (DRaCALA) for high-throughput detection of protein-metabolite interactions in bacteria. Methods in Molecular Biology. 1535, 25-41 (2017).
- Kitagawa, M., et al. Complete set of ORF clones of Escherichia coli ASKA library (A complete Set of E. coli K-12 ORF archive): Unique resources for biological research. DNA Research. 12 (5), 291-299 (2005).
- Hochstadt-Ozer, J., Cashel, M. The regulation of purine utilization in bacteria. V. Inhibition of purine phosphoribosyltransferase activities and purine uptake in isolated membrane vesicles by guanosine tetraphosphate. Journal of Biological Chemistry. 247 (21), 7067-7072 (1972).
- Zhang, Y. E., et al. p)ppGpp regulates a bacterial nucleosidase by an allosteric two-domain switch. Molecular Cell. 74 (6), 1239-1249 (2019).
- Wang, B., et al. Affinity-based capture and identification of protein effectors of the growth regulator ppGpp. Nature Chemical Biology. 15 (2), 141-150 (2019).

