Identification globale des réseaux d’interaction co-translationnelle par profilage sélectif des ribosomes
Summary
Les interactions co-traductionnelles jouent un rôle crucial dans les modifications de la chaîne naissante, le ciblage, le pliage et les voies d’assemblage. Nous décrivons ici le profilage sélectif des ribosomes, une méthode d’analyse directe in vivo de ces interactions dans le modèle eucaryote Saccharomyces cerevisiae.
Abstract
Ces dernières années, il est devenu évident que les ribosomes non seulement décodent notre ARNm, mais guident également l’émergence de la chaîne polypeptidique dans l’environnement cellulaire encombré. Les ribosomes fournissent la plate-forme pour la liaison contrôlée spatialement et cinétiquement des facteurs de ciblage membranaire, modifiant les enzymes et pliant les chaperons. Même l’assemblage en complexes oligomères d’ordre élevé, ainsi que les étapes de formation du réseau protéine-protéine, ont récemment été découverts pour être coordonnés avec la synthèse.
Nous décrivons ici le profilage sélectif des ribosomes, une méthode développée pour capturer les interactions co-translationnelles in vivo. Nous détaillerons les différentes étapes de purification d’affinité requises pour capturer les complexes de chaîne naissante de ribosomes avec des interacteurs co-traductionnels, ainsi que les étapes d’extraction de l’ARNm, d’exclusion de taille, de transcription inverse, de séquençage en profondeur et d’analyse de données volumineuses, nécessaires pour déchiffrer les interactions co-traductionnelles en résolution proche du codon.
Introduction
Lerofilage de l’ibosome RR (SeRP) est la seule méthode, à ce jour, qui capture et caractérise les interactions co-translationnelles, in vivo, de manière directe 1,2,3,4,5,6. SeRP permet un profilage global des interactions de n’importe quel facteur avec la traduction des ribosomes en résolution proche du codon 2,7.
La méthode repose sur la congélation éclair des cellules en croissance et la préservation de la traduction active. Les lysats cellulaires sont ensuite traités avec de la RNase I pour digérer tout l’ARNm dans la cellule, à l’exception des fragments d’ARNm protégés par les ribosomes appelés « empreintes de ribosomes ». L’échantillon est ensuite divisé en deux parties; une partie est directement utilisée pour l’isolement de toutes les empreintes ribosomiques cellulaires, représentant toute la traduction en cours dans la cellule. La deuxième partie est utilisée pour la purification d’affinité du sous-ensemble spécifique de ribosomes associés à un facteur d’intérêt, par exemple: enzymes modificatrices, facteurs de translocation, chaperons de repliement et interactions d’assemblage complexe. Les empreintes ribosomiques purifiées par affinité sont collectivement appelées interactome. Ensuite, les ARNm protégés par les ribosomes sont extraits et utilisés pour la génération de bibliothèque d’ADNc, suivie d’un séquençage en profondeur.
L’analyse comparative des échantillons totaux de translatome et d’interactome permet d’identifier tous les orfs associés au facteur d’intérêt, ainsi que la caractérisation de chaque profil d’interaction orf. Ce profil rapporte les séquences précises d’apparition et de terminaison de l’engagement à partir desquelles on peut déduire les codons décodés et les résidus respectifs de la chaîne polypeptidique émergente, ainsi que sur les variations de vitesse des ribosomes au cours de l’interaction 7,8. La figure 1 illustre le protocole sous forme de schéma.
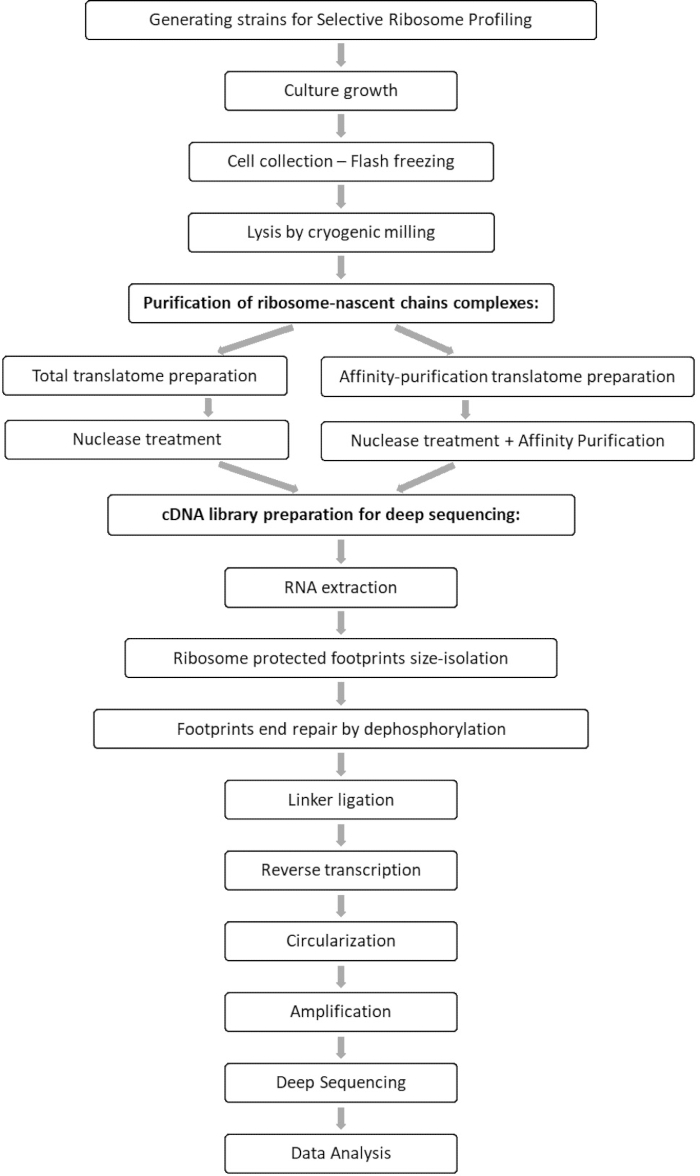
Figure 1 : Vue d’ensemble du protocole SeRP. Ce protocole peut être effectué dans son intégralité dans un délai de 7 à 10 jours. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Protocol
Representative Results
Discussion
Ici, le protocole détaille l’approche de profilage sélectif des ribosomes pour capturer les interactions co-traductionnelles dans une résolution proche du codon. Au fur et à mesure que le ribosome devient une plaque tournante pour coordonner l’émergence de la chaîne naissante dans le cytoplasme surpeuplé, il s’agit d’une méthode cruciale pour identifier et caractériser les diverses interactions co-translationnelles nécessaires pour assurer un protéome fonctionnel, ainsi que pour étudier diverses mala…
Declarações
The authors have nothing to disclose.
Acknowledgements
Nous tenons à remercier tous les membres du laboratoire pour les discussions fructueuses et Muhammad Makhzumy pour la lecture critique du manuscrit. Ce travail a été financé par la subvention 2106/20 de l’ISF (Israeli Science Foundation).
Materials
| 3'-Phosphorylated 28 nt RNA control oligonucleotide | IDT | custom order | RNase free HPLC purification; 5'-AUGUAGUCGGAGUCGAGGCGC GACGCGA/3Phos/-3' |
| Absolute ethanol | VWR | 20821 | |
| Acid phenol–chloroform | Ambion | AM9722 | |
| Antibody: mouse monclonal anti-HA | Merck | 11583816001 | 12CA5 |
| Aprotinin | Roth | A162.3 | |
| ATP* | NEB | P0756S | 10 mM |
| Bacto agar | BD | 214030 | |
| Bacto peptone | BD | 211820 | |
| Bacto tryptone | BD | 211699 | |
| Bacto yeast extract | BD | 212720 | |
| Bestatin hydrochloride | Roth | 2937.2 | |
| Chloroform | Merck | 102445 | |
| CircLigase II ssDNA Ligase* | Epicentre | CL9025K | 100 U/μL |
| Colloidal Coomassie staining solution | Roth | 4829 | |
| cOmplete, EDTA-free protease inhibitor cocktail tablets | Roche Diagnostics | 29384100 | |
| Cycloheximide | Biological Industries | A0879 | |
| DEPC treated and sterile filtered water* | Sigma | 95284 | |
| D-Glucose anhydrous | Merck | G5767-500G | |
| Diethylpyrocarbonate | Roth | K028 | |
| Dimethylsulfoxide* | Sigma-Aldrich | 276855 | |
| DNA ladder, 10 bp O'RangeRuler* | Thermo Fisher Scientific | SM1313 | |
| DNA loading dye* | Thermo Fisher Scientific | R0631 | 6× |
| DNase I, recombinant | Roche | 4716728001 | RNAse free |
| dNTP solution set* | NEB | N0446S | |
| EDTA* | Roth | 8043 | |
| Glycerol | VWR | 24388.260. | |
| Glycine solution | Sigma-Aldrich | 67419-1ML-F | 1 M |
| GlycoBlue | Ambion | AM9516 | 15 mg/mL |
| HEPES | Roth | HN78.3 | |
| HF Phusion polymerase* | NEB | M0530L | |
| HK from S. cerevisiae | Sigma-Aldrich | H6380-1.5KU | |
| Hydrochloric acid | AppliChem | A1305 | |
| Isopropanol | Sigma-Aldrich | 33539 | |
| Isopropyl β-D-1-thiogalactopyranoside | Roth | CN08 | |
| Kanamycin | Roth | T832.4 | |
| KCl | Roth | 6781.1 | |
| KH2PO4 | Roth | 3904.1 | |
| Leupeptin | Roth | CN33.4 | |
| Linker L(rt) | IDT | custom order | |
| Liquid nitrogen | |||
| MgCl2 | Roth | KK36.3 | |
| Na2HPO4 | Roth | P030.2 | |
| Na2HPO4·2H2O | Roth | T879.3 | |
| NaCl* | Invitrogen | AM97606 | 5 M |
| NaH2PO4·H2O | Roth | K300.2 | |
| NHS-activated Sepharose 4 fast-flow beads | GE Life Sciences | 17090601 | |
| Nonidet P 40 substitute | Sigma | 74385 | |
| Pepstatin A | Roth | 2936.2 | |
| Phenylmethyl sulfonyl fluoride | Roth | 6367 | |
| Precast gels | Bio-Rad | 5671034 | 10% and 12% |
| RNase I | Ambion | AM2294 | |
| SDS, 20% | Ambion | AM9820 | RNase free |
| Sodium acetate* | Ambion | AM9740 | 3 M, pH 5.5 |
| Sodium azide | Merck | S8032-100G | |
| Sodium chloride | Roth | 9265 | |
| Sodium hydroxide* | Sigma | S2770 | 1 N |
| Sucrose | Sigma-Aldrich | 16104 | |
| SUPERase-In RNase Inhibitor | Ambion | AM2694 | |
| Superscript III Reverse Transciptase* | Invitrogen | 18080-044 | |
| SYBR Gold* | Invitrogen | S11494 | |
| T4 polynucleotide kinase* | NEB | M0201L | |
| T4 RNA ligase 2* | NEB | M0242L | |
| TBE polyacrylamide gel* | Novex | EC6215BOX | 8% |
| TBE–urea polyacrylamide gel* | Novex | EC68752BOX | 10% |
| TBE–urea polyacrylamide gel* | Novex | EC6885BOX | 15% |
| TBE–urea sample buffer* | Novex | LC6876 | 2× |
| Tris | Roth | 4855 | |
| Tris* | Ambion | AM9851 | 1 M, pH 7.0 |
| Tris* | Ambion | AM9856 | 1 M, pH 8.0 |
| UltraPure 10× TBE buffer* | Invitrogen | 15581-044 | |
| * – for library preparation | |||
| gasket and spring clamp , 90 mm, | Millipore | XX1009020 | |
| ground joint flask 1 L , | Millipore | XX1504705 |
Referências
- Oh, E., et al. Selective ribosome profiling reveals the cotranslational chaperone action of trigger factor in vivo. Cell. 147 (6), 1295-1308 (2011).
- Shiber, A., et al. Cotranslational assembly of protein complexes in eukaryotes revealed by ribosome profiling. Nature. 561 (7722), 268-272 (2018).
- Becker, A. H., Oh, E., Weissman, J. S., Kramer, G., Bukau, B. Selective ribosome profiling as a tool for studying the interaction of chaperones and targeting factors with nascent polypeptide chains and ribosomes. Nature Protocols. 8 (11), 2212-2239 (2013).
- Galmozzi, C. V., Merker, D., Friedrich, U. A., Döring, K., Kramer, G. Selective ribosome profiling to study interactions of translating ribosomes in yeast. Nature Protocols. , (2019).
- Knorr, A. G., et al. Ribosome-NatA architecture reveals that rRNA expansion segments coordinate N-terminal acetylation. Nature Structural and Molecular Biology. 26 (1), 35-39 (2019).
- Matsuo, Y., Inada, T. The ribosome collision sensor Hel2 functions as preventive quality control in the secretory pathway. Cell Reports. 34 (12), (2021).
- Döring, K., et al. Profiling Ssb-Nascent chain interactions reveals principles of Hsp70-assisted folding. Cell. , (2017).
- Chartron, J. W., Hunt, K. C. L., Frydman, J. Cotranslational signal-independent SRP preloading during membrane targeting. Nature. 536 (7615), 224-228 (2016).
- Janke, C., et al. A versatile toolbox for PCR-based tagging of yeast genes: New fluorescent proteins, more markers and promoter substitution cassettes. Yeast. 21 (11), 947-962 (2004).
- Levi, O., Arava, Y. Expanding the CRISPR/Cas9 Toolbox for Gene Engineering in S. cerevisiae. Current Microbiology. 77 (3), 468-478 (2020).
- Giannoukos, G., et al. Efficient and robust RNA-seq process for cultured bacteria and complex community transcriptomes. Genome Biology. 13 (3), 23 (2012).
- . Illumina Index Adapters – Pooling Guide Available from: https://support.illumina.com/content/dam/illumina-support/documents/documentation/chemistry_documentation/experiment-design/index-adapters-pooling-guide-1000000041074-05.pdf (2019)
- Kanagawa, T. Bias and artifacts in multitemplate polymerase chain reactions (PCR). Journal of Bioscience and Bioengineering. 96 (4), 317-323 (2003).
- Bertolini, M., et al. Interactions between nascent proteins translated by adjacent ribosomes drive homomer assembly. Science. 371 (6524), (2021).
- Kramer, G., Shiber, A., Bukau, B. Mechanisms of cotranslational maturation of newly synthesized proteins. Annual Review of Biochemistry. 88, 337-364 (2019).
- Joazeiro, C. A. P. Mechanisms and functions of ribosome-associated protein quality control. Nature Reviews Molecular Cell Biology. 20 (6), 368-383 (2019).
- Beaupere, C., Chen, R. B., Pelosi, W., Labunskyy, V. M. Genome-wide quantification of translation in budding yeast by ribosome profiling. Journal of Visualized Experiments: JoVE. (130), e56820 (2017).

