선택적 리보솜 프로파일링을 통한 공동 번역 상호 작용 네트워크의 글로벌 식별
Summary
공동 번역 상호 작용은 초기 체인 수정, 타겟팅, 폴딩 및 조립 경로에서 중요한 역할을합니다. 여기에서, 우리는 선택적 리보솜 프로파일링, 생체 내에서, 모델 진핵생물 사카로마이세스 세레비시아에서 이러한 상호작용의 직접적인 분석을 위한 방법을 설명한다.
Abstract
최근 몇 년 동안, 리보솜은 우리의 mRNA를 해독 할뿐만 아니라 혼잡 한 세포 환경으로 폴리펩티드 사슬의 출현을 안내한다는 것이 분명 해졌다. 리보솜은 막 표적화 인자의 공간적 및 동역학적으로 조절된 결합, 효소 변형 및 접이식 샤페론을 위한 플랫폼을 제공한다. 심지어 고차 올리고머 복합체로의 조립뿐만 아니라 단백질-단백질 네트워크 형성 단계도 합성과 배위되는 것으로 최근에 발견되었다.
여기에서는 생체 내에서 공동 번역 상호 작용을 캡처하기 위해 개발 된 방법 인 선택적 리보솜 프로파일 링 에 대해 설명합니다. 우리는 공동 번역 상호 작용기와 함께 리보솜 – 초기 사슬 복합체를 캡처하는 데 필요한 다양한 친화성 정제 단계뿐만 아니라 mRNA 추출, 크기 배제, 역전사, 딥 시퀀싱 및 빅 데이터 분석 단계를 자세히 설명합니다 거의 코돈 분해능에서 공동 번역 상호 작용을 해독하는 데 필요합니다.
Introduction
Se lective Ribosome Profiling (SeRP)은 현재까지 1,2,3,4,5,6 직접적인 방식으로 생체 내에서 공동 번역 상호 작용을 캡처하고 특성화하는 유일한 방법입니다. SeRP는 코돈 분해능 2,7에 가까운 리보솜을 번역하는 모든 인자의 상호작용에 대한 글로벌 프로파일링을 가능하게 한다.
이 방법은 성장하는 세포의 플래시 동결과 활성 번역 보존에 의존합니다. 이어서, 세포 용해물은 RNase I로 처리되어 “리보솜 발자국”으로 불리는 리보솜-보호된 mRNA 단편을 제외한 세포 내의 모든 mRNA를 소화시킨다. 그런 다음 샘플을 두 부분으로 나눕니다. 한 부분은 모든 세포 리보솜 발자국의 분리에 직접 사용되며, 이는 세포에서 진행중인 모든 번역을 나타냅니다. 두 번째 부분은 관심 인자와 관련된 리보솜의 특정 서브세트의 친화도-정제를 위해 사용된다: 예를 들어: 변형 효소, 전좌 인자, 폴딩 샤페론, 및 복합체-어셈블리 상호작용. 친화도 정제된 리보솜 발자국은 집합적으로 인터랙텀(interactome)이라고 불린다. 이어서, 리보솜-보호된 mRNA를 추출하고 cDNA 라이브러리 생성에 사용하고, 이어서 딥 시퀀싱을 수행한다.
전체 트랜스라톰 및 인터랙토메 샘플의 비교 분석은 관심 인자와 관련된 모든 orfs의 동정뿐만 아니라 각 orf 상호작용 프로파일의 특성화를 허용한다. 이 프로파일은 정확한 결합 개시 및 종결 서열을 보고하며, 이로부터 해독된 코돈 및 신흥 폴리펩티드 쇄의 각각의 잔기를 추론할 수 있을 뿐만 아니라, 상호작용 동안 리보솜 속도 변이(7,8)에 대해서도 보고한다. 그림 1은 프로토콜을 회로도로 보여줍니다.
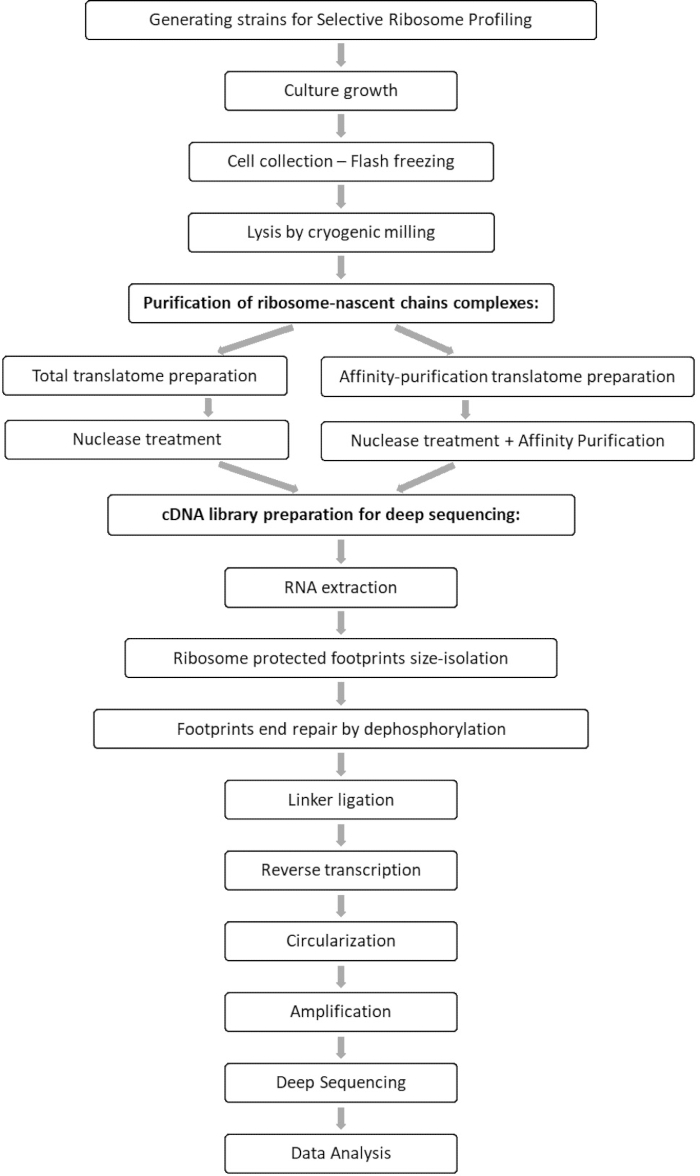
그림 1: SeRP 프로토콜의 개요 이 프로토콜은 7-10 일 이내에 전체적으로 수행 할 수 있습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
Protocol
Representative Results
Discussion
여기서, 프로토콜은 코돈 분해능에 가까운 공동-번역 상호작용을 캡처하기 위한 선택적 리보솜 프로파일링 접근법을 상세히 설명한다. 리보솜이 붐비는 세포질로 출현하는 초기 사슬을 조정하기위한 허브로 상승함에 따라, 이것은 기능적 프로테옴을 보장하고 다양한 질병을 연구하는 데 필요한 다양한 공동 번역 상호 작용을 확인하고 특성화하는 중요한 방법입니다. 현재까지, SeRP는 생체내<…
Declarações
The authors have nothing to disclose.
Acknowledgements
우리는 유익한 토론을 해준 모든 실험실 구성원들과 원고의 비판적 독서에 대한 무하마드 Makhzumy에게 감사하고 싶습니다. 이 작품은 ISF (이스라엘 과학 재단) 보조금 2106/20에 의해 지원되었습니다.
Materials
| 3'-Phosphorylated 28 nt RNA control oligonucleotide | IDT | custom order | RNase free HPLC purification; 5'-AUGUAGUCGGAGUCGAGGCGC GACGCGA/3Phos/-3' |
| Absolute ethanol | VWR | 20821 | |
| Acid phenol–chloroform | Ambion | AM9722 | |
| Antibody: mouse monclonal anti-HA | Merck | 11583816001 | 12CA5 |
| Aprotinin | Roth | A162.3 | |
| ATP* | NEB | P0756S | 10 mM |
| Bacto agar | BD | 214030 | |
| Bacto peptone | BD | 211820 | |
| Bacto tryptone | BD | 211699 | |
| Bacto yeast extract | BD | 212720 | |
| Bestatin hydrochloride | Roth | 2937.2 | |
| Chloroform | Merck | 102445 | |
| CircLigase II ssDNA Ligase* | Epicentre | CL9025K | 100 U/μL |
| Colloidal Coomassie staining solution | Roth | 4829 | |
| cOmplete, EDTA-free protease inhibitor cocktail tablets | Roche Diagnostics | 29384100 | |
| Cycloheximide | Biological Industries | A0879 | |
| DEPC treated and sterile filtered water* | Sigma | 95284 | |
| D-Glucose anhydrous | Merck | G5767-500G | |
| Diethylpyrocarbonate | Roth | K028 | |
| Dimethylsulfoxide* | Sigma-Aldrich | 276855 | |
| DNA ladder, 10 bp O'RangeRuler* | Thermo Fisher Scientific | SM1313 | |
| DNA loading dye* | Thermo Fisher Scientific | R0631 | 6× |
| DNase I, recombinant | Roche | 4716728001 | RNAse free |
| dNTP solution set* | NEB | N0446S | |
| EDTA* | Roth | 8043 | |
| Glycerol | VWR | 24388.260. | |
| Glycine solution | Sigma-Aldrich | 67419-1ML-F | 1 M |
| GlycoBlue | Ambion | AM9516 | 15 mg/mL |
| HEPES | Roth | HN78.3 | |
| HF Phusion polymerase* | NEB | M0530L | |
| HK from S. cerevisiae | Sigma-Aldrich | H6380-1.5KU | |
| Hydrochloric acid | AppliChem | A1305 | |
| Isopropanol | Sigma-Aldrich | 33539 | |
| Isopropyl β-D-1-thiogalactopyranoside | Roth | CN08 | |
| Kanamycin | Roth | T832.4 | |
| KCl | Roth | 6781.1 | |
| KH2PO4 | Roth | 3904.1 | |
| Leupeptin | Roth | CN33.4 | |
| Linker L(rt) | IDT | custom order | |
| Liquid nitrogen | |||
| MgCl2 | Roth | KK36.3 | |
| Na2HPO4 | Roth | P030.2 | |
| Na2HPO4·2H2O | Roth | T879.3 | |
| NaCl* | Invitrogen | AM97606 | 5 M |
| NaH2PO4·H2O | Roth | K300.2 | |
| NHS-activated Sepharose 4 fast-flow beads | GE Life Sciences | 17090601 | |
| Nonidet P 40 substitute | Sigma | 74385 | |
| Pepstatin A | Roth | 2936.2 | |
| Phenylmethyl sulfonyl fluoride | Roth | 6367 | |
| Precast gels | Bio-Rad | 5671034 | 10% and 12% |
| RNase I | Ambion | AM2294 | |
| SDS, 20% | Ambion | AM9820 | RNase free |
| Sodium acetate* | Ambion | AM9740 | 3 M, pH 5.5 |
| Sodium azide | Merck | S8032-100G | |
| Sodium chloride | Roth | 9265 | |
| Sodium hydroxide* | Sigma | S2770 | 1 N |
| Sucrose | Sigma-Aldrich | 16104 | |
| SUPERase-In RNase Inhibitor | Ambion | AM2694 | |
| Superscript III Reverse Transciptase* | Invitrogen | 18080-044 | |
| SYBR Gold* | Invitrogen | S11494 | |
| T4 polynucleotide kinase* | NEB | M0201L | |
| T4 RNA ligase 2* | NEB | M0242L | |
| TBE polyacrylamide gel* | Novex | EC6215BOX | 8% |
| TBE–urea polyacrylamide gel* | Novex | EC68752BOX | 10% |
| TBE–urea polyacrylamide gel* | Novex | EC6885BOX | 15% |
| TBE–urea sample buffer* | Novex | LC6876 | 2× |
| Tris | Roth | 4855 | |
| Tris* | Ambion | AM9851 | 1 M, pH 7.0 |
| Tris* | Ambion | AM9856 | 1 M, pH 8.0 |
| UltraPure 10× TBE buffer* | Invitrogen | 15581-044 | |
| * – for library preparation | |||
| gasket and spring clamp , 90 mm, | Millipore | XX1009020 | |
| ground joint flask 1 L , | Millipore | XX1504705 |
Referências
- Oh, E., et al. Selective ribosome profiling reveals the cotranslational chaperone action of trigger factor in vivo. Cell. 147 (6), 1295-1308 (2011).
- Shiber, A., et al. Cotranslational assembly of protein complexes in eukaryotes revealed by ribosome profiling. Nature. 561 (7722), 268-272 (2018).
- Becker, A. H., Oh, E., Weissman, J. S., Kramer, G., Bukau, B. Selective ribosome profiling as a tool for studying the interaction of chaperones and targeting factors with nascent polypeptide chains and ribosomes. Nature Protocols. 8 (11), 2212-2239 (2013).
- Galmozzi, C. V., Merker, D., Friedrich, U. A., Döring, K., Kramer, G. Selective ribosome profiling to study interactions of translating ribosomes in yeast. Nature Protocols. , (2019).
- Knorr, A. G., et al. Ribosome-NatA architecture reveals that rRNA expansion segments coordinate N-terminal acetylation. Nature Structural and Molecular Biology. 26 (1), 35-39 (2019).
- Matsuo, Y., Inada, T. The ribosome collision sensor Hel2 functions as preventive quality control in the secretory pathway. Cell Reports. 34 (12), (2021).
- Döring, K., et al. Profiling Ssb-Nascent chain interactions reveals principles of Hsp70-assisted folding. Cell. , (2017).
- Chartron, J. W., Hunt, K. C. L., Frydman, J. Cotranslational signal-independent SRP preloading during membrane targeting. Nature. 536 (7615), 224-228 (2016).
- Janke, C., et al. A versatile toolbox for PCR-based tagging of yeast genes: New fluorescent proteins, more markers and promoter substitution cassettes. Yeast. 21 (11), 947-962 (2004).
- Levi, O., Arava, Y. Expanding the CRISPR/Cas9 Toolbox for Gene Engineering in S. cerevisiae. Current Microbiology. 77 (3), 468-478 (2020).
- Giannoukos, G., et al. Efficient and robust RNA-seq process for cultured bacteria and complex community transcriptomes. Genome Biology. 13 (3), 23 (2012).
- . Illumina Index Adapters – Pooling Guide Available from: https://support.illumina.com/content/dam/illumina-support/documents/documentation/chemistry_documentation/experiment-design/index-adapters-pooling-guide-1000000041074-05.pdf (2019)
- Kanagawa, T. Bias and artifacts in multitemplate polymerase chain reactions (PCR). Journal of Bioscience and Bioengineering. 96 (4), 317-323 (2003).
- Bertolini, M., et al. Interactions between nascent proteins translated by adjacent ribosomes drive homomer assembly. Science. 371 (6524), (2021).
- Kramer, G., Shiber, A., Bukau, B. Mechanisms of cotranslational maturation of newly synthesized proteins. Annual Review of Biochemistry. 88, 337-364 (2019).
- Joazeiro, C. A. P. Mechanisms and functions of ribosome-associated protein quality control. Nature Reviews Molecular Cell Biology. 20 (6), 368-383 (2019).
- Beaupere, C., Chen, R. B., Pelosi, W., Labunskyy, V. M. Genome-wide quantification of translation in budding yeast by ribosome profiling. Journal of Visualized Experiments: JoVE. (130), e56820 (2017).

