Identificação Global de Redes de Interação Co-Translacional por Perfil Seletivo Ribosome
Summary
As interações co-translacionais desempenham um papel crucial nas modificações da cadeia nascente, na segmentação, na dobra e nas vias de montagem. Aqui, descrevemos o Perfil Ribossomo Seletivo, um método para in vivo, análise direta dessas interações no modelo eucariote Saccharomyces cerevisiae.
Abstract
Nos últimos anos, tornou-se evidente que ribossomos não só decodificam nosso mRNA, mas também guiam o surgimento da cadeia de polipeptídeos no ambiente celular lotado. Os ribossomos fornecem a plataforma para a ligação espacial e cineticamente controlada de fatores de segmentação de membrana, modificando enzimas e acompanhantes dobráveis. Até mesmo a montagem em complexos oligomericos de alta ordem, bem como etapas de formação de rede proteína-proteína, foram recentemente descobertas como coordenadas com síntese.
Aqui, descrevemos o Selective Ribosome Profiling, um método desenvolvido para capturar interações co-translacionais in vivo. Detalharemos as várias etapas de purificação de afinidade necessárias para capturar complexos ribossomo-nascentes, juntamente com os interlaciadores co-translacionais, bem como a extração de mRNA, exclusão de tamanho, transcrição reversa, sequenciamento profundo e etapas de análise de big data, necessárias para decifrar interações co-translacionais em resolução de quase codon.
Introduction
Selective Ribosome Profiling (SeRP) é o único método, até o momento, que captura e caracteriza interações co-translacionais, in vivo, de forma direta 1,2,3,4,5,6. O SeRP permite o perfil global de interações de qualquer fator com a tradução de ribossomos na resolução 2,7 de códon próximo.
O método se baseia no congelamento flash de células em crescimento e na preservação da tradução ativa. Os lises celulares são então tratados com RNase I para digerir todo o mRNA na célula, exceto fragmentos de mRNA protegidos por ribossomos denominados “pegadas ribossósas”. A amostra é então dividida em duas partes; uma parte é diretamente usada para o isolamento de todas as pegadas ribossômicas celulares, representando toda a tradução contínua na célula. A segunda parte é utilizada para a afinidade-purificação do subconjunto específico de ribossomos associados a um fator de interesse, por exemplo: modificação de enzimas, fatores de translocação, acompanhantes dobráveis e interações de montagem complexa. As pegadas ribossômicas purificadas por afinidade são coletivamente chamadas de interactome. Em seguida, os mRNAs protegidos por ribossomo são extraídos e usados para a geração de biblioteca cDNA, seguido de sequenciamento profundo.
A análise comparativa das amostras totais de translatome e interactome permite a identificação de todos os orfs que associam ao fator de interesse, bem como a caracterização de cada perfil de interação orf. Este perfil relata as sequências precisas de início e término de engajamento a partir das quais se pode inferir os códons decodificados e os respectivos resíduos da cadeia emergente de polipeptídeos, bem como sobre as variações de velocidade ribossosome durante a interação 7,8. A Figura 1 retrata o protocolo como um esquema.
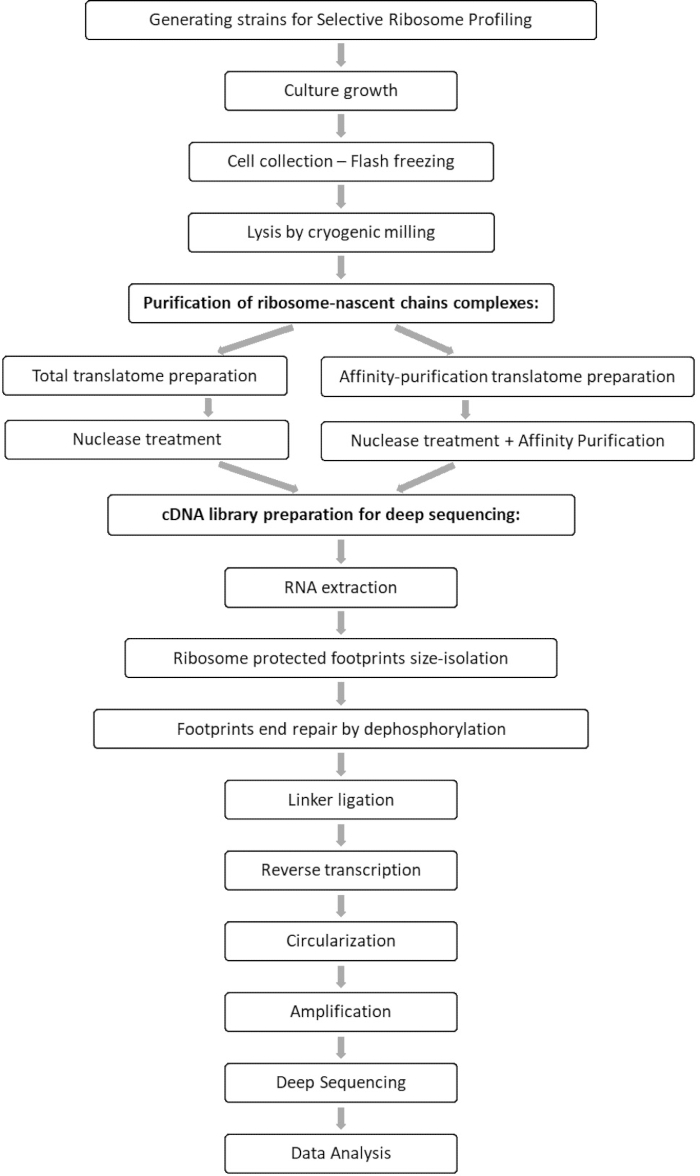
Figura 1: Uma visão geral do protocolo SeRP. Este protocolo pode ser realizado em sua totalidade dentro de 7-10 dias. Clique aqui para ver uma versão maior desta figura.
Protocol
Representative Results
Discussion
Aqui, o protocolo detalha a abordagem de Perfil Ribossomo Seletivo para capturar interações co-translacionais em resolução de códon próximo. À medida que o ribossomo surge como um centro para coordenar o surgimento da cadeia nascente no citoplasma lotado, este é um método crucial para identificar e caracterizar as diversas interações co-translacionais necessárias para garantir um proteome funcional, bem como para o estudo de várias doenças. Até o momento, o SeRP é o único método que pode capturar e car…
Declarações
The authors have nothing to disclose.
Acknowledgements
Gostaríamos de agradecer a todos os membros do laboratório por discussões frutíferas e Muhammad Makhzumy pela leitura crítica do manuscrito. Este trabalho foi financiado pela isf (Israel Science Foundation) grant 2106/20.
Materials
| 3'-Phosphorylated 28 nt RNA control oligonucleotide | IDT | custom order | RNase free HPLC purification; 5'-AUGUAGUCGGAGUCGAGGCGC GACGCGA/3Phos/-3' |
| Absolute ethanol | VWR | 20821 | |
| Acid phenol–chloroform | Ambion | AM9722 | |
| Antibody: mouse monclonal anti-HA | Merck | 11583816001 | 12CA5 |
| Aprotinin | Roth | A162.3 | |
| ATP* | NEB | P0756S | 10 mM |
| Bacto agar | BD | 214030 | |
| Bacto peptone | BD | 211820 | |
| Bacto tryptone | BD | 211699 | |
| Bacto yeast extract | BD | 212720 | |
| Bestatin hydrochloride | Roth | 2937.2 | |
| Chloroform | Merck | 102445 | |
| CircLigase II ssDNA Ligase* | Epicentre | CL9025K | 100 U/μL |
| Colloidal Coomassie staining solution | Roth | 4829 | |
| cOmplete, EDTA-free protease inhibitor cocktail tablets | Roche Diagnostics | 29384100 | |
| Cycloheximide | Biological Industries | A0879 | |
| DEPC treated and sterile filtered water* | Sigma | 95284 | |
| D-Glucose anhydrous | Merck | G5767-500G | |
| Diethylpyrocarbonate | Roth | K028 | |
| Dimethylsulfoxide* | Sigma-Aldrich | 276855 | |
| DNA ladder, 10 bp O'RangeRuler* | Thermo Fisher Scientific | SM1313 | |
| DNA loading dye* | Thermo Fisher Scientific | R0631 | 6× |
| DNase I, recombinant | Roche | 4716728001 | RNAse free |
| dNTP solution set* | NEB | N0446S | |
| EDTA* | Roth | 8043 | |
| Glycerol | VWR | 24388.260. | |
| Glycine solution | Sigma-Aldrich | 67419-1ML-F | 1 M |
| GlycoBlue | Ambion | AM9516 | 15 mg/mL |
| HEPES | Roth | HN78.3 | |
| HF Phusion polymerase* | NEB | M0530L | |
| HK from S. cerevisiae | Sigma-Aldrich | H6380-1.5KU | |
| Hydrochloric acid | AppliChem | A1305 | |
| Isopropanol | Sigma-Aldrich | 33539 | |
| Isopropyl β-D-1-thiogalactopyranoside | Roth | CN08 | |
| Kanamycin | Roth | T832.4 | |
| KCl | Roth | 6781.1 | |
| KH2PO4 | Roth | 3904.1 | |
| Leupeptin | Roth | CN33.4 | |
| Linker L(rt) | IDT | custom order | |
| Liquid nitrogen | |||
| MgCl2 | Roth | KK36.3 | |
| Na2HPO4 | Roth | P030.2 | |
| Na2HPO4·2H2O | Roth | T879.3 | |
| NaCl* | Invitrogen | AM97606 | 5 M |
| NaH2PO4·H2O | Roth | K300.2 | |
| NHS-activated Sepharose 4 fast-flow beads | GE Life Sciences | 17090601 | |
| Nonidet P 40 substitute | Sigma | 74385 | |
| Pepstatin A | Roth | 2936.2 | |
| Phenylmethyl sulfonyl fluoride | Roth | 6367 | |
| Precast gels | Bio-Rad | 5671034 | 10% and 12% |
| RNase I | Ambion | AM2294 | |
| SDS, 20% | Ambion | AM9820 | RNase free |
| Sodium acetate* | Ambion | AM9740 | 3 M, pH 5.5 |
| Sodium azide | Merck | S8032-100G | |
| Sodium chloride | Roth | 9265 | |
| Sodium hydroxide* | Sigma | S2770 | 1 N |
| Sucrose | Sigma-Aldrich | 16104 | |
| SUPERase-In RNase Inhibitor | Ambion | AM2694 | |
| Superscript III Reverse Transciptase* | Invitrogen | 18080-044 | |
| SYBR Gold* | Invitrogen | S11494 | |
| T4 polynucleotide kinase* | NEB | M0201L | |
| T4 RNA ligase 2* | NEB | M0242L | |
| TBE polyacrylamide gel* | Novex | EC6215BOX | 8% |
| TBE–urea polyacrylamide gel* | Novex | EC68752BOX | 10% |
| TBE–urea polyacrylamide gel* | Novex | EC6885BOX | 15% |
| TBE–urea sample buffer* | Novex | LC6876 | 2× |
| Tris | Roth | 4855 | |
| Tris* | Ambion | AM9851 | 1 M, pH 7.0 |
| Tris* | Ambion | AM9856 | 1 M, pH 8.0 |
| UltraPure 10× TBE buffer* | Invitrogen | 15581-044 | |
| * – for library preparation | |||
| gasket and spring clamp , 90 mm, | Millipore | XX1009020 | |
| ground joint flask 1 L , | Millipore | XX1504705 |
Referências
- Oh, E., et al. Selective ribosome profiling reveals the cotranslational chaperone action of trigger factor in vivo. Cell. 147 (6), 1295-1308 (2011).
- Shiber, A., et al. Cotranslational assembly of protein complexes in eukaryotes revealed by ribosome profiling. Nature. 561 (7722), 268-272 (2018).
- Becker, A. H., Oh, E., Weissman, J. S., Kramer, G., Bukau, B. Selective ribosome profiling as a tool for studying the interaction of chaperones and targeting factors with nascent polypeptide chains and ribosomes. Nature Protocols. 8 (11), 2212-2239 (2013).
- Galmozzi, C. V., Merker, D., Friedrich, U. A., Döring, K., Kramer, G. Selective ribosome profiling to study interactions of translating ribosomes in yeast. Nature Protocols. , (2019).
- Knorr, A. G., et al. Ribosome-NatA architecture reveals that rRNA expansion segments coordinate N-terminal acetylation. Nature Structural and Molecular Biology. 26 (1), 35-39 (2019).
- Matsuo, Y., Inada, T. The ribosome collision sensor Hel2 functions as preventive quality control in the secretory pathway. Cell Reports. 34 (12), (2021).
- Döring, K., et al. Profiling Ssb-Nascent chain interactions reveals principles of Hsp70-assisted folding. Cell. , (2017).
- Chartron, J. W., Hunt, K. C. L., Frydman, J. Cotranslational signal-independent SRP preloading during membrane targeting. Nature. 536 (7615), 224-228 (2016).
- Janke, C., et al. A versatile toolbox for PCR-based tagging of yeast genes: New fluorescent proteins, more markers and promoter substitution cassettes. Yeast. 21 (11), 947-962 (2004).
- Levi, O., Arava, Y. Expanding the CRISPR/Cas9 Toolbox for Gene Engineering in S. cerevisiae. Current Microbiology. 77 (3), 468-478 (2020).
- Giannoukos, G., et al. Efficient and robust RNA-seq process for cultured bacteria and complex community transcriptomes. Genome Biology. 13 (3), 23 (2012).
- . Illumina Index Adapters – Pooling Guide Available from: https://support.illumina.com/content/dam/illumina-support/documents/documentation/chemistry_documentation/experiment-design/index-adapters-pooling-guide-1000000041074-05.pdf (2019)
- Kanagawa, T. Bias and artifacts in multitemplate polymerase chain reactions (PCR). Journal of Bioscience and Bioengineering. 96 (4), 317-323 (2003).
- Bertolini, M., et al. Interactions between nascent proteins translated by adjacent ribosomes drive homomer assembly. Science. 371 (6524), (2021).
- Kramer, G., Shiber, A., Bukau, B. Mechanisms of cotranslational maturation of newly synthesized proteins. Annual Review of Biochemistry. 88, 337-364 (2019).
- Joazeiro, C. A. P. Mechanisms and functions of ribosome-associated protein quality control. Nature Reviews Molecular Cell Biology. 20 (6), 368-383 (2019).
- Beaupere, C., Chen, R. B., Pelosi, W., Labunskyy, V. M. Genome-wide quantification of translation in budding yeast by ribosome profiling. Journal of Visualized Experiments: JoVE. (130), e56820 (2017).

