Ko-Translasyonel Etkileşim Ağlarının Seçici Ribozom Profilleme ile Küresel Tanımlanması
Summary
Ko-translasyonel etkileşimler, yeni zincir modifikasyonlarında, hedeflemede, katlamada ve montaj yollarında çok önemli bir rol oynamaktadır. Burada, ökaryot Saccharomyces cerevisiae modelinde bu etkileşimlerin in vivo, doğrudan analizi için bir yöntem olan Seçici Ribozom Profillemeyi açıklıyoruz.
Abstract
Son yıllarda, ribozomların sadece mRNA’mızın şifresini çözmekle kalmayıp, aynı zamanda polipeptit zincirinin kalabalık hücresel ortama ortaya çıkmasına rehberlik ettiği de ortaya çıkmıştır. Ribozomlar, membran hedefleme faktörlerinin uzamsal ve kinetik olarak kontrol edilen bağlanması, enzimlerin modifiye edilmesi ve şaperonların katlanması için platform sağlar. Yüksek dereceli oligomerik komplekslere ve protein-protein ağı oluşum adımlarına montajın bile sentezle koordine edildiği yakın zamanda keşfedilmiştir.
Burada, in vivo olarak eş-translasyonel etkileşimleri yakalamak için geliştirilmiş bir yöntem olan Seçici Ribozom Profillemeyi açıklıyoruz. Ribozom-nazen-zincir komplekslerini ko-translasyonel interaktörlerle birlikte yakalamak için gereken çeşitli afinite saflaştırma adımlarını ve ayrıca kodona yakın çözünürlükte ko-translasyonel etkileşimleri deşifre etmek için gereken mRNA ekstraksiyonu, boyut dışlama, ters transkripsiyon, derin dizileme ve büyük veri analizi adımlarını detaylandıracağız.
Introduction
Selective Ribozom Profiling (SeRP), bugüne kadar, ko-translasyonel etkileşimleri, in vivo, doğrudan bir şekilde 1,2,3,4,5,6 olarak yakalayan ve karakterize eden tek yöntemdir. SeRP, ribozomların 2,7’ye yakın çözünürlükte çevrilmesiyle herhangi bir faktörün etkileşimlerinin global profillenmesini sağlar.
Yöntem, büyüyen hücrelerin flaş dondurulmasına ve aktif çevirinin korunmasına dayanır. Hücre lizatları daha sonra “ribozom ayak izleri” olarak adlandırılan ribozom korumalı mRNA fragmanları hariç hücredeki tüm mRNA’yı sindirmek için RNaz I ile muamele edilir. Numune daha sonra iki bölüme ayrılır; Bir kısım doğrudan tüm hücresel ribozomal ayak izlerinin izolasyonu için kullanılır ve hücrede devam eden tüm translasyonu temsil eder. İkinci bölüm, bir ilgi faktörü ile ilişkili ribozomların spesifik alt kümesinin afinite saflaştırılması için kullanılır, örneğin: enzimleri değiştirmek, translokasyon faktörleri, şaperonları katlamak ve karmaşık montaj etkileşimleri. Afinite saflaştırılmış ribozomal ayak izleri topluca interaktom olarak adlandırılır. Daha sonra, ribozom korumalı mRNA’lar ekstrakte edilir ve cDNA kütüphanesi üretimi için kullanılır, ardından derin dizileme yapılır.
Toplam translatom ve interaktom örneklerinin karşılaştırmalı analizi, ilgi faktörü ile ilişkili tüm orfların tanımlanmasına ve her bir orf etkileşim profilinin karakterizasyonuna izin verir. Bu profil, kodu çözülmüş kodonları ve ortaya çıkan polipeptit zincirinin ilgili kalıntılarını ve ayrıca etkileşim 7,8 sırasındaki ribozom hız değişimlerini çıkarabileceğiniz kesin katılım başlangıcı ve sonlandırma dizilerini rapor eder. Şekil 1’de protokol şematik olarak gösterilmektedir.
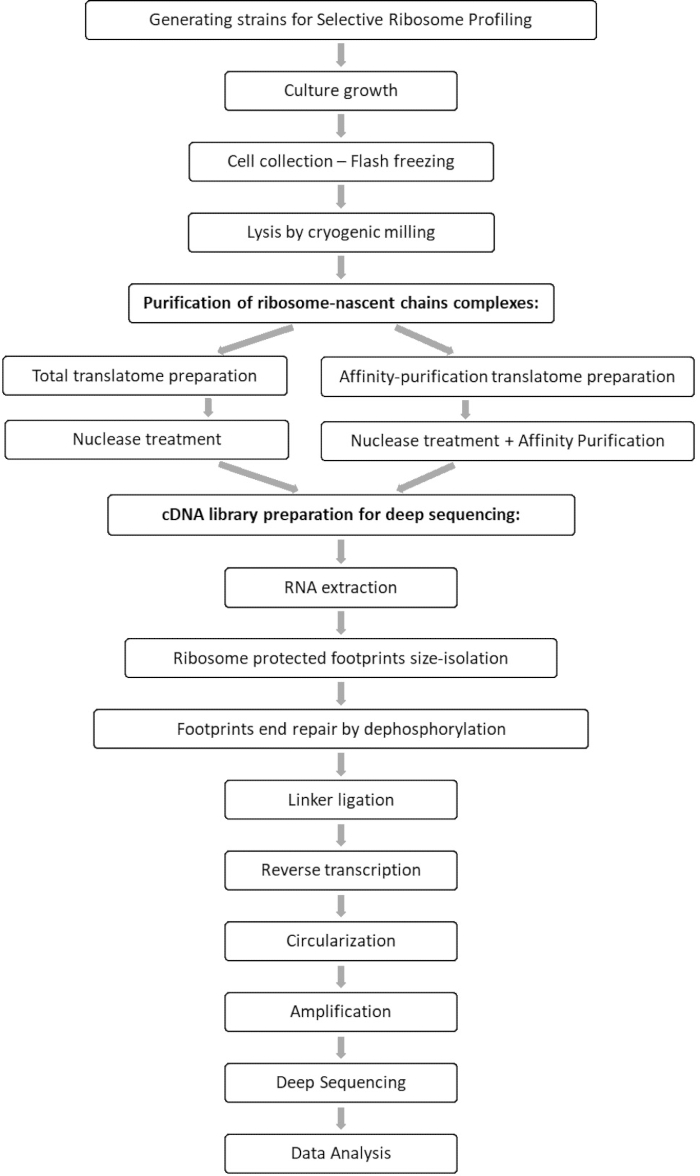
Şekil 1: SeRP protokolüne genel bakış. Bu protokol 7-10 gün içinde bütünüyle gerçekleştirilebilir. Bu şeklin daha büyük bir versiyonunu görüntülemek için lütfen buraya tıklayın.
Protocol
Representative Results
Discussion
Burada, protokol, yakın kodon çözünürlüğünde ko-translasyonel etkileşimleri yakalamak için Seçici Ribozom Profilleme yaklaşımını detaylandırmaktadır. Ribozom, kalabalık sitoplazmaya yeni zincir oluşumunu koordine etmek için bir merkez olarak yükseldiğinden, bu, fonksiyonel bir proteom sağlamak ve çeşitli hastalıkları incelemek için gereken çeşitli ko-translasyonel etkileşimleri tanımlamak ve karakterize etmek için çok önemli bir yöntemdir. Bugüne kadar, SeRP bu etkileşimleri doğrud…
Declarações
The authors have nothing to disclose.
Acknowledgements
Verimli tartışmalar için tüm laboratuvar üyelerine ve makalenin eleştirel okuması için Muhammed Makhzumy’ye teşekkür ederiz. Bu çalışma ISF (İsrail Bilim Vakfı) hibe 2106/20 tarafından finanse edilmiştir.
Materials
| 3'-Phosphorylated 28 nt RNA control oligonucleotide | IDT | custom order | RNase free HPLC purification; 5'-AUGUAGUCGGAGUCGAGGCGC GACGCGA/3Phos/-3' |
| Absolute ethanol | VWR | 20821 | |
| Acid phenol–chloroform | Ambion | AM9722 | |
| Antibody: mouse monclonal anti-HA | Merck | 11583816001 | 12CA5 |
| Aprotinin | Roth | A162.3 | |
| ATP* | NEB | P0756S | 10 mM |
| Bacto agar | BD | 214030 | |
| Bacto peptone | BD | 211820 | |
| Bacto tryptone | BD | 211699 | |
| Bacto yeast extract | BD | 212720 | |
| Bestatin hydrochloride | Roth | 2937.2 | |
| Chloroform | Merck | 102445 | |
| CircLigase II ssDNA Ligase* | Epicentre | CL9025K | 100 U/μL |
| Colloidal Coomassie staining solution | Roth | 4829 | |
| cOmplete, EDTA-free protease inhibitor cocktail tablets | Roche Diagnostics | 29384100 | |
| Cycloheximide | Biological Industries | A0879 | |
| DEPC treated and sterile filtered water* | Sigma | 95284 | |
| D-Glucose anhydrous | Merck | G5767-500G | |
| Diethylpyrocarbonate | Roth | K028 | |
| Dimethylsulfoxide* | Sigma-Aldrich | 276855 | |
| DNA ladder, 10 bp O'RangeRuler* | Thermo Fisher Scientific | SM1313 | |
| DNA loading dye* | Thermo Fisher Scientific | R0631 | 6× |
| DNase I, recombinant | Roche | 4716728001 | RNAse free |
| dNTP solution set* | NEB | N0446S | |
| EDTA* | Roth | 8043 | |
| Glycerol | VWR | 24388.260. | |
| Glycine solution | Sigma-Aldrich | 67419-1ML-F | 1 M |
| GlycoBlue | Ambion | AM9516 | 15 mg/mL |
| HEPES | Roth | HN78.3 | |
| HF Phusion polymerase* | NEB | M0530L | |
| HK from S. cerevisiae | Sigma-Aldrich | H6380-1.5KU | |
| Hydrochloric acid | AppliChem | A1305 | |
| Isopropanol | Sigma-Aldrich | 33539 | |
| Isopropyl β-D-1-thiogalactopyranoside | Roth | CN08 | |
| Kanamycin | Roth | T832.4 | |
| KCl | Roth | 6781.1 | |
| KH2PO4 | Roth | 3904.1 | |
| Leupeptin | Roth | CN33.4 | |
| Linker L(rt) | IDT | custom order | |
| Liquid nitrogen | |||
| MgCl2 | Roth | KK36.3 | |
| Na2HPO4 | Roth | P030.2 | |
| Na2HPO4·2H2O | Roth | T879.3 | |
| NaCl* | Invitrogen | AM97606 | 5 M |
| NaH2PO4·H2O | Roth | K300.2 | |
| NHS-activated Sepharose 4 fast-flow beads | GE Life Sciences | 17090601 | |
| Nonidet P 40 substitute | Sigma | 74385 | |
| Pepstatin A | Roth | 2936.2 | |
| Phenylmethyl sulfonyl fluoride | Roth | 6367 | |
| Precast gels | Bio-Rad | 5671034 | 10% and 12% |
| RNase I | Ambion | AM2294 | |
| SDS, 20% | Ambion | AM9820 | RNase free |
| Sodium acetate* | Ambion | AM9740 | 3 M, pH 5.5 |
| Sodium azide | Merck | S8032-100G | |
| Sodium chloride | Roth | 9265 | |
| Sodium hydroxide* | Sigma | S2770 | 1 N |
| Sucrose | Sigma-Aldrich | 16104 | |
| SUPERase-In RNase Inhibitor | Ambion | AM2694 | |
| Superscript III Reverse Transciptase* | Invitrogen | 18080-044 | |
| SYBR Gold* | Invitrogen | S11494 | |
| T4 polynucleotide kinase* | NEB | M0201L | |
| T4 RNA ligase 2* | NEB | M0242L | |
| TBE polyacrylamide gel* | Novex | EC6215BOX | 8% |
| TBE–urea polyacrylamide gel* | Novex | EC68752BOX | 10% |
| TBE–urea polyacrylamide gel* | Novex | EC6885BOX | 15% |
| TBE–urea sample buffer* | Novex | LC6876 | 2× |
| Tris | Roth | 4855 | |
| Tris* | Ambion | AM9851 | 1 M, pH 7.0 |
| Tris* | Ambion | AM9856 | 1 M, pH 8.0 |
| UltraPure 10× TBE buffer* | Invitrogen | 15581-044 | |
| * – for library preparation | |||
| gasket and spring clamp , 90 mm, | Millipore | XX1009020 | |
| ground joint flask 1 L , | Millipore | XX1504705 |
Referências
- Oh, E., et al. Selective ribosome profiling reveals the cotranslational chaperone action of trigger factor in vivo. Cell. 147 (6), 1295-1308 (2011).
- Shiber, A., et al. Cotranslational assembly of protein complexes in eukaryotes revealed by ribosome profiling. Nature. 561 (7722), 268-272 (2018).
- Becker, A. H., Oh, E., Weissman, J. S., Kramer, G., Bukau, B. Selective ribosome profiling as a tool for studying the interaction of chaperones and targeting factors with nascent polypeptide chains and ribosomes. Nature Protocols. 8 (11), 2212-2239 (2013).
- Galmozzi, C. V., Merker, D., Friedrich, U. A., Döring, K., Kramer, G. Selective ribosome profiling to study interactions of translating ribosomes in yeast. Nature Protocols. , (2019).
- Knorr, A. G., et al. Ribosome-NatA architecture reveals that rRNA expansion segments coordinate N-terminal acetylation. Nature Structural and Molecular Biology. 26 (1), 35-39 (2019).
- Matsuo, Y., Inada, T. The ribosome collision sensor Hel2 functions as preventive quality control in the secretory pathway. Cell Reports. 34 (12), (2021).
- Döring, K., et al. Profiling Ssb-Nascent chain interactions reveals principles of Hsp70-assisted folding. Cell. , (2017).
- Chartron, J. W., Hunt, K. C. L., Frydman, J. Cotranslational signal-independent SRP preloading during membrane targeting. Nature. 536 (7615), 224-228 (2016).
- Janke, C., et al. A versatile toolbox for PCR-based tagging of yeast genes: New fluorescent proteins, more markers and promoter substitution cassettes. Yeast. 21 (11), 947-962 (2004).
- Levi, O., Arava, Y. Expanding the CRISPR/Cas9 Toolbox for Gene Engineering in S. cerevisiae. Current Microbiology. 77 (3), 468-478 (2020).
- Giannoukos, G., et al. Efficient and robust RNA-seq process for cultured bacteria and complex community transcriptomes. Genome Biology. 13 (3), 23 (2012).
- . Illumina Index Adapters – Pooling Guide Available from: https://support.illumina.com/content/dam/illumina-support/documents/documentation/chemistry_documentation/experiment-design/index-adapters-pooling-guide-1000000041074-05.pdf (2019)
- Kanagawa, T. Bias and artifacts in multitemplate polymerase chain reactions (PCR). Journal of Bioscience and Bioengineering. 96 (4), 317-323 (2003).
- Bertolini, M., et al. Interactions between nascent proteins translated by adjacent ribosomes drive homomer assembly. Science. 371 (6524), (2021).
- Kramer, G., Shiber, A., Bukau, B. Mechanisms of cotranslational maturation of newly synthesized proteins. Annual Review of Biochemistry. 88, 337-364 (2019).
- Joazeiro, C. A. P. Mechanisms and functions of ribosome-associated protein quality control. Nature Reviews Molecular Cell Biology. 20 (6), 368-383 (2019).
- Beaupere, C., Chen, R. B., Pelosi, W., Labunskyy, V. M. Genome-wide quantification of translation in budding yeast by ribosome profiling. Journal of Visualized Experiments: JoVE. (130), e56820 (2017).

