Global identifikasjon av samoversettelsesinteraksjonsnettverk ved selektiv ribosomeprofilering
Summary
Samoversettelsesinteraksjoner spiller en avgjørende rolle i nascent-kjedemodifikasjoner, målretting, folding og monteringsveier. Her beskriver vi Selektiv Ribosome Profilering, en metode for in vivo, direkte analyse av disse interaksjonene i modellen eukaryote Saccharomyces cerevisiae.
Abstract
I de senere år har det blitt tydelig at ribosomer ikke bare dekoder mRNA-en vår, men også styrer fremveksten av polypeptidkjeden inn i det overfylte cellulære miljøet. Ribosomer gir plattformen for romlig og kinetisk kontrollert binding av membranmålrettingsfaktorer, modifiserende enzymer og brettbare anstander. Selv monteringen i høyordente oligomeriske komplekser, samt proteinproteinnettverksdannelsestrinn, ble nylig oppdaget å være koordinert med syntese.
Her beskriver vi Selective Ribosome Profiling, en metode utviklet for å fange opp co-translational interaksjoner in vivo. Vi vil detaljere de ulike affinitetsrensingstrinnene som kreves for å fange ribosom-nascent-kjedekomplekser sammen med kooversettelsesinteraksjoner, samt mRNA-ekstraksjon, størrelsesekskludering, omvendt transkripsjon, dypsekvensering og stordataanalysetrinn, som kreves for å dechiffrere kooversettelsesinteraksjoner i nesten-codon-oppløsning.
Introduction
Selective Ribosome Profiling (SeRP) er den eneste metoden, til dags dato, som fanger opp og karakteriserer co-translational interaksjoner, in vivo, på en direkte måte 1,2,3,4,5,6. SeRP muliggjør global profilering av interaksjoner av enhver faktor med oversettelse av ribosomer i nær codon-oppløsning 2,7.
Metoden er avhengig av flashfrysing av voksende celler og bevaring av aktiv oversettelse. Cellelysater behandles deretter med RNase I for å fordøye all mRNA i cellen bortsett fra ribosombeskyttede mRNA-fragmenter kalt “ribosome fotavtrykk”. Prøven deles deretter i to deler. en del brukes direkte til isolering av alle de cellulære ribosomale fotavtrykkene, som representerer alle pågående oversettelser i cellen. Den andre delen brukes til affinitetsrensing av den spesifikke undergruppen av ribosomer forbundet med en interessefaktor, for eksempel: modifisere enzymer, translokasjonsfaktorer, sammenleggbare anstander og komplekse monteringsinteraksjoner. De affinitetsrensede ribosomale fotavtrykkene betegnes samlet som interaktivitet. Deretter ekstraheres ribosombeskyttede mRNAer og brukes til cDNA-bibliotekgenerering, etterfulgt av dyp sekvensering.
Komparativ analyse av de totale translatom- og interactome-prøvene gjør det mulig å identifisere alle orfer som er knyttet til interessefaktoren, samt karakterisering av hver orf interaksjonsprofil. Denne profilen rapporterer de nøyaktige engasjements- og avslutningssekvensene som man kan utlede de dekodede codonene og de respektive rester av den fremvoksende polypeptidkjeden, samt på ribosomets hastighetsvariasjoner under samspillet 7,8. Figur 1 viser protokollen som et skjema.
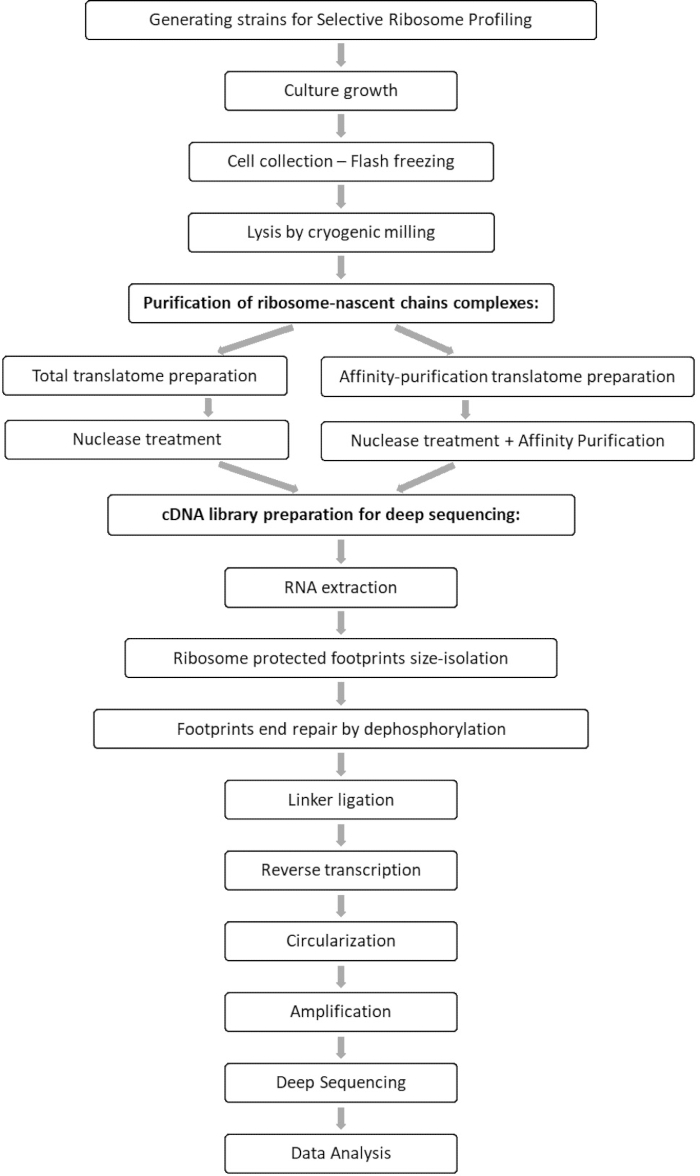
Figur 1: En oversikt over SeRP-protokollen. Denne protokollen kan utføres i sin helhet innen 7-10 dager. Klikk her for å se en større versjon av denne figuren.
Protocol
Representative Results
Discussion
Her beskriver protokollen Selektiv Ribosome Profilering-tilnærming for å fange co-oversettelsesinteraksjoner i nær codon-oppløsning. Når ribosomet stiger som et knutepunkt for koordinering av nascent-kjedens fremvekst i den overfylte cytoplasma, er dette en avgjørende metode for å identifisere og karakterisere de ulike kooversettelsesinteraksjonene som kreves for å sikre en funksjonell proteom, samt for å studere ulike sykdommer. Til dags dato er SeRP den eneste metoden som kan fange og karakterisere disse inte…
Declarações
The authors have nothing to disclose.
Acknowledgements
Vi vil takke alle laboratoriemedlemmene for fruktbare diskusjoner og Muhammad Makhzumy for den kritiske lesningen av manuskriptet. Dette arbeidet ble finansiert av ISF (Israeli Science Foundation) stipend 2106/20.
Materials
| 3'-Phosphorylated 28 nt RNA control oligonucleotide | IDT | custom order | RNase free HPLC purification; 5'-AUGUAGUCGGAGUCGAGGCGC GACGCGA/3Phos/-3' |
| Absolute ethanol | VWR | 20821 | |
| Acid phenol–chloroform | Ambion | AM9722 | |
| Antibody: mouse monclonal anti-HA | Merck | 11583816001 | 12CA5 |
| Aprotinin | Roth | A162.3 | |
| ATP* | NEB | P0756S | 10 mM |
| Bacto agar | BD | 214030 | |
| Bacto peptone | BD | 211820 | |
| Bacto tryptone | BD | 211699 | |
| Bacto yeast extract | BD | 212720 | |
| Bestatin hydrochloride | Roth | 2937.2 | |
| Chloroform | Merck | 102445 | |
| CircLigase II ssDNA Ligase* | Epicentre | CL9025K | 100 U/μL |
| Colloidal Coomassie staining solution | Roth | 4829 | |
| cOmplete, EDTA-free protease inhibitor cocktail tablets | Roche Diagnostics | 29384100 | |
| Cycloheximide | Biological Industries | A0879 | |
| DEPC treated and sterile filtered water* | Sigma | 95284 | |
| D-Glucose anhydrous | Merck | G5767-500G | |
| Diethylpyrocarbonate | Roth | K028 | |
| Dimethylsulfoxide* | Sigma-Aldrich | 276855 | |
| DNA ladder, 10 bp O'RangeRuler* | Thermo Fisher Scientific | SM1313 | |
| DNA loading dye* | Thermo Fisher Scientific | R0631 | 6× |
| DNase I, recombinant | Roche | 4716728001 | RNAse free |
| dNTP solution set* | NEB | N0446S | |
| EDTA* | Roth | 8043 | |
| Glycerol | VWR | 24388.260. | |
| Glycine solution | Sigma-Aldrich | 67419-1ML-F | 1 M |
| GlycoBlue | Ambion | AM9516 | 15 mg/mL |
| HEPES | Roth | HN78.3 | |
| HF Phusion polymerase* | NEB | M0530L | |
| HK from S. cerevisiae | Sigma-Aldrich | H6380-1.5KU | |
| Hydrochloric acid | AppliChem | A1305 | |
| Isopropanol | Sigma-Aldrich | 33539 | |
| Isopropyl β-D-1-thiogalactopyranoside | Roth | CN08 | |
| Kanamycin | Roth | T832.4 | |
| KCl | Roth | 6781.1 | |
| KH2PO4 | Roth | 3904.1 | |
| Leupeptin | Roth | CN33.4 | |
| Linker L(rt) | IDT | custom order | |
| Liquid nitrogen | |||
| MgCl2 | Roth | KK36.3 | |
| Na2HPO4 | Roth | P030.2 | |
| Na2HPO4·2H2O | Roth | T879.3 | |
| NaCl* | Invitrogen | AM97606 | 5 M |
| NaH2PO4·H2O | Roth | K300.2 | |
| NHS-activated Sepharose 4 fast-flow beads | GE Life Sciences | 17090601 | |
| Nonidet P 40 substitute | Sigma | 74385 | |
| Pepstatin A | Roth | 2936.2 | |
| Phenylmethyl sulfonyl fluoride | Roth | 6367 | |
| Precast gels | Bio-Rad | 5671034 | 10% and 12% |
| RNase I | Ambion | AM2294 | |
| SDS, 20% | Ambion | AM9820 | RNase free |
| Sodium acetate* | Ambion | AM9740 | 3 M, pH 5.5 |
| Sodium azide | Merck | S8032-100G | |
| Sodium chloride | Roth | 9265 | |
| Sodium hydroxide* | Sigma | S2770 | 1 N |
| Sucrose | Sigma-Aldrich | 16104 | |
| SUPERase-In RNase Inhibitor | Ambion | AM2694 | |
| Superscript III Reverse Transciptase* | Invitrogen | 18080-044 | |
| SYBR Gold* | Invitrogen | S11494 | |
| T4 polynucleotide kinase* | NEB | M0201L | |
| T4 RNA ligase 2* | NEB | M0242L | |
| TBE polyacrylamide gel* | Novex | EC6215BOX | 8% |
| TBE–urea polyacrylamide gel* | Novex | EC68752BOX | 10% |
| TBE–urea polyacrylamide gel* | Novex | EC6885BOX | 15% |
| TBE–urea sample buffer* | Novex | LC6876 | 2× |
| Tris | Roth | 4855 | |
| Tris* | Ambion | AM9851 | 1 M, pH 7.0 |
| Tris* | Ambion | AM9856 | 1 M, pH 8.0 |
| UltraPure 10× TBE buffer* | Invitrogen | 15581-044 | |
| * – for library preparation | |||
| gasket and spring clamp , 90 mm, | Millipore | XX1009020 | |
| ground joint flask 1 L , | Millipore | XX1504705 |
Referências
- Oh, E., et al. Selective ribosome profiling reveals the cotranslational chaperone action of trigger factor in vivo. Cell. 147 (6), 1295-1308 (2011).
- Shiber, A., et al. Cotranslational assembly of protein complexes in eukaryotes revealed by ribosome profiling. Nature. 561 (7722), 268-272 (2018).
- Becker, A. H., Oh, E., Weissman, J. S., Kramer, G., Bukau, B. Selective ribosome profiling as a tool for studying the interaction of chaperones and targeting factors with nascent polypeptide chains and ribosomes. Nature Protocols. 8 (11), 2212-2239 (2013).
- Galmozzi, C. V., Merker, D., Friedrich, U. A., Döring, K., Kramer, G. Selective ribosome profiling to study interactions of translating ribosomes in yeast. Nature Protocols. , (2019).
- Knorr, A. G., et al. Ribosome-NatA architecture reveals that rRNA expansion segments coordinate N-terminal acetylation. Nature Structural and Molecular Biology. 26 (1), 35-39 (2019).
- Matsuo, Y., Inada, T. The ribosome collision sensor Hel2 functions as preventive quality control in the secretory pathway. Cell Reports. 34 (12), (2021).
- Döring, K., et al. Profiling Ssb-Nascent chain interactions reveals principles of Hsp70-assisted folding. Cell. , (2017).
- Chartron, J. W., Hunt, K. C. L., Frydman, J. Cotranslational signal-independent SRP preloading during membrane targeting. Nature. 536 (7615), 224-228 (2016).
- Janke, C., et al. A versatile toolbox for PCR-based tagging of yeast genes: New fluorescent proteins, more markers and promoter substitution cassettes. Yeast. 21 (11), 947-962 (2004).
- Levi, O., Arava, Y. Expanding the CRISPR/Cas9 Toolbox for Gene Engineering in S. cerevisiae. Current Microbiology. 77 (3), 468-478 (2020).
- Giannoukos, G., et al. Efficient and robust RNA-seq process for cultured bacteria and complex community transcriptomes. Genome Biology. 13 (3), 23 (2012).
- . Illumina Index Adapters – Pooling Guide Available from: https://support.illumina.com/content/dam/illumina-support/documents/documentation/chemistry_documentation/experiment-design/index-adapters-pooling-guide-1000000041074-05.pdf (2019)
- Kanagawa, T. Bias and artifacts in multitemplate polymerase chain reactions (PCR). Journal of Bioscience and Bioengineering. 96 (4), 317-323 (2003).
- Bertolini, M., et al. Interactions between nascent proteins translated by adjacent ribosomes drive homomer assembly. Science. 371 (6524), (2021).
- Kramer, G., Shiber, A., Bukau, B. Mechanisms of cotranslational maturation of newly synthesized proteins. Annual Review of Biochemistry. 88, 337-364 (2019).
- Joazeiro, C. A. P. Mechanisms and functions of ribosome-associated protein quality control. Nature Reviews Molecular Cell Biology. 20 (6), 368-383 (2019).
- Beaupere, C., Chen, R. B., Pelosi, W., Labunskyy, V. M. Genome-wide quantification of translation in budding yeast by ribosome profiling. Journal of Visualized Experiments: JoVE. (130), e56820 (2017).

