DNA 메틸트랜스퍼라제 억제제를 스크리닝하기 위한 연속 형광 기반 엔도뉴클레아제 결합 DNA 메틸화 분석
Summary
DNA 메틸트랜스퍼라제는 잠재적인 항암제 표적입니다. 여기에서, DNA 메틸 트랜스퍼 라제 억제에 대한 소분자를 평가하기위한 프로토콜이 제시된다. 이 분석은 엔도뉴클레아제를 활용하여 DNA 메틸화를 형광 생성에 결합하고 효소 활성을 실시간으로 모니터링할 수 있도록 합니다.
Abstract
후성유전학적 유전자 조절의 한 형태인 DNA 메틸화는 정상적인 세포 기능에 중요합니다. 세포에서 DNA 메틸 전이 효소 (DNMT)라고하는 단백질은 DNA 메틸화 패턴을 설정하고 유지합니다. 정상적인 DNA 메틸화 패턴의 변화는 암 발생 및 진행과 관련이 있어 DNMT를 잠재적인 항암제 표적으로 만듭니다. 따라서 이러한 효소의 새로운 소분자 억제제를 식별하고 특성화하는 것이 매우 중요합니다. 이 논문은 DNA 메틸 트랜스퍼 라제 억제제를 스크리닝하는 데 사용할 수있는 프로토콜을 제시합니다. 연속 결합 동역학 분석은 잠재적인 소분자 억제제의 존재 및 부재 하에서 DNA 메틸화의 초기 속도를 결정할 수 있도록 합니다. 이 분석은 메틸 민감성 엔도 뉴 클레아 제 Gla I을 사용하여 헤미 메틸화 된 DNA 기질의 메틸화를 형광 생성에 결합시킵니다.
이 연속 분석을 통해 효소 활성을 실시간으로 모니터링할 수 있습니다. 마이크로타이터 플레이트에서 소량으로 분석을 수행하면 시약 비용이 절감됩니다. 이 분석을 사용하여 인간에서 가장 풍부한 DNMT 동종 효소 인 DNMT1의 억제제에 대한 작은 예제 스크린이 수행되었습니다. 고도로 치환 된 안트라 퀴논 천연 제품 인 락카산 A는 DNMT1의 강력한 DNA 경쟁 억제제입니다. 여기에서는 분석 프로토콜을 설명하기 위해 세 가지 잠재적 소분자 억제제(안트라퀴논 또는 1-3개의 치환기가 있는 안트라퀴논 유사 분자)를 두 가지 농도로 검사합니다. 초기 속도는 각 분자의 존재에서 관찰되는 백분율 활성을 계산하는 데 사용됩니다. 조사된 세 가지 화합물 중 하나는 DNMT1 활성의 농도 의존적 억제를 나타내며, 이는 DNMT1의 잠재적 억제제임을 나타냅니다.
Introduction
DNA 메틸화는 유전자 발현과 염색질 구조를 조절하는 중요한 후성유전학적 표시입니다. 메틸화는 주로 CpG 디 뉴클레오티드에서 발생합니다 – 시토신 다음에 구아노 신; 메틸기는 시토신의 5 위치에 추가됩니다. 올바른 DNA 메틸화 패턴, 따라서 적절한 유전자 발현은 적절한 세포 발달 및 기능에 필요합니다. 많은 질병 상태는 정상적인 메틸화 패턴 1,2,3의 변화와 관련이 있습니다. 예를 들어, 암 시작과 진행 및 DNA 메틸화 패턴의 변경 사이에는 연관성이 있습니다. 일반적으로 암세포는 메틸시토신의 전반적인 수치가 낮아 게놈 불안정성에 기여합니다. 동시에, 게놈에 존재하는 메틸 시토신은 종양 억제 유전자의 프로모터 영역에 집중되어 이러한 중요한 단백질의 유전자 침묵을 유도합니다. 특히, 후성유전학적 변화는 종양 발생과 관련된 DNA 돌연변이와 달리 역동적이고 가역적입니다. 이것은 후성 유전 학적 유전자 조절에 관여하는 단백질을 흥미로운 약물 표적 2,4로 만들었습니다.
DNA 메틸트랜스퍼라제(DNMT)는 DNA 메틸화 패턴을 생성하고 유지하는 역할을 하는 단백질입니다. 세 가지 촉매 활성 동종 효소 인 DNMT1, DNMT3a 및 DNMT3b가 인간에게 존재합니다. 개발 및 분화 과정에서 드 노보 메틸 트랜스퍼 라제 인 DNMT3a 및 DNMT3b는 메틸화 패턴을 설정합니다. 두 효소 모두 촉매 적으로 비활성 인 DNMT3L 단백질과 결합하여 증가 된 활성 1,5를 나타내는 복합체를 형성 할 수 있습니다. 세포 분열 후 딸 세포는 새로 합성된 DNA에 메틸화 표시가 없기 때문에 헤미메틸화된 DNA(듀플렉스의 한 가닥에만 메틸시토신을 포함하는 DNA)를 포함합니다. DNMT1의 주요 기능은 이 헤미메틸화 DNA를 메틸화하여 전체 메틸화 패턴 1,5를 재설정하는 것입니다.
DNMT 활동과 암 사이의 연관성은 잘 확립되어 있습니다. 전사 또는 번역 후 메커니즘에 의한 DNMT1의 과발현은 몇 가지 일반적인 발암 경로 6,7,8,9의 결과입니다. 하이포모픽 대립유전자를 사용하여 DNMT1 활성을 낮추기 위한 유전학적 접근은 Apc(Min)마우스10에서 종양 형성을 감소시킵니다. DNMT1을 녹다운시키는 안티센스 올리고뉴클레오티드는 세포 배양 및 마우스 종양 모델11,12에서 신생물을 억제합니다. 따라서 DNMT1 활성을 억제하는 것은 유망한 암 치료 접근법처럼 보입니다. 그러나 DNMT3 동종 효소가하는 역할은 그렇게 간단하지 않습니다. DNMT3a 돌연변이는 급성 골수성 백혈병13 및 골수이형성 증후군14에서 발견됩니다. 확인된 돌연변이 중 적어도 하나는 효소15의 DNA 메틸화 활성을 감소시키는 것으로 나타났다. 그러나 DNMT3b는 유방암16 및 대장 암17에서 과발현됩니다. 다양한 DNMT 동종 효소가 발암에서 서로 다른 역할을하기 때문에 동종 효소 특이 적 억제제를 식별하는 것이 중요합니다. 이러한 화합물은 치료제 개발에 유용할 뿐만 아니라 동종효소 특이적 억제제는 암 병인학에서 각 DNMT 동종효소의 역할을 해부하는 귀중한 도구가 될 것입니다.
여러 DNMT 억제제가 문헌에보고되었습니다. 공지된 DNMT 억제제는 뉴클레오시드 및 비뉴클레오사이드의 두 가지 부류로 나눌 수 있다. 뉴클레오시드 억제제는 전형적으로 시티딘 유사체이다. 이 화합물은 DNA에 통합되어 DNMT를 공유 결합적으로 포획합니다. 5- 아자 시티 딘 및 5- 아자 -2′- 데 옥시 시티 딘은 골수이 형성 증후군 및 급성 골수성 백혈병 4,18의 치료를 위해 승인되었습니다. 이들 화합물의 높은 독성, 낮은 생체이용률 및 화학적 불안정성은 문제점을 제시한다. 진행중인 연구는 차세대 뉴 클레오 시드 억제제의 효능을 조사하고 있습니다. 5-아자-2′-데옥시시티딘으로부터 유도된 SGI-110은하나의 예 19,20이다. 뉴 클레오 시드 억제제는 동종 효소 특이 적이 지 않으며 발생하는 DNMT 동종 효소를 비활성화합니다. 따라서, 뉴클레오시드-탈메틸화제로 처리하면 모든 DNMT 동종효소 4,18이 고갈된다. 비-뉴클레오시드 억제제는 억제 효과를 발휘하기 위해 DNA에 혼입될 필요가 없다. 대신, 이러한 분자는 DNMT에 직접 결합하여 동종 효소 특이 적 억제 가능성을 도입합니다. 현재까지 SGI-1027 21, 히드랄라진22, 프로카인아미드 23, RG108 및 유도체 24, 천연물 (-)-에피갈로카테킨 3-갈레이트(EGCG)25 및 락카산 A 26,27을 포함한 여러 비뉴클레오시드 억제제가 발견되었습니다. 현재까지 발견 된 대부분의 비 뉴 클레오 시드 억제제는 동종 효소 선택적이지 않거나 하나의 DNMT 동종 효소에 대해 약한 선호도를 나타냅니다. 또한, 이들 분자의 효능은 특히 세포 4,18에서 개선되어야합니다. 따라서, 보다 강력한 동종효소 선택적 DNMT 억제제를 발견하거나 개발할 필요가 있다.
DNMT의 새로운 소분자 억제제를 발견하는 데 장애물은 전통적으로 DNMT 활성28을 검사하는 데 사용되는 힘든 분석입니다. 분석은 일반적으로 여러 단계로 불연속적입니다. DNMT의 효소 활성은 여전히 방사성 S– 아데노 실 메티오닌 (SAM) 29,30,31,32,33,34를 사용하여 일상적으로 분석됩니다. DNA 메틸화에 대한 비방사성 분석법도 개발되었습니다. 예를 들어, 소화 생성물을 분리하기 위해 메틸 민감성 제한 엔도뉴클레아제 및 전기영동을 활용하는 분석이35,36에 기재되어 있다. 이러한 유형의 불연속, 다단계 분석은 약물 발견에 쉽게 적응할 수 없습니다. 2000년대 중반 이후, 더 높은 처리량을 갖는 여러 DNA 메틸화 분석법이 개발되었다28. 섬광 근접 분석을 사용하여 DNMT1 억제제37을 스크리닝하였다. 메틸 민감성 제한 엔도뉴클레아제를 활용하는 또 다른 분석을 사용하여 DNMT3a 억제제25,38을 스크리닝했습니다. 두 분석 모두 기존 DNA 메틸화 분석보다 더 높은 처리량을 허용하지만 분석에는 여러 단계가 필요하며 메틸화 활성을 실시간으로 관찰할 수 없습니다. 보다 최근에, 메틸화 반응의 한 생성물 인 S– 아데노 실 호모시스테인 (SAH)의 형성을 NADPH 산화와 관련된 340 nm에서의 분광 변화에 결합하는 연속 동역학 분석이 설명되었다39. 이 분석은 분광 신호를 생성하기 위해 세 가지 결합 효소를 사용합니다.
우리는 상업적으로 이용 가능한 단일 결합 효소를 활용하고 실시간으로 데이터를 생성할 수 있는 형광 기반 엔도뉴클레아제 결합 DNA 메틸화 분석을 개발했습니다(그림 1). 3개의 메틸시토신을 함유하는 헤어핀 올리고뉴클레오티드가 기질로서 사용된다. 기질 DNA는 5′ 말단에 형광단을 포함하고 3′ 말단에 소광제를 포함합니다. 헤미메틸화된 CpG 부위의 메틸화는 엔도뉴클레아제 Gla I에 대한 절단 부위를 생성한다 – 완전히 메틸화된 GCGC. 생성물의 GlaI 절단은 소광제로부터 형광단을 방출하고 실시간으로 형광을 발생시킨다. 분석은 DNMT의 모든 이소형의 활성을 검사하는 데 사용할 수 있습니다. 그러나 DNMT1에서는 이 동종효소가 헤미메틸화된 DNA 1,5를 우선적으로 메틸화하기 때문에 더 높은 활성이 관찰됩니다. 자가 억제 복제 초점 표적화 서열 (RFTS) 도메인이 DNMT1에서 제거되면 더욱 강력한 활성이 관찰됩니다. N- 말단 조절 영역에서 발견되는이 도메인은 촉매 부위에 결합하여 DNA 결합을 방지합니다. 처음 ~600개의 아미노산을 제거하면 전장 효소보다 훨씬 더 활성이 높은 잘린 효소가 생성됩니다(kcat/Kam에서 ~640배 증가)40. RFTS가 결여된 DNMT1(아미노산 621-1616)이라고 하는 이 활성화된 형태의 효소는 증가된 촉매력으로 인해 억제제를 더 쉽게 식별할 수 있습니다. 이 논문은 잠재적인 소분자 억제제를 스크리닝하기 위해 분석에서 RFTS가 부족한 DNMT1을 활용하는 프로토콜을 제시합니다. 엔도뉴클레아제-결합 연속 분석을 사용하여, 초기 속도는 몇 개의 작은 분자의 존재 및 부재 하에서 결정된다. 각 잠재적 억제제는 농도 의존적 DNMT1 억제를 찾기 위해 두 가지 농도에서 검사됩니다. 소분자의 존재 하에 관찰된 활성 퍼센트를 각각의 경우에 계산하였다.
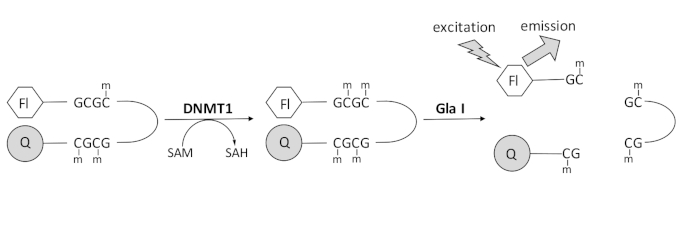
그림 1: DNA 메틸화 분석. 5′ 말단에 형광단이 있고 3′ 말단에 소광제가 있는 헤미메틸화 헤어핀 DNA가 기질로 사용됩니다. DNMT1은 S- 아데노 실 메티오닌에서 비 메틸화 된 CpG 부위로 메틸기의 전달을 촉매하여 S– 아데노 실 호모시스테인과 완전히 메틸화 된 DNA를 생성합니다. DNA 생성물은 완전히 메틸화 된 GCGC 부위를 절단하는 엔도 뉴 클레아 제 Gla I의 절단 부위를 포함합니다. 생성물 DNA의 절단은 3′ 소광기로부터 5′ 형광단을 방출하여 형광을 생성한다. 약어: Fl = 형광단; Q = 소멸제; DNMT1 = DNA 메틸 트랜스퍼 라제 1; SAM = S– 아데노 실 메티오닌; SAH = S– 아데노 실 호모시스테인. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
Protocol
Representative Results
Discussion
DNA 메틸 트랜스퍼 라제의 억제제를 확인하고 특성화하려면 효소의 활성을 측정해야합니다. DNA 메틸 트랜스퍼 라제 활성을 검사하기위한 몇 가지 방법이 존재합니다. 활동은 일반적으로 방사능을 사용하여 모니터링됩니다. SAM의 표지된 메틸기의 전사는 29,30,31,32,33,34로</…
Declarações
The authors have nothing to disclose.
Acknowledgements
저자는이 연구를 지원 한 Bucknell University와 화학과에 감사드립니다.
Materials
| 96-well Half Area Black Flat Bottom Polystyrene Not Treated Microplate | Corning | 3694 | |
| 96-Well Polystyrene Conical Bottom Plates | ThermoFisher | 249570 | |
| Bovine Serum Albumin | NEB | B9000S | |
| compound 1 | ChemBridge | 5812086 | screening compound; resuspended in DMSO to 10 mM |
| compound 2 | ChemBridge | 6722175 | screening compound; resuspended in DMSO to 10 mM |
| compound 3 | ChemBridge | 5249376 | screening compound; resuspended in DMSO to 10 mM |
| Dithiothreitol | Sigma | D0632 | |
| Gla I | SibEnzyme | E494 | methyl-sensitive endonuclease |
| Glycerol | RPI | G22025 | |
| Magnesium Chloride | Sigma | M0250 | |
| Oligonucleotide (5'-FAM-CCTATGCGmCATCAGTTTTCTGATGmCGmCATAGG-3'-Iowa Black Quencher) | IDT | custom synthesized | internally quenched hairpin DNA (substrate) |
| Potassium Glutamate | Sigma | G1501 | |
| S-adenosylmethionine | Sigma | A4377 | methyl-donating co-factor (substrate) |
| Tris Base | RPI | T60040 |
Referências
- Jurkowska, R. Z., Jurkowski, T. P., Jeltsch, A. Structure and function of mammalian DNA methyltransferases. Chembiochem. 12 (2), 206-222 (2011).
- Hamidi, T., Singh, A. K., Chen, T. Genetic alterations of DNA methylation machinery in human diseases. Epigenomics. 7 (2), 247-265 (2015).
- Norvil, A. B., Saha, D., Dar, M. S., Gowher, H. Effect of disease-associated germline mutations on structure function relationship of DNA methyltransferases. Genes. 10 (5), 369 (2019).
- Foulks, J. M., et al. Epigenetic drug discovery: targeting DNA methyltransferases. Journal of Biomolecular Screening. 17 (1), 2-17 (2012).
- Goll, M. G., Bestor, T. H. Eukaryotic cytosine methyltransferases. Annual Review of Biochemistry. 74, 481-514 (2005).
- Bigey, P., Ramchandani, S., Theberge, J., Araujo, F. D., Szyf, M. Transcriptional regulation of the human DNA Methyltransferase (dnmt1) gene. Gene. 242 (1-2), 407-418 (2000).
- Detich, N., Ramchandani, S., Szyf, M. A conserved 3′-untranslated element mediates growth regulation of DNA methyltransferase 1 and inhibits its transforming activity. Journal of Biological Chemistry. 276 (27), 24881-24890 (2001).
- MacLeod, A. R., Rouleau, J., Szyf, M. Regulation of DNA methylation by the Ras signaling pathway. Journal of Biological Chemistry. 270 (19), 11327-11337 (1995).
- Slack, A., Cervoni, N., Pinard, M., Szyf, M. DNA methyltransferase is a downstream effector of cellular transformation triggered by simian virus 40 large T antigen. Journal of Biological Chemistry. 274 (15), 10105-10112 (1999).
- Eads, C. A., Nickel, A. E., Laird, P. W. Complete genetic suppression of polyp formation and reduction of CpG-island hypermethylation in Apc(Min/+) Dnmt1-hypomorphic mice. Pesquisa do Câncer. 62, 1296-1299 (2002).
- MacLeod, A. R., Szyf, M. Expression of antisense to DNA methyltransferase mRNA induces DNA demethylation and inhibits tumorigenesis. Journal of Biological Chemistry. 270 (14), 8037-8043 (1995).
- Ramchandani, S., MacLeod, A. R., Pinard, M., von Hofe, E., Szyf, M. Inhibition of tumorigenesis by a cytosine-DNA, methyltransferase, antisense oligodeoxynucleotide. Proceedings of the National Academy Sciences of the United States of America. 94 (2), 684-689 (1997).
- Ley, T. J., et al. DNMT3A mutations in acute myeloid leukemia. New England Journal of Medicine. 363, 2424-2433 (2010).
- Walter, M. J., et al. Recurrent DNMT3A mutations in patients with myelodysplastic syndromes. Leukemia. 25 (7), 1153-1158 (2011).
- Russler-Germain, D. A., et al. The R882H DNMT3A mutation associated with AML dominantly inhibits wild-type DNMT3A by blocking its ability to form active tetramers. Cancer Cell. 25 (4), 442-454 (2014).
- Roll, J. D., Rivenbark, A. G., Jones, W. D., Coleman, W. B. DNMT3b overexpression contributes to a hypermethylator phenotype in human breast cancer cell lines. Molecular Cancer. 7, 15 (2008).
- Nosho, K., et al. DNMT3B expression might contribute to CpG island methylator phenotype in colorectal cancer. Clinical Cancer Research. 15 (11), 3663-3671 (2009).
- Erdmann, A., Halby, L., Fahy, J., Arimondo, P. B. Targeting DNA methylation with small molecules: what’s next. Journal of Medicinal Chemistry. 58 (6), 2569-2583 (2015).
- Chuang, J. C., et al. S110, a 5-Aza-2′-deoxycytidine-containing dinucleotide, is an effective DNA methylation inhibitor in vivo and can reduce tumor growth. Molecular Cancer Therapeutics. 9 (5), 1443-1450 (2010).
- Issa, J. J., et al. Safety and tolerability of guadecitabine (SGI-110) in patients with myelodysplastic syndrome and acute myeloid leukaemia: a multicentre, randomised, dose-escalation phase 1 study. Lancet Oncology. 16 (9), 1099-1110 (2015).
- Datta, J., et al. A new class of quinoline-based DNA hypomethylating agents reactivates tumor suppressor genes by blocking DNA methyltransferase 1 activity and inducing its degradation. Pesquisa do Câncer. 69 (10), 4277-4285 (2009).
- Zambrano, P., et al. A phase I study of hydralazine to demethylate and reactivate the expression of tumor suppressor genes. BMC Cancer. 5, 44 (2005).
- Lee, B. H., Yegnasubramanian, S., Lin, X., Nelson, W. G. Procainamide is a specific inhibitor of DNA methyltransferase 1. Journal of Biological Chemistry. 280 (49), 40749-40756 (2005).
- Asgatay, S., et al. Synthesis and evaluation of analogues of N-phthaloyl-l-tryptophan (RG108) as inhibitors of DNA methyltransferase 1. Journal of Medicinal Chemstry. 57 (2), 421-434 (2014).
- Ceccaldi, A., et al. C5-DNA methyltransferase inhibitors: from screening to effects on zebrafish embryo development. Chembiochem. 12 (9), 1337-1345 (2011).
- Fagan, R. L., Wu, M., Chédin, F., Brenner, C. An ultrasensitive high throughput screen for DNA methyltransferase 1-targeted molecular probes. PLoS One. 8 (11), 78752 (2013).
- Fagan, R. L., Cryderman, D. E., Kopelovich, L., Wallrath, L. L., Brenner, C. Laccaic acid A is a direct, DNA-competitive inhibitor of DNA methyltransferase 1. Journal of Biological Chemistry. 288 (33), 23858-23867 (2013).
- Eglen, R. M., Reisine, T. Screening for compounds that modulate epigenetic regulation of the transcriptome: an overview. Journal of Biomolecular Screening. 16 (10), 1137-1152 (2011).
- Holz-Schietinger, C., Matje, D. M., Reich, N. O. Mutations in DNA methyltransferase (DNMT3A) observed in acute myeloid leukemia patients disrupt processive methylation. Journal of Biological Chemistry. 287 (37), 30941-30951 (2012).
- Norvil, A. B., et al. Dnmt3b methylates DNA by a noncooperative mechanism, and its activity is unaffected by manipulations at the predicted dimer interface. Bioquímica. 57 (29), 4312-4324 (2018).
- Bashtrykov, P., Ragozin, S., Jeltsch, A. Mechanistic details of the DNA recognition by the Dnmt1 DNA methyltransferase. FEBS Letters. 586 (13), 1821-1823 (2012).
- Bashtrykov, P., et al. Targeted mutagenesis results in an activation of DNA methyltransferase 1 and confirms an autoinhibitory role of its RFTS domain. Chembiochem. 15 (5), 743-748 (2014).
- Berkyurek, A. C., et al. The DNA methyltransferase Dnmt1 directly interacts with the SET and RING finger-associated (SRA) domain of the multifunctional protein Uhrf1 to facilitate accession of the catalytic center to hemi-methylated DNA. Journal of Biological Chemistry. 289 (1), 379-386 (2014).
- Kanada, K., Takeshita, K., Suetake, I., Tajima, S., Nakagawa, A. Conserved threonine 1505 in the catalytic domain stabilizes mouse DNA methyltransferase 1. Journal of Biochemistry. 162 (4), 271-278 (2017).
- Bashtrykov, P., et al. Specificity of Dnmt1 for methylation of hemimethylated CpG sites resides in its catalytic domain. Chemistry & Biology. 19 (5), 572-578 (2012).
- Dolen, E. K., McGinnis, J. H., Tavory, R. N., Weiss, J. A., Switzer, R. L. Disease-associated mutations G589A and V590F relieve replication focus targeting sequence-mediated autoinhibition of DNA methyltransferase 1. Bioquímica. 58 (51), 5151-5159 (2019).
- Kilgore, J. A., et al. Identification of DNMT1 selective antagonists using a novel scintillation proximity assay. Journal of Biological Chemistry. 288 (27), 19673-19684 (2013).
- Ceccaldi, A., et al. Identification of novel inhibitors of DNA methylation by screening of a chemical library. ACS Chemical Biology. 8 (3), 543-548 (2013).
- Duchin, S., Vershinin, Z., Levy, D., Aharoni, A. A continuous kinetic assay for protein and DNA methyltransferase enzymatic activities. Epigenetics & Chromatin. 8, 56 (2015).
- Syeda, F., et al. The replication focus targeting sequence (RFTS) domain is a DNA-competitive inhibitor of Dnmt1. Journal of Biological Chemistry. 286 (17), 15344-15351 (2011).
- Switzer, R. L., Medrano, J., Reedel, D. A., Weiss, J. Substituted anthraquinones represent a potential scaffold for DNA methyltransferase 1-specific inhibitors. PLoS One. 14 (7), 0219830 (2019).

