Kontinuerlig fluorescensbasert endonukleasekoblet DNA-metyleringsanalyse for å screene for DNA-metyltransferasehemmere
Summary
DNA-metyltransferaser er potensielle kreftmedisinmål. Her presenteres en protokoll for å vurdere små molekyler for DNA-metyltransferasehemming. Denne analysen benytter en endonuklease for å koble DNA-metylering til fluorescensgenerering og gjør det mulig å overvåke enzymaktivitet i sanntid.
Abstract
DNA-metylering, en form for epigenetisk genregulering, er viktig for normal cellulær funksjon. I celler etablerer og opprettholder proteiner kalt DNA-metyltransferaser (DNMT) DNA-metyleringsmønsteret. Endringer i det normale DNA-metyleringsmønsteret er knyttet til kreftutvikling og progresjon, noe som gjør DNMTs potensielle kreftmedisinmål. Dermed er identifisering og karakterisering av nye små molekylhemmere av disse enzymene av stor betydning. Dette papiret presenterer en protokoll som kan brukes til å skjerme for DNA-metyltransferasehemmere. Den kontinuerlige koblede kinetikkanalysen gjør det mulig å bestemme innledende hastigheter av DNA-metylering i nærvær og fravær av potensielle små molekylhemmere. Analysen bruker den metylfølsomme endonuklease Gla I til å koble metylering av et hemimetylert DNA-substrat til fluorescensgenerering.
Denne kontinuerlige analysen gjør det mulig å overvåke enzymaktiviteten i sanntid. Gjennomføring av analysen i små mengder i mikrotiterplater reduserer kostnadene for reagenser. Ved hjelp av denne analysen ble det utført en liten eksempelskjerm for hemmere av DNMT1, den mest omfattende DNMT-isozymen hos mennesker. Det svært substituerte naturproduktet antrakinon, lakkasyre A, er en potent, DNA-kompetitiv hemmer av DNMT1. Her undersøker vi tre potensielle små molekylhemmere – antrakinoner eller antrakinonlignende molekyler med en til tre substituenter – i to konsentrasjoner for å beskrive analyseprotokollen. Innledende hastigheter brukes til å beregne prosentaktiviteten observert i nærvær av hvert molekyl. En av tre undersøkte forbindelser utviser konsentrasjonsavhengig hemming av DNMT1-aktivitet, noe som indikerer at det er en potensiell hemmer av DNMT1.
Introduction
DNA-metylering er et viktig epigenetisk merke som regulerer genuttrykk og kromatinstruktur. Metylering forekommer hovedsakelig i CpG-dinukleotider – cytosin etterfulgt av guanosin; metylgruppen tilsettes til 5-posisjonen av cytosin. Korrekte DNA-metyleringsmønstre, og dermed riktig genuttrykk, er nødvendig for passende cellulær utvikling og funksjon. Mange sykdomstilstander har vært assosiert med endringer i det normale metyleringsmønsteret 1,2,3. For eksempel er det en sammenheng mellom kreftinitiering og progresjon og endringer i DNA-metyleringsmønsteret. Vanligvis viser kreftceller lavere generelle nivåer av metylcytosin, noe som bidrar til ustabilitet i genomet. Samtidig er metylcytosinet som er tilstede i genomet konsentrert i promotorområdene av tumorundertrykkende gener, noe som fører til geninaktivering av disse viktige proteinene. Spesielt er epigenetiske endringer dynamiske og reversible, i motsetning til DNA-mutasjonene forbundet med tumorigenese. Dette har gjort proteinene involvert i epigenetisk genregulering interessante legemiddelmål 2,4.
DNA-metyltransferaser (DNMT) er proteinene som er ansvarlige for å generere og opprettholde DNA-metyleringsmønstre. Tre katalytisk aktive isoenzymer, DNMT1, DNMT3a og DNMT3b, finnes hos mennesker. Under utvikling og differensiering etablerer de novo metyltransferaser, DNMT3a og DNMT3b, metyleringsmønstre. Begge enzymene kan binde det katalytisk inaktive DNMT3L-proteinet for å danne komplekser som viser økt aktivitet 1,5. Etter celledeling inneholder datterceller hemimetylert DNA – DNA som inneholder metylcytosin i bare en streng av dupleks – fordi det nylig syntetiserte DNA er blottet for metyleringsmerker. Hovedfunksjonen til DNMT1 er å metylere dette hemimetylerte DNA, og dermed gjenopprette det fulle metyleringsmønsteret 1,5.
Koblinger mellom DNMT aktivitet og kreft er godt etablert. Overekspresjon av DNMT1, enten ved transkripsjonelle eller posttranslasjonelle mekanismer, er en konsekvens av flere vanlige onkogene veier 6,7,8,9. Genetiske tilnærminger til lavere DNMT1-aktivitet ved bruk av hypomorfe alleler resulterer i redusert tumordannelse hos Apc(Min)mus10. Antisense oligonukleotider som knockdown DNMT1 hemmer neoplasi i cellekultur og musetumormodeller11,12. Dermed virker hemming av DNMT1-aktivitet som en lovende kreftbehandlingsmetode. Rollene DNMT3-isozymene spiller er imidlertid ikke så enkle. DNMT3a-mutasjoner finnes ved akutt myelogen leukemi13 og myelodysplastisk syndrom14. Minst en av de identifiserte mutasjonene har vist seg å redusere DNA-metyleringsaktiviteten til enzymet15. DNMT3b er imidlertid overuttrykt i brystkreft16 og kolorektal kreft17. Med de forskjellige DNMT-isoenzymene som spiller forskjellige roller i karsinogenese, vil identifisering av isozymespesifikke hemmere være kritisk. Ikke bare vil disse forbindelsene være nyttige for utvikling av terapeutiske midler, men isozyme-spesifikke hemmere vil også være et uvurderlig verktøy for å dissekere rollen til hver DNMT-isozyme i kreftetiologi.
Flere DNMT-hemmere er rapportert i litteraturen. Kjente DNMT-hemmere kan deles inn i to klasser: nukleosid og ikke-nukleosid. Nukleosidhemmere er vanligvis cytidinanaloger. Disse forbindelsene er inkorporert i DNA og kovalent felle DNMTs. 5-azacytidin og 5-aza-2′-deoksycytidin er godkjent for behandling av myelodysplastisk syndrom og akutt myelogen leukemi 4,18. Den høye toksisiteten, lav biotilgjengelighet og kjemisk ustabilitet av disse forbindelsene gir problemer. Pågående arbeid undersøker effekten av neste generasjon nukleosidhemmere; SGI-110, avledet fra 5-aza-2′-deoksycytidin, er et eksempel 19,20. Nukleosidhemmere er ikke isozymespesifikke og vil inaktivere ethvert dnmt-isoenzym som oppstår. Derfor resulterer behandling med et nukleosid-demetyleringsmiddel i uttømming av alle DNMT-isoenzymer 4,18. Ikke-nukleosidhemmere trenger ikke å bli innlemmet i DNA for å utøve sine hemmende effekter. I stedet binder disse molekylene seg direkte til DNMT, og introduserer muligheten for isozymespesifikk inhibering. Flere ikke-nukleosidhemmere har blitt oppdaget til dags dato, inkludert SGI-1027 21, hydralazin22, prokainamid 23, RG108 og derivater 24, og naturlige produkter, (−)-epigallocatechin 3-gallate (EGCG)25 og lakkasyre A26,27. De fleste av de ikke-nukleosidhemmere oppdaget til dags dato er ikke isozyme-selektive eller vise svake preferanser for en DNMT isozyme. I tillegg må styrken til disse molekylene forbedres, spesielt i celler 4,18. Dermed er det behov for å oppdage eller utvikle mer potente, isozymeselektive DNMT-hemmere.
Et hinder for å oppdage nye små molekylhemmere av DNMTs er de arbeidskrevende analysene som tradisjonelt brukes til å undersøke DNMT-aktivitet28. Analyser er vanligvis diskontinuerlige med flere trinn. Den enzymatiske aktiviteten til DNMTs er fortsatt rutinemessig analysert ved bruk av radioaktiv S-adenosylmetionin (SAM) 29,30,31,32,33,34. Ikke-radioaktive analyser for DNA-metylering er også utviklet. For eksempel er analyser som benytter metylfølsomme restriksjonsendonukleaser og elektroforese for å skille fordøyelsesproduktene beskrevet35,36. Denne typen diskontinuerlige, flertrinnsanalyser er ikke lett mottagelige for narkotikaforskning. Siden midten av 2000-tallet har flere DNA-metyleringsanalyser med høyere gjennomstrømning blitt utviklet28. En scintillasjonsnærhetsanalyse ble brukt til å screene for DNMT1-hemmere37. En annen analyse ved bruk av en metylfølsom restriksjonsendonuklease ble brukt til å screene for DNMT3a-hemmere25,38. Mens begge analysene tillot høyere gjennomstrømning enn tradisjonelle DNA-metyleringsanalyser, krever analysene flere trinn og tillater ikke observasjon av metyleringsaktivitet i sanntid. Mer nylig er det beskrevet en kontinuerlig kinetikkanalyse som parrer dannelsen av S-adenosylhomocystein (SAH), et produkt av metyleringsreaksjonen, til den spektroskopiske forandringen ved 340 nm assosiert med NADPH-oksidasjon39. Denne analysen bruker tre koblingsenzymer for å generere et spektroskopisk signal.
Vi utviklet en fluorescensbasert endonukleasekoblet DNA-metyleringsanalyse som benytter et enkelt kommersielt tilgjengelig koblingsenzym og kan generere data i sanntid (figur 1). Et hårnålsoligonukleotid som inneholder tre metylcytosiner brukes som substrat. Substrat-DNA inneholder en fluorofor på 5′-enden og en slukker på 3′-enden. Metylering av det hemimetylerte CpG-området genererer spaltningsstedet for endonuklease Gla I – fullt metylert GCGC. Gla I-spaltning av produktet oligonukleotid frigjør fluoroforen fra slukkeren og genererer fluorescens i sanntid. Analysen kan brukes til å undersøke aktiviteten til en hvilken som helst isoform av DNMT; Imidlertid observeres høyere aktivitet med DNMT1, da dette isozymet fortrinnsvis metylerer hemimetylert DNA 1,5. Enda mer robust aktivitet observeres hvis domenet autoinhibitory Replication Foci Targeting Sequence (RFTS) fjernes fra DNMT1. Dette domenet, som finnes i N-terminalreguleringsområdet, binder seg til det katalytiske stedet og forhindrer DNA-binding. Fjerning av de første ~ 600 aminosyrene resulterer i et avkortet enzym som er betydelig mer aktivt enn enzymet i full lengde (~ 640 ganger økning i kcat / Km) 40. Denne aktiverte formen av enzymet, referert til som RFTS-manglende DNMT1 (aminosyrer 621-1616), muliggjør lettere identifisering av inhibitorer på grunn av sin økte katalytiske kraft. Dette papiret presenterer en protokoll for å bruke RFTS-manglende DNMT1 i analyser for å skjerme for potensielle små molekylhemmere. Ved bruk av endonukleasekoblet kontinuerlig analyse bestemmes starthastigheten i nærvær og fravær av noen få små molekyler. Hver potensiell hemmer undersøkes i to konsentrasjoner for å se etter konsentrasjonsavhengig DNMT1-hemming. Den prosentvise aktiviteten observert i nærvær av de små molekylene ble beregnet i hvert tilfelle.
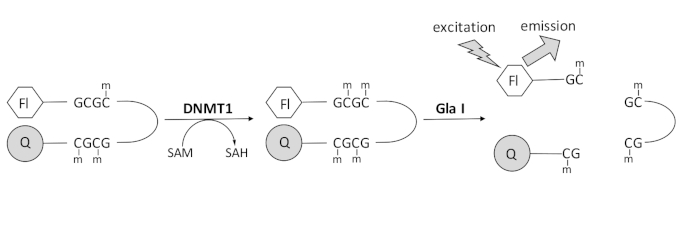
Figur 1: DNA-metyleringsanalyse. Et hemimetylert hårnåls-DNA med en fluorofor på 5′-enden og en slukker på 3′-enden brukes som substrat. DNMT1 katalyserer overføringen av metylgruppen fra S-adenosylmetionin til det ikke-metylerte CpG-stedet, genererer S-adenosylhomocystein og fullt metylert DNA. DNA-produktet inneholder spaltningsstedet for endonuklease Gla I, som klipper fullt metylerte GCGC-steder. Spaltning av produkt-DNA frigjør 5′-fluoroforen fra 3′-slukkeren, og genererer fluorescens. Forkortelser: Fl = fluorofor; Q = slukke; DNMT1 = DNA-metyltransferase 1; SAM = S-adenosylmethionine; SAH = S-adenosylhomocystein. Klikk her for å se en større versjon av dette tallet.
Protocol
Representative Results
Discussion
For å identifisere og karakterisere inhibitorer av DNA-metyltransferaser, må enzymets aktivitet måles. Det finnes flere metoder for å undersøke DNA-metyltransferaseaktivitet. Aktivitet overvåkes vanligvis ved hjelp av radioaktivitet; overføring av den merkede metylgruppen av SAM kan kvantifiseres 29,30,31,32,33,34. Gel…
Declarações
The authors have nothing to disclose.
Acknowledgements
Forfatterne takker Bucknell University og Institutt for kjemi for deres støtte til dette arbeidet.
Materials
| 96-well Half Area Black Flat Bottom Polystyrene Not Treated Microplate | Corning | 3694 | |
| 96-Well Polystyrene Conical Bottom Plates | ThermoFisher | 249570 | |
| Bovine Serum Albumin | NEB | B9000S | |
| compound 1 | ChemBridge | 5812086 | screening compound; resuspended in DMSO to 10 mM |
| compound 2 | ChemBridge | 6722175 | screening compound; resuspended in DMSO to 10 mM |
| compound 3 | ChemBridge | 5249376 | screening compound; resuspended in DMSO to 10 mM |
| Dithiothreitol | Sigma | D0632 | |
| Gla I | SibEnzyme | E494 | methyl-sensitive endonuclease |
| Glycerol | RPI | G22025 | |
| Magnesium Chloride | Sigma | M0250 | |
| Oligonucleotide (5'-FAM-CCTATGCGmCATCAGTTTTCTGATGmCGmCATAGG-3'-Iowa Black Quencher) | IDT | custom synthesized | internally quenched hairpin DNA (substrate) |
| Potassium Glutamate | Sigma | G1501 | |
| S-adenosylmethionine | Sigma | A4377 | methyl-donating co-factor (substrate) |
| Tris Base | RPI | T60040 |
Referências
- Jurkowska, R. Z., Jurkowski, T. P., Jeltsch, A. Structure and function of mammalian DNA methyltransferases. Chembiochem. 12 (2), 206-222 (2011).
- Hamidi, T., Singh, A. K., Chen, T. Genetic alterations of DNA methylation machinery in human diseases. Epigenomics. 7 (2), 247-265 (2015).
- Norvil, A. B., Saha, D., Dar, M. S., Gowher, H. Effect of disease-associated germline mutations on structure function relationship of DNA methyltransferases. Genes. 10 (5), 369 (2019).
- Foulks, J. M., et al. Epigenetic drug discovery: targeting DNA methyltransferases. Journal of Biomolecular Screening. 17 (1), 2-17 (2012).
- Goll, M. G., Bestor, T. H. Eukaryotic cytosine methyltransferases. Annual Review of Biochemistry. 74, 481-514 (2005).
- Bigey, P., Ramchandani, S., Theberge, J., Araujo, F. D., Szyf, M. Transcriptional regulation of the human DNA Methyltransferase (dnmt1) gene. Gene. 242 (1-2), 407-418 (2000).
- Detich, N., Ramchandani, S., Szyf, M. A conserved 3′-untranslated element mediates growth regulation of DNA methyltransferase 1 and inhibits its transforming activity. Journal of Biological Chemistry. 276 (27), 24881-24890 (2001).
- MacLeod, A. R., Rouleau, J., Szyf, M. Regulation of DNA methylation by the Ras signaling pathway. Journal of Biological Chemistry. 270 (19), 11327-11337 (1995).
- Slack, A., Cervoni, N., Pinard, M., Szyf, M. DNA methyltransferase is a downstream effector of cellular transformation triggered by simian virus 40 large T antigen. Journal of Biological Chemistry. 274 (15), 10105-10112 (1999).
- Eads, C. A., Nickel, A. E., Laird, P. W. Complete genetic suppression of polyp formation and reduction of CpG-island hypermethylation in Apc(Min/+) Dnmt1-hypomorphic mice. Pesquisa do Câncer. 62, 1296-1299 (2002).
- MacLeod, A. R., Szyf, M. Expression of antisense to DNA methyltransferase mRNA induces DNA demethylation and inhibits tumorigenesis. Journal of Biological Chemistry. 270 (14), 8037-8043 (1995).
- Ramchandani, S., MacLeod, A. R., Pinard, M., von Hofe, E., Szyf, M. Inhibition of tumorigenesis by a cytosine-DNA, methyltransferase, antisense oligodeoxynucleotide. Proceedings of the National Academy Sciences of the United States of America. 94 (2), 684-689 (1997).
- Ley, T. J., et al. DNMT3A mutations in acute myeloid leukemia. New England Journal of Medicine. 363, 2424-2433 (2010).
- Walter, M. J., et al. Recurrent DNMT3A mutations in patients with myelodysplastic syndromes. Leukemia. 25 (7), 1153-1158 (2011).
- Russler-Germain, D. A., et al. The R882H DNMT3A mutation associated with AML dominantly inhibits wild-type DNMT3A by blocking its ability to form active tetramers. Cancer Cell. 25 (4), 442-454 (2014).
- Roll, J. D., Rivenbark, A. G., Jones, W. D., Coleman, W. B. DNMT3b overexpression contributes to a hypermethylator phenotype in human breast cancer cell lines. Molecular Cancer. 7, 15 (2008).
- Nosho, K., et al. DNMT3B expression might contribute to CpG island methylator phenotype in colorectal cancer. Clinical Cancer Research. 15 (11), 3663-3671 (2009).
- Erdmann, A., Halby, L., Fahy, J., Arimondo, P. B. Targeting DNA methylation with small molecules: what’s next. Journal of Medicinal Chemistry. 58 (6), 2569-2583 (2015).
- Chuang, J. C., et al. S110, a 5-Aza-2′-deoxycytidine-containing dinucleotide, is an effective DNA methylation inhibitor in vivo and can reduce tumor growth. Molecular Cancer Therapeutics. 9 (5), 1443-1450 (2010).
- Issa, J. J., et al. Safety and tolerability of guadecitabine (SGI-110) in patients with myelodysplastic syndrome and acute myeloid leukaemia: a multicentre, randomised, dose-escalation phase 1 study. Lancet Oncology. 16 (9), 1099-1110 (2015).
- Datta, J., et al. A new class of quinoline-based DNA hypomethylating agents reactivates tumor suppressor genes by blocking DNA methyltransferase 1 activity and inducing its degradation. Pesquisa do Câncer. 69 (10), 4277-4285 (2009).
- Zambrano, P., et al. A phase I study of hydralazine to demethylate and reactivate the expression of tumor suppressor genes. BMC Cancer. 5, 44 (2005).
- Lee, B. H., Yegnasubramanian, S., Lin, X., Nelson, W. G. Procainamide is a specific inhibitor of DNA methyltransferase 1. Journal of Biological Chemistry. 280 (49), 40749-40756 (2005).
- Asgatay, S., et al. Synthesis and evaluation of analogues of N-phthaloyl-l-tryptophan (RG108) as inhibitors of DNA methyltransferase 1. Journal of Medicinal Chemstry. 57 (2), 421-434 (2014).
- Ceccaldi, A., et al. C5-DNA methyltransferase inhibitors: from screening to effects on zebrafish embryo development. Chembiochem. 12 (9), 1337-1345 (2011).
- Fagan, R. L., Wu, M., Chédin, F., Brenner, C. An ultrasensitive high throughput screen for DNA methyltransferase 1-targeted molecular probes. PLoS One. 8 (11), 78752 (2013).
- Fagan, R. L., Cryderman, D. E., Kopelovich, L., Wallrath, L. L., Brenner, C. Laccaic acid A is a direct, DNA-competitive inhibitor of DNA methyltransferase 1. Journal of Biological Chemistry. 288 (33), 23858-23867 (2013).
- Eglen, R. M., Reisine, T. Screening for compounds that modulate epigenetic regulation of the transcriptome: an overview. Journal of Biomolecular Screening. 16 (10), 1137-1152 (2011).
- Holz-Schietinger, C., Matje, D. M., Reich, N. O. Mutations in DNA methyltransferase (DNMT3A) observed in acute myeloid leukemia patients disrupt processive methylation. Journal of Biological Chemistry. 287 (37), 30941-30951 (2012).
- Norvil, A. B., et al. Dnmt3b methylates DNA by a noncooperative mechanism, and its activity is unaffected by manipulations at the predicted dimer interface. Bioquímica. 57 (29), 4312-4324 (2018).
- Bashtrykov, P., Ragozin, S., Jeltsch, A. Mechanistic details of the DNA recognition by the Dnmt1 DNA methyltransferase. FEBS Letters. 586 (13), 1821-1823 (2012).
- Bashtrykov, P., et al. Targeted mutagenesis results in an activation of DNA methyltransferase 1 and confirms an autoinhibitory role of its RFTS domain. Chembiochem. 15 (5), 743-748 (2014).
- Berkyurek, A. C., et al. The DNA methyltransferase Dnmt1 directly interacts with the SET and RING finger-associated (SRA) domain of the multifunctional protein Uhrf1 to facilitate accession of the catalytic center to hemi-methylated DNA. Journal of Biological Chemistry. 289 (1), 379-386 (2014).
- Kanada, K., Takeshita, K., Suetake, I., Tajima, S., Nakagawa, A. Conserved threonine 1505 in the catalytic domain stabilizes mouse DNA methyltransferase 1. Journal of Biochemistry. 162 (4), 271-278 (2017).
- Bashtrykov, P., et al. Specificity of Dnmt1 for methylation of hemimethylated CpG sites resides in its catalytic domain. Chemistry & Biology. 19 (5), 572-578 (2012).
- Dolen, E. K., McGinnis, J. H., Tavory, R. N., Weiss, J. A., Switzer, R. L. Disease-associated mutations G589A and V590F relieve replication focus targeting sequence-mediated autoinhibition of DNA methyltransferase 1. Bioquímica. 58 (51), 5151-5159 (2019).
- Kilgore, J. A., et al. Identification of DNMT1 selective antagonists using a novel scintillation proximity assay. Journal of Biological Chemistry. 288 (27), 19673-19684 (2013).
- Ceccaldi, A., et al. Identification of novel inhibitors of DNA methylation by screening of a chemical library. ACS Chemical Biology. 8 (3), 543-548 (2013).
- Duchin, S., Vershinin, Z., Levy, D., Aharoni, A. A continuous kinetic assay for protein and DNA methyltransferase enzymatic activities. Epigenetics & Chromatin. 8, 56 (2015).
- Syeda, F., et al. The replication focus targeting sequence (RFTS) domain is a DNA-competitive inhibitor of Dnmt1. Journal of Biological Chemistry. 286 (17), 15344-15351 (2011).
- Switzer, R. L., Medrano, J., Reedel, D. A., Weiss, J. Substituted anthraquinones represent a potential scaffold for DNA methyltransferase 1-specific inhibitors. PLoS One. 14 (7), 0219830 (2019).

